Patogenicidad y modulación inmune del circovirus porcino 3
Patogenicidad y modulación inmune del circovirus porcino 3
 Dengjin Chen1
Dengjin Chen1  Lei Zhang1
Lei Zhang1  Shengkui Xu2*
Shengkui Xu2*- 1Laboratorio Clave de Productos Biológicos Veterinarios y Medicamentos Químicos, Ministerio de Agricultura y Asuntos Rurales, Centro de Investigación de Ingeniería y Tecnología para el Diseño y Preparación de Vacunas de Péptidos Veterinarios de Beijing, Institutos Zhongmu de China Industria de Ganadería Co., Ltd., Beijing, China
- 2Laboratorio Clave de Medicina Veterinaria Tradicional China de Beijing, Facultad de Ciencia y Tecnología Animal, Universidad de Agricultura de Beijing, Beijing, China
Los circovirus porcinos (PCV) son miembros del género Circovirus de la familia Circoviridae, y se han descubierto cuatro especies de PCV que se denominan PCV1-PCV4, respectivamente. Con el primer informe de PCV3 en Estados Unidos en 2016, se encontró que la variante patógena estaba asociada con varias características clínicas, llamadas enfermedad asociada al circovirus porcino (PCVAD, por sus siglas en inglés), que incluyen inflamación multisistémica, síndrome de dermatitis y nefropatía porcina (PDNS, por sus siglas en inglés), trastornos reproductivos, trastornos respiratorios o digestivos. Cada vez hay más datos experimentales que el PCV3 está muy extendido en todo el mundo, pero el fracaso del aislamiento y la propagación del virus ha puesto obstáculos en el camino de la investigación del PCV3. Además, un gran número de informes demuestran que el PCV3 suele coinfectar con otros patógenos en cerdos. Por lo tanto, es necesario discutir a fondo si la PCV3 por sí sola causa manifestaciones clínicas. Además, la respuesta inmunitaria de la célula huésped se activó durante la infección por PCV3, y las proteínas codificadas por PCV3 pueden regular las respuestas inmunitarias para facilitar su replicación. Un conocimiento profundo de la patogénesis de la PCV3 y de las estrategias de regulación inmunitaria es fundamental para la prevención de la PCVAD. En esta revisión, se resumieron los avances en patogenicidad y modulación inmune innata de PCV3, lo que podría profundizar en la comprensión de este virus y las enfermedades relacionadas con PCV3.
1 Introducción
Los circovirus porcinos (PCV), los virus de ADN más pequeños conocidos en mamíferos, son miembros del género Circovirus de la familia Circoviridae. Se han descubierto al menos cuatro especies de PCV en cerdos y se han denominado PCV1 a PCV4, respectivamente, según el análisis de homología del genoma. En la década de 1970, PCV1 se encontró por primera vez en células PK-15 y luego demostró que no era patógeno en estudios posteriores (1, 2). Posteriormente, se encontró una cepa variante de PCV2 en América del Norte y Europa y se cree que es responsable de múltiples presentaciones clínicas, como el síndrome de desgaste multisistémico posterior al destete (3-5). Por lo tanto, se llevaron a cabo extensos estudios y se lograron grandes avances en la patogénesis viral y los mecanismos de regulación inmune en los siguientes 20 años. En 2016, se informó de un nuevo virus, que causa síntomas similares a los de PCV2, y se denominó PCV3 (6). Además, la PCV4 se descubrió recientemente en 2019 (7), y la información sobre la patogénesis y las implicaciones clínicas de la PCV4 es limitada.
La PCV3 se detectó inicialmente en los Estados Unidos en cerdos que padecían dermatitis porcina y síndrome de nefropatía (SDPD) utilizando tecnología de secuenciación de alto rendimiento (6). Sin embargo, numerosos estudios retrospectivos sugieren que el patógeno ha estado circulando entre los cerdos durante mucho tiempo en Asia, Europa, América del Sur y América del Norte (8-14). En particular, la mayoría de los cerdos PDNS son PCV3 positivos, pero no PCV2, y la enfermedad similar a PDNS en lechones se reprodujo con éxito en base a un clon infeccioso de ADN PCV3 (15), lo que indica que PCV3 es suficiente para causar PDNS solamente, y se debe realizar mucha investigación para revelar su biología y patogénesis. Además, la PCV3 fue muy prevalente en jabalíes (16-21). Por lo tanto, las poblaciones silvestres pueden ser un reservorio importante y podrían representar un riesgo concreto de propagación a las poblaciones domésticas.
En general, todos los PCV comparten una morfología similar, que consiste en un genoma circular cerrado de ADN monocatenario (22). El genoma consistió en aproximadamente 1760 nucleótidos (nt), 1767 nt y 2000 nt en PCV1, PCV2 y PCV3 (23), respectivamente. Además, la secuencia de PCV3 fue solo homóloga en un 48% a PCV2, aunque comparten morfologías genómicas similares (24). Un análisis filogenético adicional también demostró la baja similitud entre PCV3 y otros PCV, como 45,5 y 43,2% para PCV1 y PCV4, respectivamente (25). Estudios recientes informaron que el nuevo PCV3 podría originarse en murciélagos debido a su alta homología con algunos circovirus de murciélagos tanto a nivel de nucleótidos como de aminoácidos (26, 27).
Los PCV consisten en tres marcos de lectura abiertos (ORF) principales dispuestos en las hebras de la forma replicativa (28). ORF1 se localiza en la hebra positiva y se identifica como la región más conservada del genoma del circovirus (29), codificando para las proteínas Rep y Rep′ implicadas en el inicio de la replicación del virus (30); ORF2 se localiza en la cadena viral de ADN negativo y codifica la única proteína estructural (Cap), que es la proteína viral más inmunogénica y posee múltiples funciones (31); ORF3 está orientado en la hebra negativa, que codifica para una proteína no estructural que regula la apoptosis de la célula huésped en PCV1 y PCV2 (32, 33), mientras que su función sigue siendo desconocida en PCV3 (6, 34). Tradicionalmente, la PCV3 se divide en tres clados principales (PCV3a, PCV3b y PCV3c) en función de las variaciones de aminoácidos (35, 36), y la PCV3a podría clasificarse además en subclados, incluidos PCV3a-1, PCV3a-2 y un clado intermedio (PCV3a-IM) (26, 27, 35, 36).
Desde el primer informe de PCV3, se ha detectado ampliamente en los pulmones, el corazón, los riñones y otros órganos o tejidos (15) y ha causado una variedad de manifestaciones clínicas (37). La investigación actual demuestra que varios factores de confusión afectan las manifestaciones clínicas y los cambios patológicos de los cerdos enfermos (38). La PCV3 puede replicarse en casi todos los tejidos, especialmente en las células inmunitarias, donde causa daños celulares específicos, como la apoptosis celular y la supresión inmunitaria. Además, la infección por PCV3 podría aumentar significativamente las citoquinas proinflamatorias y causar inflamación multisistémica en lechones y cerdas (15). Por lo tanto, la interacción entre la infección por PCV3 y las respuestas inmunitarias es fundamental para su patogenicidad. En esta revisión, analizaremos y organizaremos la patogenicidad y los mecanismos de modulación inmunitaria de la PCV3.
2 Patogenicidad de PCV3 y sus enfermedades asociadas
Una serie de síntomas clínicos causados por los PCV se conocen colectivamente como enfermedades asociadas al circovirus porcino (PCVAD, por sus siglas en inglés). En los últimos 20 años, la PCV2 ha sido considerada como la principal causa de PCVAD. La PCV2 prolifera principalmente en los ganglios linfáticos e induce la apoptosis de las células inmunitarias para disminuir la inmunidad de los cerdos infectados. También se han logrado avances interesantes en la patogenicidad de PCV3. Jiang et al. reprodujeron con éxito una enfermedad similar a la PDNS basada en PCV3 sola o PCV3 en combinación con un estimulador inmunológico en animales (15). De la evidencia se desprende claramente que la infección por PCV3 conduce a una variedad de síntomas clínicos y patológicos, que incluyen PDNS, trastornos reproductivos, enfermedades inflamatorias sistémicas (39), trastornos respiratorios (34, 40, 41), diarrea (40, 42) y signos del sistema nervioso central (43, 44). En este trabajo describimos los avances recientes en los signos clínicos más consistentes asociados a la infección por PCV3 (Figura 1).
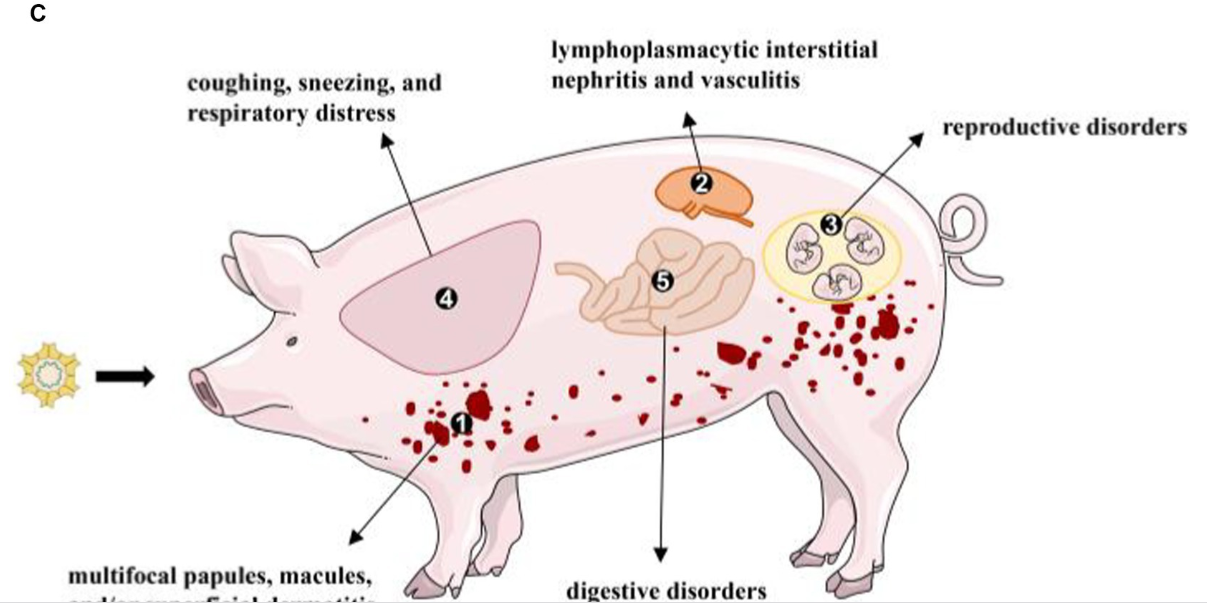
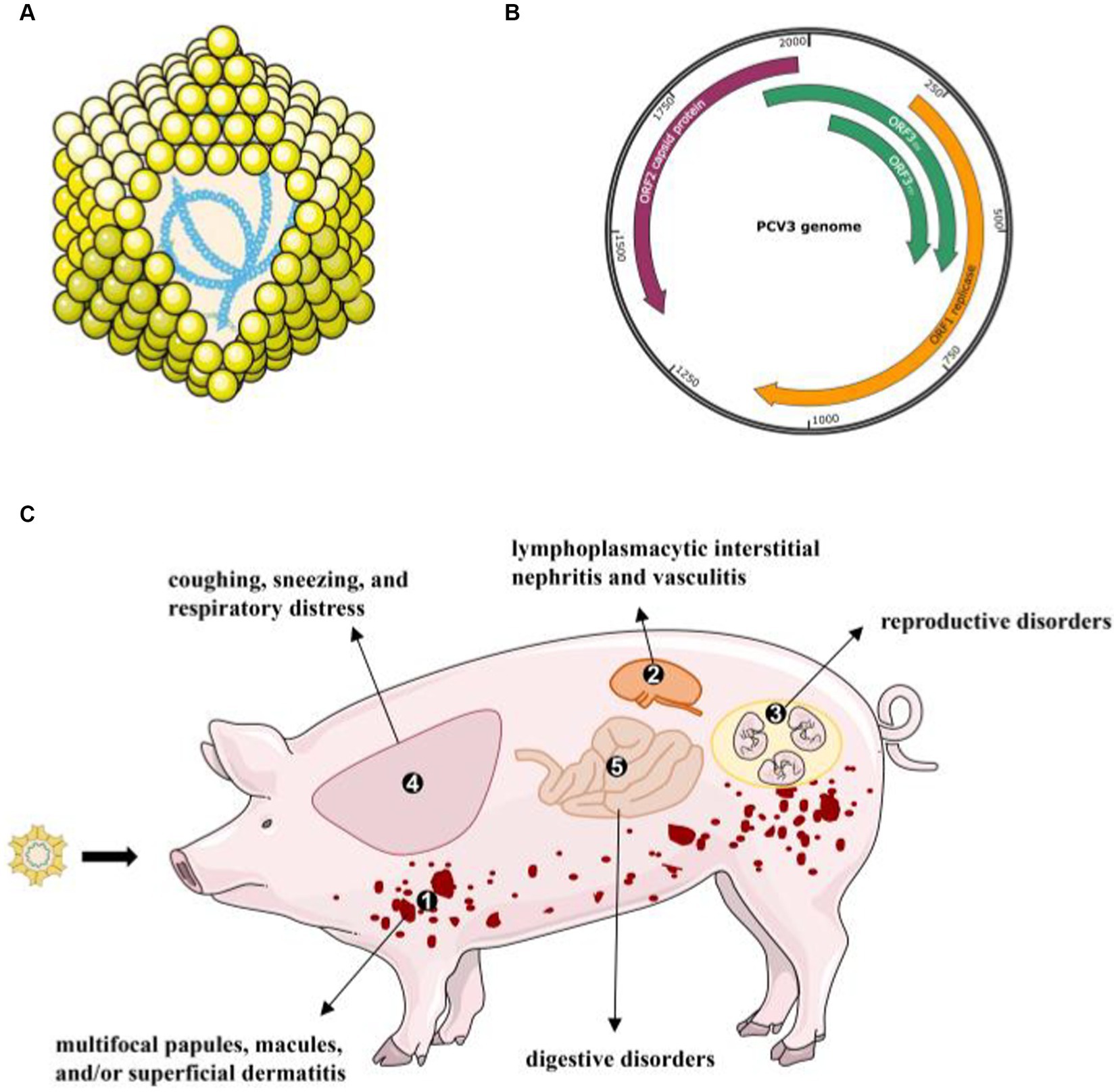 Figura 1. La estructura del virión de PCV3 y sus enfermedades o signos clínicos (A) PCV3 tiene un genoma de ADN circular monocatenario, y la cápside consta de 12 pentámeros pentagonales en forma de trompeta. (B) El genoma de PCV3 tiene una longitud de aproximadamente 2000 nt y contiene tres ORF principales: ORF1, ORF2 y ORF3 (45). (C) Una variedad de síntomas clínicos y patológicos están asociados con la infección por PCV3: (<>) piel: pápulas multifocales, máculas y/o dermatitis superficial; (<>) riñón: nefritis intersticial linfoplasmocítica y vasculitis; (<>) trastornos reproductivos: aborto, muerte fetal y fetos momificados; (<>) pulmón: tos, estornudos y dificultad respiratoria asociada con neumonía intersticial linfoplasmocítica; (<>) trastornos digestivos.
Figura 1. La estructura del virión de PCV3 y sus enfermedades o signos clínicos (A) PCV3 tiene un genoma de ADN circular monocatenario, y la cápside consta de 12 pentámeros pentagonales en forma de trompeta. (B) El genoma de PCV3 tiene una longitud de aproximadamente 2000 nt y contiene tres ORF principales: ORF1, ORF2 y ORF3 (45). (C) Una variedad de síntomas clínicos y patológicos están asociados con la infección por PCV3: (<>) piel: pápulas multifocales, máculas y/o dermatitis superficial; (<>) riñón: nefritis intersticial linfoplasmocítica y vasculitis; (<>) trastornos reproductivos: aborto, muerte fetal y fetos momificados; (<>) pulmón: tos, estornudos y dificultad respiratoria asociada con neumonía intersticial linfoplasmocítica; (<>) trastornos digestivos.
2.1 Inflamación multisistémica
La PCV3 se descubrió inicialmente en cerdas y fetos abortados que presentaban síntomas clínicos similares a los de la SDNT y disfunción reproductiva (6). El PDNS es principalmente perjudicial para los cerdos de destete y de engorde, con la aparición ocasional de lechones. La enfermedad tiene una baja tasa de mortalidad, pero una alta incidencia y un largo curso, y se caracteriza clínicamente por eritema irregular de pápulas multifocales y dermatitis superficial. En la actualidad, los estudios han demostrado que la enfermedad similar a la PDNS suele ser la forma más grave de la enfermedad que está presente en cerdos infectados natural y experimentalmente con PCV3 (15, 46, 47). En las cerdas se pueden observar lesiones típicas de la SDN, como vasculitis necrotizante y glomerulonefritis (48, 49). Las cerdas infectadas con PCV3 y los fetos abortados mostraron inflamación multiorgánica (39) y vasculitis multisistémica con un prominente tropismo cutáneo y renal causado por reacciones de hipersensibilidad tipo III (50). Se ha notificado una amplia gama de vasculitis, desde local hasta sistémica, en cerdos infectados por PCV3 (6, 15, 39, 44, 46, 51). Además, también se ha encontrado vasculitis en el corazón, los riñones y los tejidos intestinales de cerdos inoculados con PCV3 (51, 52). Además, la patología hepática, como la linfadenitis granulomatosa, se ha demostrado en el experimento de provocación con PCV3 (15, 53). Además, se detectó un alto nivel de quimiocinas y mediadores proinflamatorios en los cerdos infectados por PCV3 (15), y puede ser responsable de la inflamación multisistémica, de las respuestas inmunitarias del huésped y de las lesiones observadas anteriormente descritas.
2.2 Trastornos reproductivos
Aunque la PCV3 no siempre dio lugar a signos sintomáticos en las cerdas, el virus fue ampliamente colonizado en primerizas, lechones débiles, y en los fetos y la placenta (51, 54-58), la PCV3 también podría transmitirse verticalmente a través del calostro, el semen y la placenta (59). El trastorno reproductivo es una enfermedad que reduce el rendimiento de las cerdas, incluyendo la infertilidad, el aborto, la muerte fetal y la debilidad o malformación neonatal (60), afectando el desarrollo de la salud de la industria porcina mundial. Los estudios existentes han demostrado que se detectan altos niveles de PCV3 en granjas porcinas con fracaso reproductivo mediante pruebas de qPCR (40, 61), y se pueden encontrar títulos altos de PCV3 en los fetos abortados (46, 57), lo que indica que la infección por PCV3 podría afectar directamente a los fetos (44). Aunque la PCV3 puede detectarse en cerdas con o sin trastornos reproductivos, las tasas positivas de PCV3 son mucho más altas en cerdas con fallo reproductivo que en cerdas sanas mediante ensayos de qPCR (61). Además, se detectaron altas cargas virales de PCV3 mediante qPCR en los diversos tejidos, incluidos el corazón fetal, el timo, los ganglios linfáticos y la placenta, lo que sugiere un tropismo tisular adicional de PCV3 (62). Curiosamente, el virus también podría detectarse en múltiples tejidos de momias o fetos nacidos muertos, como las células del trofoblasto, la placenta y el cordón umbilical (21, 44, 62, 63). Por lo tanto, se pensó que la infección de los fetos contribuía al fracaso reproductivo inducido por PCV3. Sin embargo, el mecanismo subyacente al fracaso reproductivo inducido por PCV3 sigue sin estar claro y requiere más estudios.
2.3 Trastornos respiratorios o digestivos
Los cerdos, especialmente los lechones destetados, infectados con PCV3 suelen presentar diversos síntomas clínicos, incluyendo enfermedades respiratorias y trastornos digestivos (6, 35, 40, 64). Sin embargo, la asociación entre el estado circulante de PCV3 y la manifestación respiratoria clínica no se conoce bien. Zhai et al. analizaron sistémicamente y encontraron que la tasa de positividad de PCV3 se correlacionaba positivamente con los síntomas respiratorios (40). Del mismo modo, un porcentaje mucho mayor de cerdos destetados con diarrea fueron positivos para PCV3 que los no diarreicos (40). Otro informe llegó a una conclusión similar, había una estrecha asociación entre la infección por PCV3 y las enfermedades digestivas o respiratorias (35). Aunque los resultados anteriores demostraron que el PCV3 estaba potencialmente asociado con la enfermedad respiratoria y la diarrea porcina, era imprescindible presentar pruebas directas. Para responder a esta pregunta, se inoculó por vía intranasal a lechones libres de patógenos específicos (SPF) un virus PCV3 obtenido del clon de ADN infeccioso de Jiang et al. y encontraron que la infección por virus aumentaba las tasas de enfermedad respiratoria (15). En este estudio se observó una variedad de síntomas respiratorios clínicos, como tos, estornudos y dificultad respiratoria.
Estudios anteriores demostraron que la PCV3 era capaz de replicarse en los pulmones y que la infección por virus podía activar el sistema inmunitario innato y desencadenar la liberación de citoquinas inflamatorias (39, 65). Los signos respiratorios clínicos suelen ir acompañados de neumonía intersticial y pleuritis, así como de otros tipos de lesiones pulmonares (66). La neumonía provocará congestión alveolar y edema, y muchas secreciones inflamatorias en la cavidad alveolar afectarán el intercambio de oxígeno. Sin embargo, hay algunos resultados contradictorios. Los cerdos solo demostraron infección subclínica sin ningún cambio pulmonar patológico y no mostraron evidencia de replicación de PCV3 en el tejido pulmonar (51, 52). Otro estudio mostró que la infección por el virus PCV3 rescatada en ratones de Kunming no afectó a los tejidos u órganos de los grupos de control o infectados (67). Por el momento, no hay suficiente información sobre los factores que contribuyen al tropismo pulmonar y a la patogénesis de la PCV3.
Un informe muestra que existe una tendencia a la diarrea en lechones lactantes y destetados PCV3 positivos (42). Con base en inmunohistoquímica, se determinó que el antígeno PCV3 estaba presente en todos los órganos, incluido el tracto intestinal (65, 68). Consistentemente, los cerdos infectados desarrollaron diarrea, con degeneración y necrosis del epitelio del intestino delgado en el estudio de lesiones similares a PDNS (15). Los lechones inoculados con PCV3 mostraron un alto nivel de células positivas para PCV3 dentro de sus tejidos del intestino delgado (69). Los estudios experimentales de infección revelaron que los lechones inoculados con PCV3 mostraron una serie de lesiones en el intestino delgado, incluyendo atrofia de vellosidades y criptas, infiltración eosinofílica y linfocitaria, necrosis de células epiteliales de la mucosa y linfocitos, y un pequeño número de células plasmáticas (42, 69). La microbiota intestinal está formada por billones de bacterias y desempeña un papel fundamental en la modulación de la inmunidad del huésped; Los cambios en la microbiota están estrechamente relacionados con la progresión de las enfermedades. Por lo tanto, puede ser una forma eficaz de prevenir la infección por PCV3 a través de la regulación o los cambios de la microbiota intestinal.
3 Modulación inmunitaria de PCV3
La PCV3 ha sido ampliamente reportada en todo el mundo desde su descubrimiento y se caracteriza por lesiones típicas de PDNS. Desafortunadamente, solo se ha reportado recientemente un aislamiento exitoso de PCV3 en cultivo celular (70), y puede haber varios factores desconocidos asociados con la enfermedad. Los resultados cada vez mayores han demostrado que PCV3 desencadena la respuesta inmunitaria de las células huésped cuando invaden las células huésped (Figura 2). Se pueden encontrar altos niveles del genoma PCV3 en lechones abortados y nacidos débiles, especialmente en el timo y los ganglios linfáticos (62). Además, algunas detecciones en animales encontraron que PCV3 exhibe una infección persistente (18) o asintomática (63). En general, el virus tiende a replicarse en casi todos los tejidos de los cerdos, y prefiere replicarse en las células inmunitarias, y causa daños celulares específicos.
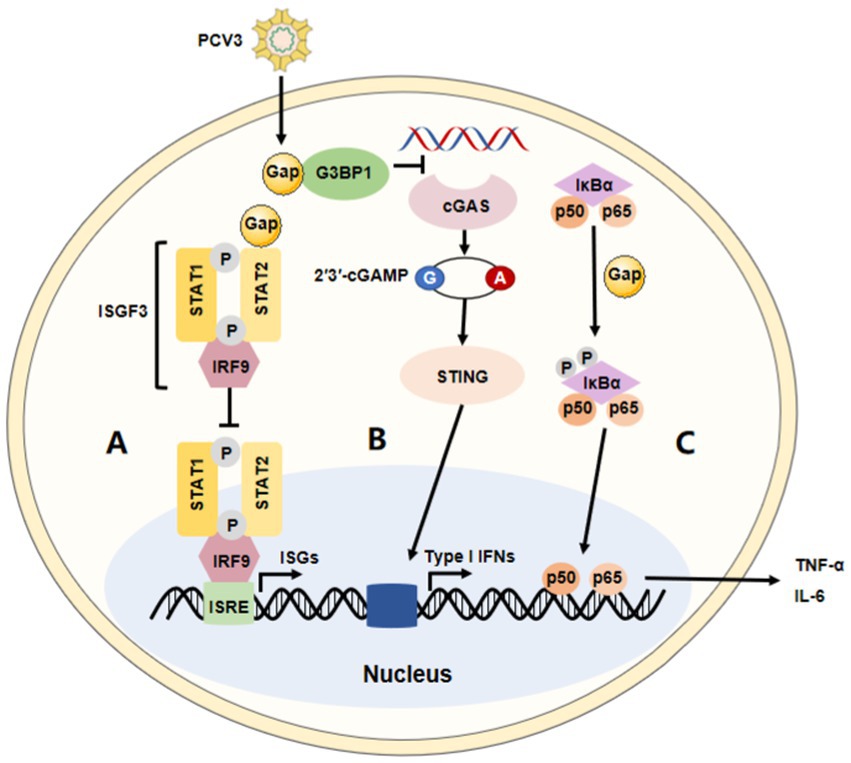 Figura 2. La modulación inmunitaria de PCV3. Resumen de las principales modulaciones inmunitarias de la PCV3. (A) PCV3 Cap interactúa con el dominio de transactivación de STAT2 e inhibe la activación del promotor ISRE inducida por IRF9-S2C, ayudando a PCV3 a escapar de la inmunidad innata del huésped meditada por interferón tipo I. (B) PCV3 Cap interactúa con G3BP1 para evitar que cGAS reconozca el ADN, lo que afecta la producción de interferón tipo I a través de la vía cGAS-STING. (C) La proteína PCV3 Cap induce la activación de NF-κB y regula al alza la expresión de citoquinas proinflamatorias.
Figura 2. La modulación inmunitaria de PCV3. Resumen de las principales modulaciones inmunitarias de la PCV3. (A) PCV3 Cap interactúa con el dominio de transactivación de STAT2 e inhibe la activación del promotor ISRE inducida por IRF9-S2C, ayudando a PCV3 a escapar de la inmunidad innata del huésped meditada por interferón tipo I. (B) PCV3 Cap interactúa con G3BP1 para evitar que cGAS reconozca el ADN, lo que afecta la producción de interferón tipo I a través de la vía cGAS-STING. (C) La proteína PCV3 Cap induce la activación de NF-κB y regula al alza la expresión de citoquinas proinflamatorias.
3.1 Respuesta inmunitaria innata
3.1.1 Respuesta al interferón de tipo I
Los diferentes virus emplean una amplia gama de estrategias defensivas para combatir la inmunidad innata. Sin embargo, las estrategias de evasión inmunitaria de la PCV3 no se comprenden completamente. Se ha reportado que la proteína Cap podría inhibir significativamente la actividad del elemento de respuesta estimulado por IFN (ISRE) al interactuar con el dominio de transactivación de transcripción (STAT2) (56, 71). Varios estudios han demostrado que PCV2 no solo activa la señalización de cGAS/STING y RIG-like receptor (RLR), sino que también aumenta la expresión de IFN-β, lo que promueve la proliferación de PCV2. Mientras tanto, en este proceso, sorprendentemente, no parece haber ninguna apariencia que impida su replicación a través de la vía de señalización NF-κB (72-74). Estos hallazgos son consistentes con los de los estudios sobre PCV3. Se ha reportado que la proteína PCV3 Cap inhibe la vía de señalización del interferón tipo I al inhibir la actividad del promotor de ISRE. Mecánicamente, Cap interactúa con el dominio de transactivación de STAT2 y se une a ISRE para evitar la unión de STAT2 e ISRE (71). Por lo tanto, PCV3 podría evadir la inmunidad innata del huésped mediada por IFN. Además, PCV3 Cap también puede interactuar con G3BP1 e inhibir la inducción de interferón tipo I (75). De acuerdo con el análisis proteómico, la infección por PCV3 en lechones con SPF infectados con clones infecciosos de PCV3 aumentó varios factores relacionados con IFN, como IFIT3, ISG15, etc., en el tejido pulmonar (47). Otro estudio realizado en células T HEK-293 encontró que, aunque no se observó que PCV3 Cap contribuyera a la expresión controlada de IFN-β, el nivel de ARNm de RIG-I/MDA5 podría aumentar significativamente con la proteína Cap en la vía RLRs (76). Por lo tanto, estos estudios indican que la regulación de la respuesta al interferón tipo I puede ser una estrategia importante para el escape inmune del huésped por PCV3. Además, un artículo reciente informó que la proteína PCV3 Cap está involucrada en la autofagia celular, otro mecanismo que se cree que es utilizado de manera efectiva por los virus para mejorar su replicación y persistencia (77). Mecánicamente, la proteína indujo la autofagia completa al inhibir la fosforilación de la diana de rapamicina en mamíferos en las células T HEK-293, incluida la formación de autofagosomas y vesículas autofágicas, así como la transformación de LC3-I a LC3-II (78). Por supuesto, la vía ubiquitina-proteasoma también está involucrada en este proceso. En definitiva, la información en esta área es necesaria para comprender el papel de la patogenicidad y la inmunidad innata durante la infección por PCV3.
3.1.2 Respuestas inflamatorias
Según estudios previos, existe una estrecha asociación entre la PCV3 y la inflamación multisistémica, así como las enfermedades respiratorias, la diarrea, la miocarditis, la encefalitis, la periarteritis, etc. (6, 34, 39, 44, 51, 67, 68, 79, 80). Los estudios han demostrado que las respuestas inflamatorias no controladas podrían contribuir a un daño significativo en cerdos infectados con PCV3 (51, 79). Se demostró que la infección por PCV3 tiene efectos reguladores sobre la expresión de IL-8 mediante la evaluación de la respuesta inmune innata in vivo (52). Significativamente, se observaron niveles más altos de mediadores proinflamatorios y quimiocinas en lechones infectados con PCV3 (15). En un estudio reciente, se demostró que PCV3 Cap es un factor crítico en la activación de la señalización NF-κB mediante la regulación positiva de mediadores proinflamatorios como RIG-I y MDA5 en las células T HEK-293 (76). Esto puede proporcionar una base para la patogénesis de PCV3 y la inmunidad innata del huésped. Por lo tanto, la inducción de un estado proinflamatorio crónico de la enfermedad da lugar a una desregulación de la inmunidad innata en cerdos infectados por PCV3, lo que puede proporcionar una posible explicación para los signos clínicos posteriores. La proteína PCV3 Cap parece activar algunas vías de señalización, pero el mecanismo aún no está claro y necesita más investigación.
3.2 Respuestas inmunitarias mediadas por células
Como consecuencia de la subversión inmune innata, la reducción de la respuesta de las células T, el compromiso de la capacidad de presentación de antígenos y el desequilibrio de la secreción inmunosupresora de citoquinas, la respuesta inmune adaptativa contra PCV2 se vio gravemente influenciada (81). Por ejemplo, las proteínas de PCV2 (como Cap y Rep) estimulan las células secretoras de IFN-γ específicas del antígeno (82-84). Mientras tanto, la infección de PCV2 podría afectar directamente a los timocitos inmaduros, por ejemplo, mediante la inhibición de la selección de timocitos, lo que resulta en alteraciones de la inmunidad de las células T auxiliares (85). Incluso cuando la respuesta inmunitaria a la PCV2 es baja, los cerdos siguen mostrando síntomas clínicos protectores, lo que indica que las respuestas inmunitarias mediadas por células, específicamente las respuestas mediadas por Th1, desempeñan un papel crucial en la inmunidad protectora. Un estudio en el que se utilizaron lechones destetados convencionales infectados con PCV3 recombinante y PBMC descubrió que eran incapaces de responder a la estimulación mitógena, pero no está claro si este efecto es permanente o si la energía de los linfocitos puede revertirse (15). Como resultado del análisis proteómico de cerdos infectados con PCV3, se encontró que tanto los loci SLA-I como II en el tejido pulmonar estaban significativamente regulados al alza (47). Al mismo tiempo, se ha reportado que la mayor parte de la afluencia de linfocitos está asociada con poblaciones de células T, independientemente de la respuesta periférica a PCV3 (52). Sin embargo, es necesario un estudio más detallado de la inmunidad celular mediada por PCV3 para profundizar en la patogénesis de PCV3.
3.3 Inmunopatogenia
Todos los tipos patógenos de PCV están muy extendidos y se han detectado tanto en cerdos sanos como enfermos. Tienen una variedad de manifestaciones clínicas y a menudo causan infecciones sistémicas crónicas. Los resultados de la investigación actual han sugerido que el trastorno de la homeostasis del sistema inmunitario puede ser el factor clave que conduce a la patogénesis de la infección por PCV. En experimentos de infección por PCV2, el virus podría interactuar con componentes del sistema inmunitario, incluidas las células inmunitarias, para perjudicar la inmunidad innata y adaptativa. La desregulación de la respuesta inmunitaria puede dar lugar a una serie de consecuencias graves, como la rápida regulación al alza de los factores proinflamatorios, la formación de complejos inmunitarios, la disminución de la capacidad de presentación de antígenos y la necrosis de linfocitos y células inmunitarias. Por ejemplo, varios estudios han demostrado que las infecciones de PCV3 y PCV2 están relacionadas con la formación de inmunocomplejos (15, 86). Aunque existen pocos informes sobre la inmunosupresión de PCV3 hasta la fecha, teniendo en cuenta la similitud de los síntomas clínicos y la patogenicidad entre PCV3 y PCV2, creemos que PCV3 también puede tener una fuerte inmunosupresión, especialmente en el caso de coinfección con otros virus, que también vale la pena explorar en el siguiente estudio.
4 Otros
4.1 Coinfección
En los últimos años, la infección mixta o coinfección multipatógena se ha vuelto más común en la práctica clínica, especialmente con una variedad de virus porcinos, incluidos los PCV. La coinfección no solo conduce a enfermedades más graves que cualquier infección por un solo virus, sino que también tiene un impacto negativo en el sistema inmunológico de los cerdos y agrava la complejidad de las enfermedades de las granjas porcinas. Hay informes de que las tasas de coinfección de PCV3 con el virus del síndrome respiratorio y reproductivo porcino, el virus de la peste porcina clásica, el virus de la pseudorrabia, el virus de la diarrea epidémica porcina (PEDV) y el parvovirus porcino (PPV) fueron de 36,36, 6,92, 14,53, 27,27 y 74,2% en algunas granjas porcinas, respectivamente (87-91), lo que indica que la coinfección de PCV3 con otros patógenos es común en las granjas porcinas. Además, la coinfección de PCV2 y PCV3 se reportó principalmente en granjas porcinas, aunque comparten un genoma similar y pertenecen al mismo género. Un estudio de muestras de suero de cerdos clínicamente sanos de los principales países europeos mostró una tasa positiva del 3% de coinfección PCV2-PCV3 en cerdos de engorde (92). Y un informe encontró que las tasas de coinfección por PCV2 y PCV3 aumentaron gradualmente del 3.4 % en el 2016 al 16.1 % en el 2018 en el Medio Oeste de los Estados Unidos (93). Además, los estudios han informado de que la tasa positiva de coinfección por PCV2 y PCV3 en diferentes regiones de China oscila entre el 6,78 y el 19,7% (94, 95). Estos resultados indican que la prevalencia de la coinfección por PCV2 y PCV3 está muy extendida en todo el mundo y ha aumentado gradualmente en los últimos años.
4.2 Vacunación
La vacunación ha demostrado ser eficaz y exitosa para la prevención de PCV2 y otros patógenos virales. Las vacunas comerciales contra la PCV2 son en su mayoría vacunas inactivadas o de subunidades basadas en Cap, que proporcionan una inmunidad protectora favorable contra otros tipos de PCV2 (68). ORF2 codifica la única proteína estructural de la cápside, y la identidad entre PCV2 y PCV3 fue menor. Por lo tanto, es posible que las vacunas basadas en PCV2 solo proporcionen una protección limitada contra PCV3. Como era de esperar, no hubo correlación entre la vacunación contra PCV2 y la circulación de PCV3, lo que indica que la protección cruzada entre PCV2 y PCV3 fue pobre (96). Sin embargo, no existen vacunas comerciales basadas en PCV3 para prevenir la infección por PCV3. Por lo tanto, es urgente desarrollar medidas eficaces como anticuerpos universales y vacunas para prevenir la enfermedad en el futuro.
5 Conclusión y perspectiva
El PCV3 estaba muy extendido en todo el mundo, incluidos Asia, Europa y América (8-13), según estudios epidemiológicos retrospectivos, aunque se descubrió por primera vez en cerdos con signos clínicos similares a PDNS en 2016 (6). En general, se considera patógena y se asocia con varios síntomas similares a los de la PCV2, como el PDNS, el fallo reproductivo y las enfermedades respiratorias. Es posible que los síntomas descritos en este artículo no sean los principales signos clínicos de la PCV3, y puede haber otros factores contribuyentes. Por lo tanto, el impacto de PCV3 sigue siendo controvertido hasta cierto punto, y se requiere más información para comprender su posible patogenicidad. Las células testiculares PK-15 y porcinas fueron susceptibles a PCV2 y se utilizaron para el aislamiento y propagación de PCV2 (97), pero la PCV3 en su mayoría no logró ser aislada y propagada en estas células de paso (70). Además, solo se informó de un modelo de infección por PCV3. Por lo tanto, las restricciones anteriores dificultan la investigación de la patogénesis de PCV3. Los datos acumulados indicaron que el PCV3 a menudo está coinfectado con PCV2, PPV, PEDV y otros patógenos, que no solo conducen a la gravedad de la enfermedad, sino que también se convierten en una seria amenaza para el desarrollo saludable del sistema de la industria porcina (56, 58, 98). Además, la infección por PCV3 tiene una baja tasa de mortalidad, pero una alta morbilidad y un largo curso de la enfermedad, lo que puede ser una gran amenaza para la industria porcina. Por lo tanto, es imperativo seguir evaluando e investigando sistemáticamente la prevalencia de la coinfección y la patogenicidad de la PCV3 con otros patógenos y desarrollar una vacuna eficaz contra la PCV3.
Contribuciones de los autores
DC: Curación de datos, Investigación, Metodología, Recursos, Redacción – borrador original, Redacción – revisión y edición. LZ: Conceptualización, Redacción – revisión y edición. SX: Adquisición de fondos, Administración de proyectos, Recursos, Supervisión, Validación, Redacción, revisión y edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Esta investigación fue apoyada por el Programa de Investigación y Desarrollo de la Comisión Municipal de Educación de Beijing (KM202310020004).
Conflicto de intereses
DC y LZ fueron empleados por los Institutos Zhongmu de China Animal Husbandry Industry Co., Ltd.
El autor restante declara que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un potencial conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Referencias
1. Tischer, I, Rasch, R y Tochtermann, G. Caracterización de partículas similares a papovavirus y picornavirus en líneas celulares permanentes de riñón de cerdo. Zentralbl Bakteriol Orig A. (1974) 226:153–67.
2. Tischer, I, Mields, W, Wolff, D, Vagt, M y Griem, W. Estudios sobre epidemiología y patogenicidad del circovirus porcino. Arch Virol. (1986) 91:271–6. doi: 10.1007/BF01314286
3. Allan, GM, Mc Neilly, F, Meehan, BM, Kennedy, S, Mackie, DP, Ellis, JA, et al. Aislamiento y caracterización de circovirus de cerdos con síndromes de emaciación en España, Dinamarca e Irlanda del Norte. Microbiol veterinario. (1999) 66:115–23. doi: 10.1016/S0378-1135(99)00004-8
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Wellenberg, GJ, Pesch, S, Berndsen, FW, Steverink, PJ, Hunneman, W, Van der Vorst, TJ, et al. Aislamiento y caracterización del circovirus porcino tipo 2 de cerdos que muestran signos de síndrome de emaciación multisistémica post-destete en los Países Bajos. Veterinario Q. (2000) 22:167–72. doi: 10.1080/01652176.2000.9695049
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Allan, GM, McNeilly, F, Kennedy, S, Daft, B, Clarke, EG, Ellis, JA, et al. Aislamiento de virus porcinos similares al circovirus de cerdos con una enfermedad de desgaste en EE.UU. y Europa. J Vet Diagn Investig. (1998) 10:3–10. doi: 10.1177/104063879801000102
6. Palinski, R, Piñeyro, P, Shang, P, Yuan, F, Guo, R, Fang, Y, et al. Un nuevo circovirus porcino relacionado lejanamente con circovirus conocidos se asocia con el síndrome de dermatitis y nefropatía porcina y el fracaso reproductivo. J Virol. (2017) 91:e01879. doi: 10.1128/JVI.01879-16
7. Zhang, HH, Hu, WQ, Li, JY, Liu, TN, Zhou, JY, Opriessnig, T, et al. Nuevas especies de circovirus identificadas en cerdos de granja designados como circovirus porcino 4, provincia de Hunan. China Transbound Emerg Dis. (2020) 67:1057–61. doi: 10.1111/tbed.13446
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Faccini, S, Barbieri, I, Gilioli, A, Sala, G, Gibelli, LR, Moreno, A, et al. Detección y caracterización genética del circovirus porcino tipo 3 en Italia. Transbound Emerg Dis. (2017) 64:1661–4. doi: 10.1111/tbed.12714
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Franzo, G, Legnardi, M, Hjulsager, CK, Klaumann, F, Larsen, LE, Segales, J, et al. La secuenciación del genoma completo de cepas de campo de circovirus porcino 3 de Dinamarca, Italia y España demuestra una gran heterogeneidad genética dentro de Europa. Transbound Emerg Dis. (2018) 65:602–6. doi: 10.1111/tbed.12836
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Ku, X, Chen, F, Li, P, Wang, Y, Yu, X, Fan, S, et al. Identificación y caracterización genética del circovirus porcino tipo 3 en China. Transbound Emerg Dis. (2017) 64:703–8. doi: 10.1111/tbed.12638
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Kwon, T, Yoo, SJ, Park, CK y Lyoo, YS. Prevalencia del nuevo circovirus porcino 3 en las poblaciones de cerdos coreanos. Microbiol veterinario. (2017) 207:178–80. doi: 10.1016/j.vetmic.2017.06.013
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Saraiva, GL, Vidigal, PMP, Assao, VS, Fajardo, MLM, Loreto, ANS, Fietto, JLR, et al. Detección retrospectiva y caracterización genética de cepas porcinas de circovirus 3 (PCV3) identificadas entre 2006 y 2007 en Brasil. Virus. (2019) 11:201. doi: 10.3390/v11030201
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Stadejek, T, Wozniak, A, Milek, D, y Biernacka, K. Primera detección de circovirus porcino tipo 3 en granjas porcinas comerciales en Polonia. Transbound Emerg Dis. (2017) 64:1350–3. doi: 10.1111/tbed.12672
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Chen, D, Huang, Y, Guo, Y, Wang, L, Zhang, Y, Zhou, L, et al. Análisis de prevalencia y evolución del circovirus porcino 3 en China de 2018 a 2022. Animales. (2022) 12:1588. doi: 10.3390/ani12121588
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Jiang, H, Wang, D, Wang, J, Zhu, S, She, R, Ren, X, et al. Inducción de dermatitis porcina y síndrome de nefropatía en lechones por infección por circovirus porcino tipo 3. J Virol. (2019) 93:e02018. doi: 10.1128/JVI.02045-18
16. Amoroso, MG, Serra, F, Esposito, C, D’Alessio, N, Ferrara, G, Cioffi, B, et al. Prevalencia de infección por circovirus porcino tipos 2 y 3 en la población de jabalíes de la región de Campania (sur de Italia). Animales. (2021) 11:3215. doi: 10.3390/ani11113215
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Franzo, G, Tucciarone, CM, Drigo, M, Cecchinato, M, Martini, M, Mondin, A, et al. Primer informe de susceptibilidad de jabalíes al circovirus porcino tipo 3: alta prevalencia en el Parque Regional de Colli Euganei (Italia) en ausencia de signos clínicos. Transbound Emerg Dis. (2018) 65:957–62. doi: 10.1111/tbed.12905
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. Klaumann, F, Dias-Alves, A, Cabezón, O, Mentaberre, G, Castillo-Contreras, R, López-Béjar, M, et al. El circovirus porcino 3 es muy prevalente en suero y tejidos y puede infectar persistentemente al jabalí (Sus scrofa scrofa). Transbound Emerg Dis. (2019) 66:91–101. doi: 10.1111/tbed.12988
19. Prinz, C, Stillfried, M, Neubert, LK y Denner, J. Detección de PCV3 en jabalíes alemanes. Virol J. (2019) 16:25. doi: 10.1186/s12985-019-1133-9
20. Souza, T, Gava, D, Schaefer, R, Leme, RA, Porto, GDS, y Alfieri, AA. Cepas porcinas de campo de circovirus 3a en jabalíes de vida libre en el estado de Paraná, Brasil. Animales. (2021) 11:1634. doi: 10.3390/ani11061634
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Dei Giudici, S, Franzoni, G, Bonelli, P, Angioi, PP, Zinellu, S, Deriu, V, et al. Caracterización genética de cepas de circovirus porcino 3 circulantes en cerdos y jabalíes sardos. Patógenos. (2020) 9:344. doi: 10.3390/pathogens9050344
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Tischer, I, Gelderblom, H, Vettermann, W, y Koch, MA. Virus porcino muy pequeño con ADN monocatenario circular. Naturaleza. (1982) 295:64–6. doi: 10.1038/295064a0
23. Zhang, D, He, K, Wen, L y Fan, H. Análisis genético y filogenético de una nueva cepa de circovirus porcino tipo 2 (PCV2) en China. Arch Virol. (2015) 160:3149–51. doi: 10.1007/s00705-015-2615-8
24. Yuzhakov, AG, Raev, SA, Alekseev, KP, Grebennikova, TV, Verkhovsky, OA, Zaberezhny, AD, et al. Primera detección y secuencia completa del genoma del circovirus porcino tipo 3 en Rusia. Genes de virus. (2018) 54:608–11. doi: 10.1007/s11262-018-1582-z
25. Guo, Z, Li, X, Deng, R y Zhang, G. Detección y características genéticas del circovirus porcino 3 basado en fluidos orales de cerdos asintomáticos en China Central. BMC Vet Res. (2019) 15:200. doi: 10.1186/s12917-019-1952-3
26. Li, G, He, W, Zhu, H, Bi, Y, Wang, R, Xing, G, et al. Origen, diversidad genética y dinámica evolutiva del nuevo circovirus porcino 3. Adv Sci. (2018) 5:1800275. doi: 10.1002/advs.201800275
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Li, G, Wang, H, Wang, S, Xing, G, Zhang, C, Zhang, W, et al. Conocimientos sobre la adaptabilidad genética y del huésped del circovirus porcino emergente 3. Virulencia. (2018) 9:1301–13. doi: 10.1080/21505594.2018.1492863
Resumen de PubMed | Texto completo de CrossRef | Google Académico
28. Rosario, K, Breitbart, M, Harrach, B, Segales, J, Delwart, E, Biagini, P, et al. Revisión de la taxonomía de la familia Circoviridae: establecimiento del género Cyclovirus y eliminación del género Gyrovirus. Arch Virol. (2017) 162:1447–63. doi: 10.1007/s00705-017-3247-y
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Mankertz, A, Caliskan, R, Hattermann, K, Hillenbrand, B, Kurzendoerfer, P, Mueller, B, et al. Biología molecular del circovirus porcino: análisis de la expresión génica y la replicación viral. Microbiol veterinario. (2004) 98:81–8. doi: 10.1016/j.vetmic.2003.10.014
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Rosario, K, Duffy, S, y Breitbart, M. Una guía de campo para los virus eucariotas circulares de ADN monocatenario: conocimientos obtenidos de la metagenómica. Arch Virol. (2012) 157:1851–71. doi: 10.1007/s00705-012-1391-y
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Nawagitgul, P, Harms, PA, Morozov, I, Thacker, BJ, Sorden, SD, Lekcharoensuk, C, et al. Ensayos de inmunoabsorción enzimática basados en circovirus porcino indirecto (PCV) tipo 2 modificado y basados en proteína de cápside recombinante (ORF2) para la detección de anticuerpos contra PCV. Clin Diagn Lab Immunol. (2002) 9:33–40. doi: 10.1128/CDLI.9.1.33-40.2002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Karuppannan, AK, y Kwang, J. ORF3 of porcine circovirus 2 enhance the in vitro and in vivo spread of the virus. Virología. (2011) 410:248–56. doi: 10.1016/j.virol.2010.11.009
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Lin, WL, Chien, MS, Wu, PC, Lai, CL y Huang, C. La proteína no estructural ORF2 del circovirus porcino tipo 3 induce la apoptosis en células mononucleares de sangre periférica porcina. Abrir Virol J. (2011) 5:148–53. doi: 10.2174/1874357901105010148
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Kim, SH, Park, JY, Jung, JY, Kim, HY, Park, YR, Lee, KK, et al. Detección y caracterización genética del circovirus porcino 3 de fetos abortados y cerdos con enfermedad respiratoria en Corea. J Vet Sci. (2018) 19:721–4. doi: 10.4142/jvs.2018.19.5.721
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Qi, S, Su, M, Guo, D, Li, C, Wei, S, Feng, L, et al. Detección molecular y análisis filogenético del circovirus porcino tipo 3 en 21 provincias de China durante 2015-2017. Transbound Emerg Dis. (2019) 66:1004–15. doi: 10.1111/tbed.13125
Resumen de PubMed | Texto completo de CrossRef | Google Académico
36. Fu, X, Fang, B, Ma, J, Liu, Y, Bu, D, Zhou, P, et al. Información sobre las características epidémicas y la historia evolutiva del nuevo circovirus porcino tipo 3 en el sur de China. Transbound Emerg Dis. (2018) 65:E296–303. doi: 10.1111/tbed.12752
37. Kroeger, M, Temeeyasen, G, y Piñeyro, PE. Cinco años de circovirus porcino 3: qué hemos aprendido sobre la enfermedad clínica, la patogenia inmunitaria y el diagnóstico. Virus Res. (2022) 314:198764. doi: 10.1016/j.virusres.2022.198764
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Sirisereewan, C, Thanawongnuwech, R, y Kedkovid, R. Comprensión actual de la patogénesis del circovirus porcino 3. Patógenos. (2022) 11:64. doi: 10.3390/pathogens11010064
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Phan, TG, Giannitti, F, Rossow, S, Marthaler, D, Knutson, TP, Li, L, et al. Detección de un nuevo circovirus PCV3 en cerdos con inflamación cardíaca y multisistémica. Virol J. (2016) 13:184. doi: 10.1186/s12985-016-0642-z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Zhai, SL, Zhou, X, Zhang, H, Hause, BM, Lin, T, Liu, R, et al. Epidemiología comparada del circovirus porcino tipo 3 en cerdos con diferentes presentaciones clínicas. Virol J. (2017) 14:222. doi: 10.1186/s12985-017-0892-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
41. Kedkovid, R, Woonwong, Y, Arunorat, J, Sirisereewan, C, Sangpratum, N, Lumyai, M, et al. Infección por circovirus porcino tipo 3 (PCV3) en cerdos de engorde de una granja tailandesa que padece complejo de enfermedad respiratoria porcina (PRDC). Microbiol veterinario. (2018) 215:71–6. doi: 10.1016/j.vetmic.2018.01.004
42. Zhang, F, Yuan, W, Li, Z, Zhang, Y, Zeng, X, Zhao, M, et al. Circovirus porcino tipo 3 en granjas porcinas con diarrea en Jiangxi, China: prevalencia, secuencia genómica y patogenicidad. Animales. (2020) 10:2324. doi: 10.3390/ani10122324
Resumen de PubMed | Texto completo de CrossRef | Google Académico
43. Chen, GH, Mai, KJ, Zhou, L, Wu, RT, Tang, XY, Wu, JL, et al. Detección y secuenciación del genoma del circovirus porcino 3 en cerdos neonatos con temblores congénitos en el sur de China. Transbound Emerg Dis. (2017) 64:1650–4. doi: 10.1111/tbed.12702
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Arruda, B, Piñeyro, P, Derscheid, R, Hause, B, Byers, E, Dion, K, et al. Enfermedad asociada a PCV3 en la cabaña porcina de los Estados Unidos. Los microbios emergentes infectan. (2019) 8:684–98. doi: 10.1080/22221751.2019.1613176
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Fux, R, Sockler, C, Link, EK, Renken, C, Krejci, R, Sutter, G, et al. La caracterización completa del genoma de los aislados de circovirus porcino tipo 3 revela la existencia de dos grupos distintos de cepas de virus. Virol J. (2018) 15:25. doi: 10.1186/s12985-018-0929-3
46. Bera, BC, Choudhary, M, Anand, T, Virmani, N, Sundaram, K, Choudhary, B, et al. Detección y caracterización genética del circovirus porcino 3 (PCV3) en cerdos de la India. Transbound Emerg Dis. (2020) 67:1062–7. doi: 10.1111/tbed.13463
47. Jiang, H, Wei, L, Wang, D, Wang, J, Zhu, S, She, R, et al. La proteómica cuantitativa basada en ITRAQ revela los primeros perfiles de proteoma de lechones infectados con circovirus porcino tipo 3. J Proteoma. (2020) 212:103598. doi: 10.1016/j.jprot.2019.103598
48. Wellenberg, GJ, Stockhofe-Zurwieden, N, de Jong, MF, Boersma, WJ y Elbers, AR. Los títulos excesivos de anticuerpos contra el circovirus porcino tipo 2 pueden desencadenar el desarrollo de dermatitis porcina y síndrome de nefropatía: un estudio de casos y controles. Microbiol veterinario. (2004) 99:203–14. doi: 10.1016/j.vetmic.2004.01.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
49. Opriessnig, T, Meng, XJ y Halbur, PG. Enfermedad asociada al circovirus porcino tipo 2: actualización de la terminología actual, manifestaciones clínicas, patogénesis, diagnóstico y estrategias de intervención. J Vet Diagn Investig. (2007) 19:591–615. doi: 10.1177/104063870701900601
Resumen de PubMed | Texto completo de CrossRef | Google Académico
50. Thibault, S, Drolet, R, Germain, MC, D’Allaire, S, Larochelle, R, y Magar, R. Vasculitis necrotizante cutánea y sistémica en cerdos. Veterinario Pathol. (1998) 35:108–16. doi: 10.1177/030098589803500204
Resumen de PubMed | Texto completo de CrossRef | Google Académico
51. Mora-Díaz, J, Piñeyro, P, Shen, H, Schwartz, K, Vannucci, F, Li, G, et al. Aislamiento de PCV3 a partir de casos perinatales y reproductivos de enfermedad asociada a PCV3 y caracterización in vivo de la replicación de PCV3 en cerdos en crecimiento con CD/CD. Virus. (2020) 12:219. doi: 10.3390/v12020219
Resumen de PubMed | Texto completo de CrossRef | Google Académico
52. Temeeyasen, G, Lierman, S, Arruda, BL, Main, R, Vannucci, F, Gimenez-Lirola, LG, et al. Patogenicidad y respuesta inmunitaria frente a la infección porcina por circovirus tipo 3 en cerdos derivados de cesáreas y privados de calostro. J Gen Virol. (2021) 102:1502. doi: 10.1099/jgv.0.001502
53. Zhang, J, Liu, Z, Zou, Y, Zhang, N, Wang, D, Tu, D, et al. Primera detección molecular de circovirus porcino tipo 3 en perros en China. Genes de virus. (2018) 54:140–4. doi: 10.1007/s11262-017-1509-0
54. Saporiti, V, Martorell, S, Cruz, TF, Klaumann, F, Correa-Fiz, F, Balasch, M, et al. Frecuencia de detección y análisis filogenético del circovirus porcino 3 (PCV-3) en cerdas primíparas y multíparas sanas y sus fetos momificados y mortinatos. Patógenos. (2020) 9:533. doi: 10.3390/pathogens9070533
Resumen de PubMed | Texto completo de CrossRef | Google Académico
55. Vargas-Bermúdez, DS, Vargas-Pinto, MA, Mogollón, JD, y Jaime, J. La infección de campo de una primeriza y su camada demuestra la transmisión vertical y el efecto sobre el fracaso reproductivo causado por el circovirus porcino tipo 3 (PCV3). BMC Vet Res. (2021) 17:150. doi: 10.1186/s12917-021-02862-5
Resumen de PubMed | Texto completo de CrossRef | Google Académico
56. Dal Santo, AC, Cezario, KC, Bennemann, PE, Machado, SA, y Martins, M. Secuencias del genoma completo del circovirus porcino 3 (PCV3) y alta prevalencia en fetos momificados de granjas comerciales en Brasil. Microb Pathog. (2020) 141:104027. doi: 10.1016/j.micpath.2020.104027
57. Saporiti, V, Valls, L, Maldonado, J, Pérez, M, Correa-Fiz, F, Segales, J, et al. Detección de circovirus porcino 3 en fetos abortados y lechones nacidos muertos a partir de casos de fallo reproductivo porcino. Virus. (2021) 13:264. doi: 10.3390/v13020264
Resumen de PubMed | Texto completo de CrossRef | Google Académico
58. Serena, MS, Cappuccio, JA, Barrales, H, Metz, GE, Aspitia, CG, Lozada, I, et al. Primera detección y caracterización genética del circovirus porcino tipo 3 (PCV3) en Argentina y su asociación con el fracaso reproductivo. Transbound Emerg Dis. (2021) 68:1761–6. doi: 10.1111/tbed.13893
Resumen de PubMed | Texto completo de CrossRef | Google Académico
59. Chen, S, Zhang, L, Li, X, Niu, G y Ren, L. Avances recientes en epidemiología y patobiología del circovirus porcino 3. Virus. (2021) 13:1944. doi: 10.3390/v13101944
Resumen de PubMed | Texto completo de CrossRef | Google Académico
60. Sánchez, RE, Nauwynck, HJ, McNeilly, F, Allan, GM, y Pensaert, MB. Infección por circovirus porcino 2 en fetos porcinos inoculados en diferentes etapas de gestación. Microbiol veterinario. (2001) 83:169–76. doi: 10.1016/S0378-1135(01)00425-4
61. Zou, Y, Zhang, N, Zhang, J, Zhang, S, Jiang, Y, Wang, D, et al. Detección molecular y análisis de secuencia de circovirus porcino tipo 3 en sueros de cerdas de granjas con antecedentes prolongados de problemas reproductivos en Hunan. China Arch Virol. (2018) 163:2841–7. doi: 10.1007/s00705-018-3914-7
Resumen de PubMed | Texto completo de CrossRef | Google Académico
62. Deim, Z, Dencso, L, Erdelyi, I, Valappil, SK, Varga, C, Posa, A, et al. Detección de circovirus porcino tipo 3 en una granja porcina húngara con fallos reproductivos. Rec. Veterinario (2019) 185:84. doi: 10.1136/vr.104784
63. Zheng, S, Wu, X, Zhang, L, Xin, C, Liu, Y, Shi, J, et al. Aparición de circovirus porcino 3 sin signos de infección clínica en la provincia de Shandong. Transbound Emerg Dis. (2017) 64:1337–41. doi: 10.1111/tbed.12667
64. Yang, K, Zhang, M, Liu, Q, Cao, Y, Zhang, W, Liang, Y, et al. Epidemiología y evolución de los virus emergentes similares a los circovirus porcinos en cerdos con síntomas de disentería hemorrágica y diarrea en China Central de 2018 a 2021. Virus. (2021) 13:2282. doi: 10.3390/v13112282
Resumen de PubMed | Texto completo de CrossRef | Google Académico
65. De Conti, ER, Resende, TP, Marshall-Lund, L, Rovira, A, y Vannucci, FA. Lesiones histológicas y sitios de replicación de PCV3 en cerdos infectados de forma natural. Animales. (2021) 11:1520. doi: 10.3390/ani11061520
Resumen de PubMed | Texto completo de CrossRef | Google Académico
66. Saporiti, V, Cruz, TF, Correa-Fiz, F, Núñez, JI, Sibila, M, y Segales, J. Frecuencia similar de detección de circovirus porcino 3 (PCV-3) en muestras de suero de cerdos afectados por trastornos digestivos o respiratorios y cerdos clínicamente sanos de la misma edad. Transbound Emerg Dis. (2020) 67:199–205. doi: 10.1111/tbed.13341
Resumen de PubMed | Texto completo de CrossRef | Google Académico
67. Jiang, Z, Wu, J, Jiang, M, Xie, Y, Bu, W, Liu, C, et al. Una técnica novedosa para la construcción de la clonación infecciosa del circovirus porcino tipo 3. Microbiol frontal. (2020) 11:1067. doi: 10.3389/fmicb.2020.01067
Resumen de PubMed | Texto completo de CrossRef | Google Académico
68. Opriessnig, T, Karuppannan, AK, Castro, A, y Xiao, CT. Circovirus porcinos: estado actual, lagunas de conocimiento y desafíos. Virus Res. (2020) 286:198044. doi: 10.1016/j.virusres.2020.198044
69. Hou, L, Wang, J, Zhang, W, Quan, R, Wang, D, Zhu, S, et al. Alteraciones dinámicas de la microbiota intestinal en lechones porcinos infectados por circovirus tipo 3. Microbiol frontal. (2020) 11:1360. doi: 10.3389/fmicb.2020.01360
Resumen de PubMed | Texto completo de CrossRef | Google Académico
70. Oh, T, y Chae, C. Primer aislamiento y caracterización genética del circovirus porcino tipo 3 utilizando células primarias de riñón porcino. Microbiol veterinario. (2020) 241:108576. doi: 10.1016/j.vetmic.2020.108576
Resumen de PubMed | Texto completo de CrossRef | Google Académico
71. Shen, H, Liu, X, Zhang, P, Wang, S, Liu, Y, Zhang, L, et al. El circovirus porcino 3 Cap inhibe la señalización del interferón tipo I a través de la interacción con STAT2. Virus Res. (2020) 275:197804. doi: 10.1016/j.virusres.2019.197804
72. Medzhitov, R. Reconocimiento de microorganismos y activación de la respuesta inmunitaria. Naturaleza. (2007) 449:819–26. doi: 10.1038/nature06246
73. Dixit, V, y Mak, TW. Señalización NF-kappaB. Son muchos los caminos que conducen a Madrid. Células. (2002) 111:615–9. doi: 10.1016/S0092-8674(02)01166-2
74. Shen, H, Liu, X, Zhang, P, Wang, L, Liu, Y, Zhang, L, et al. Caracterización genómica de un circovirus porcino tipo 3 en el sur de China. Transbound Emerg Dis. (2018) 65:264–6. doi: 10.1111/tbed.12639
75. Zhang, P, Shen, H, Liu, X, Wang, S, Liu, Y, Xu, Z, et al. El circovirus porcino tipo 3 Cap inhibe la inducción de interferón tipo I a través de la interacción con G3BP1. Front Vet Sci. (2020) 7:594438. doi: 10.3389/fvets.2020.594438
Resumen de PubMed | Texto completo de CrossRef | Google Académico
76. Liu, X, Shen, H, Zhang, X, Liang, T, Ban, Y, Yu, L, et al. La proteína de la cápside del circovirus porcino tipo 3 induce la activación de NF-kappaB y regula al alza la expresión de citoquinas proinflamatorias en las células HEK-293T. Arch Virol. (2021) 166:2141–9. doi: 10.1007/s00705-021-05104-z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
77. Parzych, KR, y Klionsky, DJ. Una visión general de la autofagia: morfología, mecanismo y regulación. Señal redox de antioxidante. (2014) 20:460–73. doi: 10.1089/ars.2013.5371
Resumen de PubMed | Texto completo de CrossRef | Google Académico
78. Geng, SC, Li, XL y Fang, WH. La proteína de la cápside del circovirus porcino 3 induce la autofagia en las células HEK293T al inhibir la fosforilación de la diana de la rapamicina en mamíferos. J Zhejiang Univ Sci B. (2020) 21:560–70. doi: 10.1631/jzus. B1900657
Resumen de PubMed | Texto completo de CrossRef | Google Académico
79. Sun, J, Wei, L, Lu, Z, Mi, S, Bao, F, Guo, H, et al. Estudio retrospectivo de la infección porcina por circovirus 3 en China. Transbound Emerg Dis. (2018) 65:607–13. doi: 10.1111/tbed.12853
Resumen de PubMed | Texto completo de CrossRef | Google Académico
80. Franzo, G, Delwart, E, Fux, R, Hause, B, Su, S, Zhou, J, et al. Genotipado del circovirus porcino 3 (PCV-3) en la actualidad: ¿tiene sentido? Virus. (2020) 12:265. doi: 10.3390/v12030265
Resumen de PubMed | Texto completo de CrossRef | Google Académico
81. Yang, N, Li, J, Yang, Q, Qiao, J, Cui, D, Liu, F, et al. Reducción de la capacidad de presentación de antígenos y modificación de la expresión de citoquinas inflamatorias/inmunosupresoras de células dendríticas derivadas de monocitos inducidas a partir de sangre periférica de lechones infectados con circovirus porcino tipo 2. Arch Virol. (2018) 163:1231–9. doi: 10.1007/s00705-018-3735-8
Resumen de PubMed | Texto completo de CrossRef | Google Académico
82. Fort, M, Sibila, M, Nofrarias, M, Pérez-Martín, E, Olvera, A, Mateu, E, et al. Circovirus porcino tipo 2 (PCV2) Las proteínas cap y rep están implicadas en el desarrollo de la inmunidad mediada por células tras la infección por PCV2. Veterinario Inmunol Inmunopatíaco. (2010) 137:226–34. doi: 10.1016/j.vetimm.2010.05.013
Resumen de PubMed | Texto completo de CrossRef | Google Académico
83. Jung, BK, Kim, HR, Lee, YH, Jang, H, y Chang, KS. Comparación de las respuestas inmunitarias a las vacunas PCV2 Replicasa-Capsid y Capsid Virus-like particle en ratones. J Microbiol Biotechnol. (2019) 29:482–8. doi: 10.4014/jmb.1809.09032
84. Koinig, HC, Talker, SC, Stadler, M, Ladinig, A, Graage, R, Ritzmann, M, et al. La vacunación contra PCV2 induce células T coproductoras de IFN-gamma/TNF-alfa con un papel potencial en la protección. Res. Veterinaria. (2015) 46:20. doi: 10.1186/s13567-015-0157-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
85. Klausmann, S, Sydler, T, Summerfield, A, Lewis, FI, Weilenmann, R, Sidler, X, et al. Reprogramación de células T a través de la expresión dirigida de correceptores CD4 y receptores de células T en timocitos maduros por infecciones latentes de células circovirales porcinas tipo 2 de miembros de la familia Circoviridae en el timo. Los microbios emergentes infectan. (2015) 4:e15. doi: 10.1038/emi.2015.15
86. Langohr, IM, Stevenson, GW, Nelson, EA, Lenz, SD, HogenEsch, H, Wei, H, et al. Lesiones vasculares en cerdos infectados experimentalmente con circovirus porcino tipo 2 serogrupo B. Vet Pathol. (2010) 47:140–7. doi: 10.1177/0300985809352793
87. Zheng, LL, Chai, LY, Tian, RB, Zhao, Y, Chen, HY y Wang, ZY. Detección simultánea del virus del síndrome respiratorio y reproductivo porcino y del circovirus porcino 3 mediante PCR dúplex en tiempo real SYBR green capital I, con sede en Ucrania. Sondas de células molares. (2020) 49:101474. doi: 10.1016/j.mcp.2019.101474
Resumen de PubMed | Texto completo de CrossRef | Google Académico
88. Zheng, HH, Zhang, SJ, Cui, JT, Zhang, J, Wang, L, Liu, F, et al. Detección simultánea del virus de la peste porcina clásica y del circovirus porcino 3 mediante PCR cuantitativa de fluorescencia en tiempo real dúplex basada en el verde I de SYBR. Sondas de células molares. (2020) 50:101524. doi: 10.1016/j.mcp.2020.101524
89. Tian, RB, Jin, Y, Xu, T, Zhao, Y, Wang, ZY y Chen, HY. Desarrollo de un ensayo de PCR en tiempo real dúplex basado en SYBR verde I para la detección del virus de la pseudorrabia y el circovirus porcino 3. Sondas de células molares. (2020) 53:101593. doi: 10.1016/j.mcp.2020.101593
Resumen de PubMed | Texto completo de CrossRef | Google Académico
90. Han, HY, Zheng, HH, Zhao, Y, Tian, RB, Xu, PL, Hou, HL, et al. Desarrollo de un ensayo de PCR cuantitativa de fluorescencia en tiempo real dúplex basado en SYBR Green I para la detección simultánea del virus de la diarrea epidémica porcina y el circovirus porcino 3. Sondas de células molares. (2019) 44:44–50. doi: 10.1016/j.mcp.2019.02.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
91. Ha, Z, Xie, CZ, Li, JF, Wen, SB, Zhang, KL, Nan, FL, et al. Detección molecular y caracterización genómica del circovirus porcino 3 en cerdos del noreste de China. BMC Vet Res. (2018) 14:321. doi: 10.1186/s12917-018-1634-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
92. Saporiti, V, Huerta, E, Correa-Fiz, F, Grosse Liesner, B, Duran, O, Segales, J, et al. Detección y genotipado de circovirus porcino 2 (PCV-2) y detección de circovirus porcino 3 (PCV-3) en sueros procedentes de cerdos de engorde de diferentes países europeos. Transbound Emerg Dis. (2020) 67:2521–31. doi: 10.1111/tbed.13596
Resumen de PubMed | Texto completo de CrossRef | Google Académico
93. Wang, Y, Noll, L, Lu, N, Porter, E, Stoy, C, Zheng, W, et al. Diversidad genética y prevalencia del circovirus porcino tipo 3 (PCV3) y tipo 2 (PCV2) en el Medio Oeste de los EE. UU. durante 2016-2018. Transbound Emerg Dis. (2020) 67:1284–94. doi: 10.1111/tbed.13467
Resumen de PubMed | Texto completo de CrossRef | Google Académico
94. Xia, D, Huang, L, Xie, Y, Zhang, X, Wei, Y, Liu, D, et al. La prevalencia y la diversidad genética de los circovirus porcinos tipos 2 y 3 en el noreste de China de 2015 a 2018. Arch Virol. (2019) 164:2435–49. doi: 10.1007/s00705-019-04336-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
95. Xu, T, Zhang, YH, Tian, RB, Hou, CY, Li, XS, Zheng, LL, et al. Prevalencia y análisis genético del circovirus porcino tipo 2 (PCV2) y tipo 3 (PCV3) entre 2018 y 2020 en China Central. Infectar Genet Evol. (2021) 94:105016. doi: 10.1016/j.meegid.2021.105016
Resumen de PubMed | Texto completo de CrossRef | Google Académico
96. Woźniak, A, Miłek, D, Bąska, P, y Stadejek, T. ¿El circovirus porcino tipo 3 (PCV3) interfiere con la eficacia de la vacuna contra el circovirus porcino tipo 2 (PCV2)? Transbound Emerg Dis. (2019) 66:1454–61. doi: 10.1111/tbed.13221
Resumen de PubMed | Texto completo de CrossRef | Google Académico
97. Roca, M, Balasch, M, Segales, J, Calsamiglia, M, Viaplana, E, Urniza, A, et al. Caracterización in vitro e in vivo de un clon infeccioso de una cepa europea de circovirus porcino tipo 2. J Gen Virol. (2004) 85:1259–66. doi: 10.1099/vir.0.79836-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
98. Mai, J, Wang, D, Zou, Y, Zhang, S, Meng, C, Wang, A, et al. Alto estado de coinfección del nuevo parvovirus porcino 7 con circovirus porcino 3 en cerdas que experimentaron fallos reproductivos. Front Vet Sci. (2021) 8:695553. doi: 10.3389/fvets.2021.695553
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: circovirus porcino 3 (PCV3), enfermedad asociada al circovirus porcino (PCVAD), avance, patogenicidad, modulación inmune
Cita: Chen D, Zhang L y Xu S (2023) Patogenicidad y modulación inmunitaria del circovirus porcino 3. Frente. Vet. Sci. 10:1280177. doi: 10.3389/fvets.2023.1280177
Recibido: 19 de agosto de 2023; Aceptado: 25 de octubre de 2023;
Publicado: 27 de noviembre de 2023.
Editado por:
Francesca Grippi, Instituto Zooprofiláctico Experimental de Sicilia (IZSSi), Italia
Revisado por:
Sonalika Mahajan, Instituto Indio de Investigación Veterinaria (IVRI), India
Nader Sobhy, Universidad de Zagazig, Egipto
Derechos de autor © 2023 Chen, Zhang y Xu. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Shengkui Xu, skxu0721@163.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía