Primera detección y caracterización molecular del virus del síndrome respiratorio y reproductivo porcino
Primera detección y caracterización molecular del virus del síndrome respiratorio y reproductivo porcino en Namibia, África
 Umberto Molini1,2*
Umberto Molini1,2*  Lauren M. Coetzee2,3†
Lauren M. Coetzee2,3†  María Y. Hemberger1
María Y. Hemberger1  Bernard Chiwome1
Bernard Chiwome1  Sigfrido Khaiseb2
Sigfrido Khaiseb2  William G. Dundon4
William G. Dundon4  Giovanni Franzo5
Giovanni Franzo5- 1Facultad de Medicina Veterinaria, Facultad de Ciencias de la Salud y Medicina Veterinaria, Universidad de Namibia, Windhoek, Namibia
- 2Laboratorio Veterinario Central (CVL), Windhoek, Namibia
- 3Facultad de Medicina Veterinaria, Universidad de Teramo, Teramo, Italia
- 4Laboratorio de Producción y Sanidad Animal, Sección de Producción y Sanidad Animal, Departamento de Ciencias y Aplicaciones Nucleares, División Mixta FAO/OIEA, Organismo Internacional de Energía Atómica, Viena (Austria)
- 5Departamento de Medicina, Producción y Sanidad Animal, Universidad de Padua, Legnaro, Legnaro, Italia
Introducción: El sector porcino en África desempeña un papel importante en las economías locales, contribuyendo a la mitigación de la pobreza y la subsistencia de la comunidad. Además, la agricultura intensiva está adquiriendo progresivamente más importancia en la región. Por lo tanto, cualquier enfermedad que afecte a las poblaciones porcinas puede tener efectos perjudiciales en las comunidades locales. El Síndrome Reproductivo y Respiratorio Porcino (PRRS) es una de las enfermedades infecciosas más importantes que afectan a los cerdos en todo el mundo, pero la información sobre su epidemiología en África es extremadamente limitada.
Material y métodos: En el presente estudio, 147 cerdos sanos sacrificados, procedentes de 15 granjas intensivas y rurales de Namibia, fueron analizados mediante RT-PCR y los genes ORF7 de las muestras positivas fueron secuenciados para su posterior caracterización genética y análisis filogenético. Además, 55 jabalíes también fueron evaluados utilizando el mismo enfoque.
Resultados: En total, 7 de los 147 cerdos (4,76%) dieron positivo, todos ellos procedentes de 3 granjas rurales (con una frecuencia de detección dentro de la piara superior al 14%) caracterizadas por fuertes vínculos epidemiológicos. Todas las muestras industriales de cerdos y jabalíes fueron negativas. El análisis de la secuencia reveló que todas las cepas pertenecían a la especie Betaarterivirus suid1, anteriormente conocida como PRRSV tipo I, y probablemente fueron importadas de Europa hace al menos 6 años, evolucionando de forma independiente a partir de entonces. No se pudo determinar cuándo y cómo se produjo la primera introducción debido a la ausencia de otras secuencias africanas para la comparación.
Discusión: El presente trabajo proporciona la primera detección y caracterización de la epidemiología molecular del PRRSV en Namibia. Sobre la base de los presentes hallazgos, la presencia del PPRSV parece marginal y limitada a las explotaciones domésticas. Si bien las medidas de bioseguridad aplicadas en las explotaciones industriales parecen ser eficaces para prevenir la introducción del virus, la circulación del PRRSV en entornos rurales sigue representando una amenaza potencial y, teniendo en cuenta las implicaciones socioeconómicas de las enfermedades del ganado que disminuyen el rendimiento de los animales en las zonas rurales, debe fomentarse la vigilancia activa para actuar con prontitud contra las amenazas emergentes y garantizar el bienestar de las poblaciones porcinas locales.
1 Introducción
La industria porcina está experimentando un crecimiento significativo en varios países africanos, lo que refleja un aumento de la demanda interna. Aproximadamente el 5% de los cerdos de todo el mundo se crían en África, principalmente en la región subsahariana. La producción porcina en Namibia, aunque no es la mayor del continente, desempeña un papel importante en la sociedad y la economía locales (1). Las grandes granjas comerciales están dispersas por todo el país y se caracterizan por medidas avanzadas de gestión y bioseguridad, especialmente dirigidas a la prevención de la introducción de la peste porcina africana (PPA) (2). Sin embargo, la mayor parte de la producción porcina local se basa en actividades de pequeños agricultores. La producción porcina contribuye significativamente a la reducción de la pobreza, al empleo femenino y juvenil y garantiza la subsistencia y el bienestar de las familias y las comunidades en entornos rurales y periurbanos (3, 4). Según los datos facilitados por la Unión Agrícola de Namibia (NAU), la producción local de carne de cerdo en Namibia supone entre el 45 y el 50% del consumo, siendo el déficit cubierto por las importaciones procedentes de Europa, siendo Alemania y España las principales fuentes. En agosto de 2022 se prohibieron las importaciones procedentes de Sudáfrica debido a la fiebre aftosa. Un total de 14.752 cerdos fueron sacrificados en Namibia entre enero y abril de 2023. Hay una población aproximada de 40.000 cerdos en Namibia, ubicados principalmente en tres distritos: Mariental, Windhoek y Tsumeb.
Ningún país africano ha comenzado todavía a exportar carne de porcino, aunque se sabe que existe un comercio razonable de animales vivos y productos derivados del cerdo a nivel regional.
El impacto de las enfermedades en los cerdos puede tener enormes consecuencias económicas para los medios de subsistencia de los agricultores y la generación de ingresos, tanto a nivel familiar como comunitario y regional. El impacto de las enfermedades se traduce en pérdidas de ingresos para los agricultores y en el posible cierre de los mercados (4).
En Namibia se ha documentado la presencia y amplia circulación de varios patógenos porcinos (5-8). Sin embargo, no se dispone de información sobre una de las enfermedades víricas más devastadoras que afectan a la industria porcina en los países de altos ingresos: el Síndrome Respiratorio y Reproductivo Porcino (PRRS) (9, 10). El PRRS es causado por dos especies virales, Betaarterivirus suid 1 y Betaarterivirus suid 2, pertenecientes al género Betaarterivirus, familia Arteriviridae.1 Estos virus se conocían anteriormente como virus del síndrome respiratorio y reproductivo porcino 1 (PRRSV-1), o de tipo europeo, y virus del síndrome respiratorio y reproductivo porcino 2 (PRRSV-2), o de tipo americano (11). Se caracterizan por un genoma de ARN monocatenario de sentido positivo [ssRNA(+)] de aproximadamente 15 kb. Alrededor de tres cuartas partes del genoma están ocupadas por el marco de lectura abierto (ORF) 1a y ORF1ab, que codifican 14 proteínas no estructurales, mientras que la región terminal consta de ocho ORF parcialmente superpuestos (ORF2a, ORF2b, ORF3, ORF4, ORF5, ORF5a, ORF6 y ORF7) que codifican las proteínas estructurales (12). Al igual que otros virus de ARN, el PRRSV muestra una alta tasa evolutiva y de recombinación, lo que provoca la aparición continua de nuevas variantes sobre las que pueden actuar la deriva genética y las presiones selectivas, lo que conduce a la heterogeneidad genética, fenotípica y biológica observada de las cepas circulantes (13-15). Debido a esta alta variabilidad y rápida evolución, la secuenciación de regiones genómicas relativamente cortas es suficiente para proporcionar información útil sobre la epidemiología molecular de estos virus. Entre los diferentes ORF, el ORF5 y el ORF7 son los más utilizados para reconstruir los vínculos epidemiológicos entre granjas porcinas, productores comerciales, regiones, países, etc. (16-18).
La infección por PRRSV es responsable de trastornos reproductivos en las cerdas, signos respiratorios, disminución de la ganancia media diaria y mortalidad en animales en crecimiento/engorde, aunque el impacto puede variar en función de la especie y la cepa víricas, así como del estado general del huésped y del estado inmunitario. Los costos asociados con el manejo de la enfermedad por virus de la peste de pequeños rumiantes también incluyen los costos de los antimicrobianos relacionados con una mayor susceptibilidad a las infecciones secundarias, las estrategias de control y la vacunación (19).
A pesar de la relevancia de esta infección, solo se dispone de datos limitados de África (20). No se disponía de información sobre el PPPRSV en Namibia antes de este estudio. Namibia alberga una biodiversidad notable, incluidas varias especies silvestres que pueden entrar en contacto con animales domésticos infectados. Además de los cerdos domésticos, el jabalí es el único otro huésped que ha demostrado ser susceptible a la infección por el PRRSV, aunque en general se considera que el impacto del PRRSV en la fauna silvestre es limitado. Sin embargo, actualmente no se dispone de información sobre el impacto del virus de la peste de pequeños rumiantes en las especies silvestres africanas, especialmente en aquellas que han demostrado ser susceptibles a las infecciones por otros patógenos porcinos (5-7).
Para colmar esta laguna de información, se recogieron varias muestras de poblaciones de cerdos rurales y comerciales, así como de jabalíes salvajes, para evaluar la presencia del PRRSV en Namibia y caracterizar genéticamente las cepas que pudieran detectarse.
2 Materiales y métodos
2.1 Recogida y procesamiento de muestras
Se recogieron muestras (amígdalas o ganglios linfáticos) de 147 cerdos sanos sacrificados (de 5 a 6 meses de edad, aproximadamente 75-100 kg) entre marzo de 2018 y mayo de 2023. Todos los animales procedían de 15 granjas porcinas, que comprendían 3 instalaciones industriales y 12 operaciones de traspatio. Estas granjas de cerdos estaban ubicadas en seis regiones diferentes de Namibia: Khomas, Hardap, Kunene, Omaheke, Otjozondjupa y Erongo. Todas las granjas de cerdos de traspatio involucradas en el estudio consistieron en 30 a 100 animales y observaron un nivel medio de bioseguridad. Por el contrario, las tres granjas industriales de cerdos mantenían rebaños de aproximadamente 1.400 animales y se adherían a un alto y estricto nivel de bioseguridad.
Además, las amígdalas de 55 jabalíes, que viven en la zona propiedad de cuatro explotaciones ganaderas de las regiones de Khomas y Otjozondjupa, y que se someten a campañas periódicas de caza, se recogieron en el momento del sacrificio entre junio de 2019 y junio de 2023, y se incluyeron en el estudio.
La toma de muestras fue realizada por un veterinario con especialización en Salud Pública Veterinaria durante la inspección post mortem en el matadero. Todas las muestras se recogieron en recipientes herméticos estériles y debidamente etiquetados. Los tejidos se extrajeron de los cadáveres utilizando bisturíes desechables estériles y pinzas de metal, que se flamearon cuidadosamente con un quemador Bunsen portátil para cada muestra. Después de la recolección, todas las muestras fueron transportadas refrigeradas al laboratorio (+4°C). Las amígdalas o ganglios linfáticos se homogeneizaron en 1 mL de solución salina estéril tamponada con fosfato (PBS) utilizando el TissueLyser LT (Qiagen, Alemania). El ARN total se extrajo de las muestras homogeneizadas utilizando el High Pure Viral Nucleic Acid Kit (Hoffman-La Roche, Suiza) con un volumen de elución de 100 μL, siguiendo las instrucciones del fabricante.
El ORF7 de cada muestra se amplificó utilizando un método de RT-PCR de un solo paso, como lo describen Oleksiewicz et al. (21). En resumen, ORF7 se amplificó utilizando el par de cebadores ORF7F (5′ GCC CCT GCC CAG CAC G 3′) y ORF7R (5′ TCG CCC TAA TTG AAT AGG TGA 3′), lo que resultó en un amplicón de 637 pb (21). Se aplicó el siguiente perfil térmico: transcripción inversa a 50 °C durante 30 min, desnaturalización inicial a 94 °C durante 2 min, seguida de 40 ciclos de desnaturalización a 94 °C durante 15 s, recocido a 55 °C durante 20 s y elongación a 68 °C durante 50 s. A esto le siguió un paso final de elongación a 68 °C durante 10 minutos.
2.2 Secuenciación y análisis filogenético
Los amplicones de las muestras positivas se purificaron utilizando un sistema de limpieza de gel y PCR Wizard SV (Promega) y se secuenciaron comercialmente por LGC Genomics (Berlín, Alemania). Las secuencias de las muestras positivas se editaron y ensamblaron utilizando el paquete de software Staden versión 2.0.0b8. Todas las secuencias obtenidas se enviaron a la base de datos GenBank con los números de acceso OR604620-OR604626.
Las secuencias de ORF7 se analizaron preliminarmente utilizando BLAST (22) y posteriormente se descargó una colección de mil secuencias relacionadas y se alineó con las de Namibia utilizando MAFFT (23). Se reconstruyó un árbol filogenético de máxima verosimilitud utilizando IQ-TREE (24) seleccionando el modelo de sustitución con el menor criterio de información de Akaike (AIC), calculado por JModelTest (25). Se realizaron diez mil réplicas ultrarrápidas de bootstrap para evaluar la fiabilidad del clado.
El tiempo transcurrido entre el origen del clado namibio y su detección se estimó mediante la realización de un análisis coalescente seriado utilizando BEAST 1.10 (26), seleccionando el lognormal relajado (27) y el Skygrid (28) como parámetro de reloj molecular y tamaño de la población viral, respectivamente. Para cada análisis, se realizó una ejecución independiente de 100 millones de generaciones. Los resultados se analizaron utilizando el trazador 1.7 (29) después de la eliminación de un quemado del 20% y se aceptaron solo si el tamaño de la muestra estimada (ESS) era superior a 200 y la convergencia y la mezcla eran adecuadas. La estimación de los parámetros se resumió en términos de media y 95% de densidad posterior más alta (95HPD). Los árboles de máxima credibilidad del clado (MCC) se construyeron y anotaron utilizando TreeAnnotator (paquete BEAST).
3 Resultados
Siete de 147 cerdos (4,76%; Tabla 1) procedentes de 3 de las 15 granjas porcinas que participaron en el estudio dieron positivo para PRRSV mediante RT-PCR. El gen ORF7 se secuenció con éxito en todas las muestras positivas. Ninguna de las 55 muestras de jabalíes dio positivo para el PRRSV. Entre las 15 granjas porcinas, solo tres instalaciones de traspatio ubicadas en la región de Hardap dieron positivo para el PRRSV, con una prevalencia dentro del rebaño que osciló entre el 14,29 y el 30%, mientras que ninguna de las granjas industriales mostró evidencia de PRRSV.
El análisis BLAST demostró que las secuencias provenían de virus que pertenecían a la especie Betaarterivirus suid1, con un porcentaje de identidad de aproximadamente el 95% con la secuencia más estrechamente relacionada.
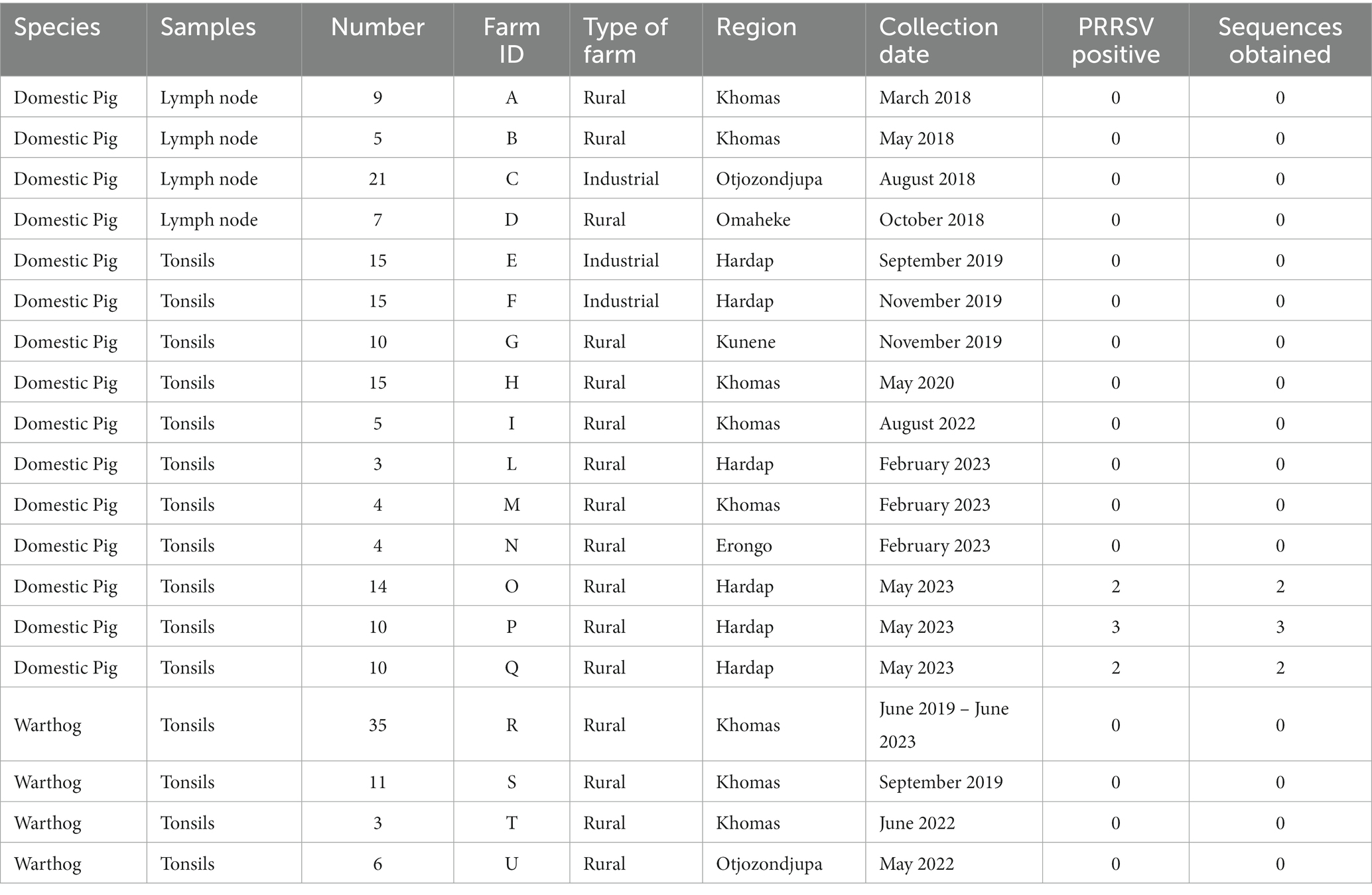
El análisis filogenético confirmó que las cepas de Namibia formaban parte de una larga rama procedente de un conglomerado europeo, incluidos los virus recogidos, en particular, en Italia (Figura 1).
 Figura 1. Árbol filogenético de máxima verosimilitud basado en una colección de secuencias completas de ORF7. El clado namibio se ha resaltado en rojo, mientras que el clado utilizado para el análisis filodinámico (véase la Figura 2) se muestra en azul.
Figura 1. Árbol filogenético de máxima verosimilitud basado en una colección de secuencias completas de ORF7. El clado namibio se ha resaltado en rojo, mientras que el clado utilizado para el análisis filodinámico (véase la Figura 2) se muestra en azul.
Basado en la distancia genética y la tasa evolutiva estimada (es decir, 6.029∙10−3 [95HPD, 5.188∙10−3 –6.922∙10−3] sustitución/sitio/año), se calculó una evolución independiente de al menos 5,961 años (media = 8,011; [95HPD, 5.961–9.743]; Figura 2).
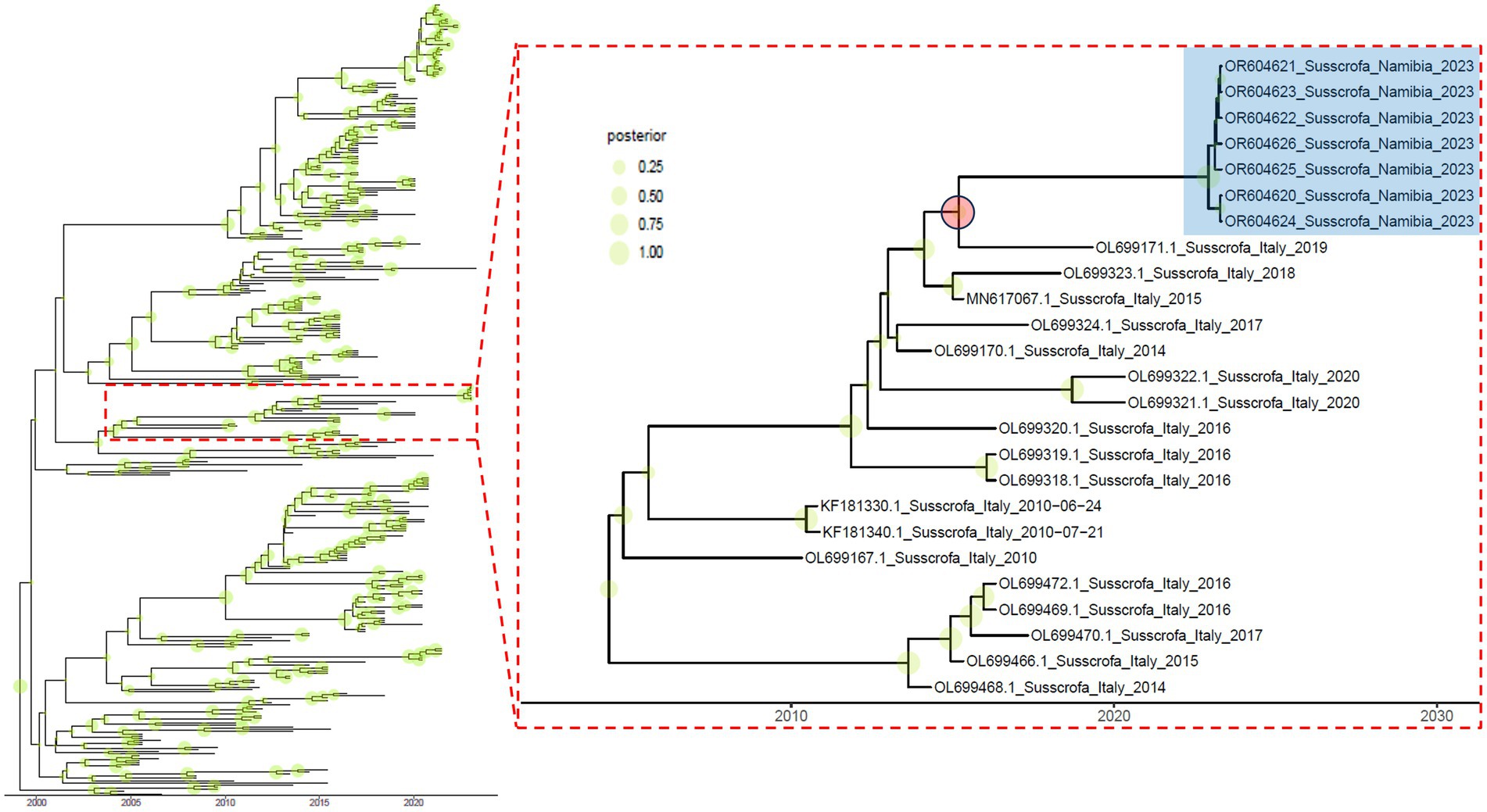 Figura 2. Árbol filogenético de credibilidad máxima del clado calibrado en el tiempo basado en cepas de Namibia y afines. Los círculos verdes sombreados cuyo tamaño es proporcional a la probabilidad posterior se informan en el nodo del árbol correspondiente. El subclado que contiene las cepas de Namibia (resaltado en azul) se indica en el prospecto derecho. El círculo rojo indica el ancestro probable y el tiempo de divergencia relativa.
Figura 2. Árbol filogenético de credibilidad máxima del clado calibrado en el tiempo basado en cepas de Namibia y afines. Los círculos verdes sombreados cuyo tamaño es proporcional a la probabilidad posterior se informan en el nodo del árbol correspondiente. El subclado que contiene las cepas de Namibia (resaltado en azul) se indica en el prospecto derecho. El círculo rojo indica el ancestro probable y el tiempo de divergencia relativa.
4 Discusión
El PRRS es una de las enfermedades porcinas más importantes a nivel mundial, que supone una enorme carga económica para la industria porcina (19). Aunque la porcicultura intensiva africana solo tiene un impacto marginal en el escenario global, tanto las granjas comerciales como las de traspatio juegan un papel significativo en la sociedad local, con implicaciones en el bienestar y el sustento de la población que van más allá de la mera relevancia económica (4). La fuente de proteínas de alta calidad, el alivio de la pobreza y el empoderamiento de las mujeres son algunos ejemplos de los beneficios multifacéticos de la producción porcina en varios países africanos, incluida Namibia. Por lo tanto, cualquier enfermedad que dañe a este sector puede tener graves efectos perjudiciales a una escala más amplia. A pesar del impacto del virus del coronavirus en África, la información sobre la epidemiología del virus del PRRSV en África es casi inexistente. El primer informe del virus se remonta a 2004 en Sudáfrica, seguido de un segundo brote en 2007 (20). Algunos estudios más recientes han revelado una alta frecuencia de detección del PRRSV en Uganda y Nigeria, por métodos serológicos y moleculares (30-33). Entre abril de 2018 y diciembre de 2019, se notificó una frecuencia de PRRSV tipo 1 y tipo 2 de 24,65 y 2,73 %, respectivamente, mediante un ensayo comercial de RT-PCR en tiempo real en Uganda (33).
El presente estudio reporta la presencia de PRRSV en la población porcina de Namibia por primera vez y, lo que es más significativo, la primera caracterización genética de cepas africanas.
La prevalencia general fue significativamente menor que la notificada en los países de ingresos altos y en el estudio anterior de Uganda. Ninguna de las muestras analizadas procedentes de granjas industriales fue positiva, lo que sugiere la eficacia de las medidas de bioseguridad aplicadas para controlar la PPA, pero también para prevenir la introducción del PRRSV (8, 34). Este hallazgo confirma lo que ya se ha notificado para el PCV-3 en Namibia. Por otro lado, se observó que tres granjas rurales estaban infectadas, con una frecuencia de detección dentro de la granja superior al 14%. Las cepas identificadas eran genéticamente idénticas. Debido a la alta tasa evolutiva (es decir, 6.029∙10−3 [95HPD: 5.188∙10–3 −6.922∙10−3] sustitución/sitio/año) del PRRSV estimado en este documento y de acuerdo con estudios previos (15), este hallazgo sugiere una introducción extremadamente reciente. Las tres granjas tenían fuertes vínculos epidemiológicos entre ellas debido al intercambio de algunos verracos con fines de cría, una rutina que, por lo tanto, debería desalentarse enérgicamente y reemplazarse por el uso de semen certificado. Por lo tanto, la rápida propagación de la cepa identificada es muy plausible y encaja bien con el notable potencial de difusión del PRRSV ya demostrado en granjas intensivas de países de ingresos altos.
En comparación con el escenario internacional, no se pudo detectar ninguna secuencia estrechamente relacionada. Sobre la base de la tasa evolutiva estimada del PRRSV, se pueden plantear hipótesis de al menos 6 años de evolución independiente. Las secuencias relacionadas más cercanas formaban parte de un clado italiano de PRRV tipo I. Sin embargo, hay que subrayar que Italia estaba ampliamente sobrerrepresentada en el conjunto de datos de secuencias ORF7. Por lo tanto, estas conclusiones deben evaluarse con cautela, ya que no se produce ningún comercio de cerdos o productos conexos entre Italia y Namibia. Por otro lado, la circulación del PRRSV entre los países europeos es significativa (14). Por lo tanto, se puede proponer una introducción de cepas europeas con un mayor grado de confianza, como ya se ha visto para varios otros patógenos animales, incluidos los de los cerdos. No se observó la importación de otros continentes, en particular de Asia, a menudo descrita para otros patógenos (35-38), pero sin embargo debe considerarse e investigarse con estudios más extensos.
Desde 2019 está prohibida la importación namibia de semen y animales vivos procedentes de Europa y Sudáfrica con fines reproductivos. Sin embargo, una introducción anterior desde Europa encaja bien con la larga e independiente evolución estimada para el clado actual. No se puede determinar dónde y cómo se produjo este evento de introducción debido a la ausencia de datos moleculares comparables de otros países africanos. El informe sobre el PRRSV tipo I en Uganda (33) puede sugerir que el PRRSV se introdujo en otros países africanos que tienen una industria porcina más desarrollada y que luego se extendió a Namibia a través de movimientos transfronterizos de animales. Sin embargo, tampoco se puede excluir una introducción directa en Namibia. Deberían realizarse muestreos y secuenciaciones más intensivos de los PRRSV en otros países africanos para comprender mejor la epidemiología molecular del virus en el continente y comprender su dinámica poblacional, sus patrones de introducción y propagación.
Solo unas pocas granjas, ubicadas en un área geográfica restringida y con fuertes vínculos epidemiológicos entre sí, se mostraron infectadas, en ausencia de signos clínicos detectables. Esta evidencia, combinada con el estatus de libre de PRRSV de las granjas intensivas, sugiere una circulación limitada del virus en Namibia. Desafortunadamente, a pesar de los esfuerzos, la financiación y el cumplimiento de los agricultores impidieron una investigación más exhaustiva en el estudio actual. Del mismo modo, la inclusión de más regiones de Namibia sería útil para aumentar la representatividad de los datos obtenidos. Por lo tanto, se necesitan más estudios para obtener una comprensión más completa del escenario epidemiológico, la caracterización de los factores de riesgo y el cálculo del impacto económico real.
Finalmente, no se encontró evidencia de la presencia de PRRSV en jabalíes, lo que apoya el tropismo restringido del huésped de este virus. Por otra parte, los niveles aparentemente bajos de circulación del PRRSV en Namibia también pueden explicar por qué no se ha detectado el virus en otras especies.
El presente trabajo es el primer paso en el estudio y caracterización de la epidemiología molecular del PRRSV en África. Aunque en Namibia se ha demostrado la presencia de cepas de tipo I del PRRSV, muy probablemente originarias de Europa, su importancia parece marginal y limitada a las explotaciones domésticas. Si bien las medidas de bioseguridad, como el acceso limitado de personas y vehículos, la limpieza y desinfección periódicas de las instalaciones, el mantenimiento de un rebaño cerrado, la compartimentación de las explotaciones, la presencia de vallas alrededor de las instalaciones y la inspección diaria de los animales aplicadas en las granjas industriales parecen eficaces para prevenir la introducción del virus, la circulación del virus del virus de la peste de pequeños rumiantes en las explotaciones rurales sigue representando una amenaza potencial para las explotaciones industriales. como se ha demostrado anteriormente en los países de ingresos altos (17). Además, debido a las consecuencias socioeconómicas de las enfermedades del ganado que disminuyen el rendimiento de los animales incluso en las zonas rurales, se debe sugerir un seguimiento activo para actuar rápidamente contra las amenazas emergentes y garantizar el bienestar de la población local.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres de los repositorios y los números de acceso se pueden encontrar en: https://www.ncbi.nlm.nih.gov/genbank/, OR604620, OR604621, OR604622, OR604623, OR604624, OR604625, OR604626.
Declaración ética
Los estudios en animales fueron aprobados por el Comité de Ética Descentralizado de Neudamm. Los estudios se llevaron a cabo de acuerdo con la legislación local y los requisitos institucionales. No se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio debido a que las muestras utilizadas para la actividad de investigación «primera detección y caracterización molecular del virus reproductivo y respiratorio porcino (PRRSV) en Namibia, África» fueron muestras secundarias obtenidas de cerdos domésticos y jabalíes, que fueron recolectadas originalmente durante proyectos de investigación anteriores realizados en mataderos porcinos de Namibia y autorizadas por la Descentralizada de Neudamm Comité de Ética de la Universidad de Namibia. Por lo tanto, no se requirió una aprobación ética específica para este estudio.
Contribuciones de los autores
UM: Conceptualización, Obtención de Fondos, Investigación, Recursos, Supervisión, Redacción – borrador original, Redacción – revisión y edición. LC: Curación de datos, Análisis formal, Redacción – revisión y edición. MH: Curación de datos, redacción, revisión y edición. BC: Curación de datos, redacción, revisión y edición. SK: Curación de datos, administración de proyectos, redacción, revisión y edición. WD: Adquisición de fondos, Metodología, Recursos, Redacción – revisión y edición. GF: Conceptualización, Curación de datos, Análisis formal, Metodología, Software, Redacción – borrador original, Redacción – revisión y edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Esta labor contó con el apoyo del Departamento de Medicina, Producción y Sanidad Animal de la Universidad de Padua [subvención número BIRD225455/22] y de la Red VETLAB de la Iniciativa sobre Usos Pacíficos (PUI) del OIEA. Las secuencias se generaron por conducto de los Servicios de Secuenciación del subprograma de Producción y Sanidad Animal de la División de la Organización Mixta de las Naciones Unidas para la Agricultura y la Alimentación de las Naciones Unidas y el Organismo Internacional de Energía Atómica (OIEA).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Notas
1. ^https://ictv.global/taxonomy
Referencias
1. María Luisa Penrith. Demografía de los cerdos en África En: Curso Regional de Capacitación (África) Análisis del riesgo de importación de la peste porcina africana. Organización Mundial de Sanidad Animal (OMSA). (2021).
2. Fasina, F. Una visión general de las cadenas de valor de la producción y comercialización porcina, África occidental y central. Organización Mundial de Sanidad Animal (OMSA). (2021).
3. Okai, E. K. (2019). Grandes oportunidades para los criadores de cerdos en África Occidental. Disponible en: https://www.thepigsite.com/articles/big-opportunities-for-pig-farmers-in-west-africa (consultado el 10 de octubre de 2023).
4. Weka, R, Bwala, D, Adedeji, Y, Ifende, I, Davou, A, Ogo, N, et al. Rastreo de los cerdos domésticos en África En: G Kušec e ID Kušec, editores. Rastreo del cerdo doméstico. Rijeka: IntechOpen (2021)
5. Molini, U, Coetzee, LM, Hemberger, MY, Khaiseb, S, Cattoli, G, y Dundon, WG. Pruebas que indican la transmisión del parvovirus porcino 1 entre jabalíes y cerdos domésticos en Namibia. Vet Res Commun. (2023) 47:981–5. doi: 10.1007/s11259-022-10038-1
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Molini, U, Franzo, G, Gous, L, Moller, S, Hemberger, YM, Chiwome, B, et al. Se identificaron tres genotipos diferentes de circovirus porcino 2 (PCV-2) en cerdos y jabalíes en Namibia. Arch Virol. (2021) 166:1723–8. doi: 10.1007/s00705-021-05035-9
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Molini, U, Franzo, G, Settypalli, TBK, Hemberger, MY, Khaiseb, S, Cattoli, G, et al. Coinfecciones virales de jabalíes en Namibia con el virus de la peste porcina africana y el parvovirus porcino 1. Animales. (2022) 12:1697. doi: 10.3390/ani12131697
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Molini, U, Marruchella, G, Matheus, F, Hemberger, YM, Chiwome, B, Khaiseb, S, et al. Investigación molecular de la infección por circovirus porcino tipo 3 en cerdos en Namibia. Patógenos. (2021) 10:585. doi: 10.3390/pathogens10050585
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Nieuwenhuis, N, Duinhof, TF y Van Nes, A. Papers: análisis económico de los brotes del virus del síndrome respiratorio y reproductivo porcino en nueve rebaños de cerdas. Rec. Veterinario (2012) 170:225. doi: 10.1136/vr.100101
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Segalés, J, y Mateu, E. Un mundo, una salud: la amenaza de las infecciones virales emergentes y reemergentes de los cerdos. Transbound Emerg Dis. (2012) 59:1–2. doi: 10.1111/j.1865-1682.2011.01303.X
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Ruedas-Torres, I, Rodríguez-Gómez, IM, Sánchez-Carvajal, JM, Larenas-Muñoz, F, Pallarés, FJ, Carrasco, L, et al. El rompecabezas de la virulencia del PRRSV. Microbiol veterinario. (2021) 260:109168. doi: 10.1016/J.VETMIC.2021.109168
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Dokland, T. La biología estructural del PRRSV. Virus Res. (2010) 154:86–97. doi: 10.1016/j.virusres.2010.07.029
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Duffy, S. ¿Por qué las tasas de mutación del virus de ARN son tan altas? PLoS Biol. (2018) 16:E3000003. doi: 10.1371/journal.pbio.3000003
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Franzo, G, Faustini, G, Legnardi, M, Cecchinato, M, Drigo, M, y Tucciarone, CM. Reconstrucción filodinámica y filogeográfica del virus del síndrome respiratorio y reproductivo porcino (PRRSV) en Europa: patrones y determinantes. Transbound Emerg Dis. (2022) 69:E2175–84. doi: 10.1111/tbed.14556
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Stadejek, T, Stankevicius, A, Murtaugh, MP, y Oleksiewicz, MB. Evolución molecular del PRRSV en Europa: estado actual de la cuestión. Microbiol veterinario. (2013) 165:21–8. doi: 10.1016/j.vetmic.2013.02.029
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Balka, G, Podgórska, K, Brar, MS, Bálint, Á, Cadar, D, Celer, V, et al. Diversidad genética del PRRSV 1 en Europa Central y Oriental en 1994-2014: origen y evolución del virus en la región. Sci Rep. (2018) 8:7811. DOI: 10.1038/s41598-018-26036-w
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Franzo, G, Barbierato, G, Pesente, P, Legnardi, M, Tucciarone, CM, Sandri, G, et al. Epidemiología del síndrome respiratorio y reproductivo porcino (Prrs) en una empresa porcina integrada del norte de Italia: una amenaza multinivel que requiere intervenciones multinivel. Virus. (2021) 13:2510. doi: 10.3390/v13122510
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Shi, M, Lemey, P, Singh Brar, M, Suchard, MA, Murtaugh, MP, Carman, S, et al. La propagación del virus del síndrome respiratorio y reproductivo porcino tipo 2 (PRRSV) en América del Norte: un enfoque filogeográfico. Virología. (2013) 447:146–54. doi: 10.1016/j.virol.2013.08.028
19. Montaner-Tarbes, S, del Portillo, HA, Montoya, M, y Fraile, L. Lagunas clave en el conocimiento del virus del síndrome reproductivo respiratorio porcino (PRRSV). Front Vet Sci. (2019) 6:435543. doi: 10.3389/FVETS.2019.00038/BIBTEX
20. Beltrán-Alcrudo, D, Lubroth, J, Njeumi, F, Pinto, J, DeLaRocque, S, Martin, V, et al. Concienciación regional sobre el síndrome reproductivo y respiratorio porcino (PRRS). FAO. (2007) 2. doi: 10.1002/9781119506287.ch26
21. Oleksiewicz, MB, Bøtner, A, Madsen, KG y Storgaard, T. Detección sensible y tipificación del virus del síndrome respiratorio y reproductivo porcino mediante la amplificación por RT-PCR de genes virales completos. Microbiol veterinario. (1998) 64:7–22. doi: 10.1016/S0378-1135(98)00254-5
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Madden, T. La herramienta de análisis de secuencia BLAST En: La herramienta de análisis de secuencia BLAST. Estados Unidos: Centro Nacional de Información Biotecnológica (2013). 1–17.
23. Standley, K. Software de alineación de secuencia múltiple MAFFT versión 7: mejoras en el rendimiento y la facilidad de uso (describe la versión 7). Mol Biol Evol. (2013) 30:772–80. DOI: 10.1093/molbev/mst010
24. Nguyen, LT, Schmidt, HA, Von Haeseler, A, y Minh, BQ. IQ-TREE: un algoritmo estocástico rápido y eficaz para estimar filogenias de máxima verosimilitud. Mol Biol Evol. (2015) 32:268–74. DOI: 10.1093/Molbev/MSU300
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Darriba, D, Taboada, GL, Doallo, R, y Posada, D. JModelTest 2: más modelos, nuevas heurísticas y computación paralela. Métodos Nat. (2012) 9:772. doi: 10.1038/nmeth.2109
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Suchard, MA, Lemey, P, Baele, G, Ayres, DL, Drummond, AJ y Rambaut, A. Integración de datos filogenéticos y filodinámicos bayesianos utilizando BEAST 1.10. Virus Evol. (2018) 4:vey016. doi: 10.1093/ve/vey016
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Drummond, AJ, Ho, SYW, Phillips, MJ y Rambaut, A. Filogenética relajada y datación con confianza. PLoS Biol. (2006) 4:E88-E710. doi: 10.1371/journal.pbio.0040088
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Hill, V, y Baele, G. Estimación bayesiana de la dinámica poblacional pasada en BEAST 1.10 utilizando el modelo coalescente Skygrid. Mol Biol Evol. (2019) 36:2620–8. doi: 10.1093/molbev/msz172
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Rambaut, A, Drummond, AJ, Xie, D, Baele, G, y Suchard, MA. Resumen posterior en filogenética bayesiana utilizando el trazador 1.7. Syst Biol. (2018) 67:901–4. doi: 10.1093/sysbio/syy032
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Aiki-Raji, CO, Adebiyi, AI, Abiola, JO y Oluwayelu, DO. Prevalencia del virus del síndrome respiratorio y reproductivo porcino y anticuerpos contra el parvovirus porcino en cerdos comerciales, suroeste de Nigeria. Beni Suef Univ J Aplicación Básica de Ciencias. (2018) 7:80–3. doi: 10.1016/j.bjbas.2017.07.006
31. Dione, M, Masembe, C, Akol, J, Amia, W, Kungu, J, Lee, HS, et al. La importancia de la bioseguridad en las granjas: seroprevalencia y factores de riesgo de patógenos bacterianos y virales en los sistemas de pequeños productores porcinos en Uganda. Acta Trop. (2018) 187:214–21. doi: 10.1016/j.actatropica.2018.06.025
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Meseko, C, y Oluwayelu, D. Evidencia de anticuerpos contra el virus del síndrome respiratorio y reproductivo porcino en la cría comercial de cerdos en Nigeria: un estudio preliminar. Trop Vet. (2014) 32:97–103. doi: 10.4314/TV. V32I3-4
33. Oba, P, Dione, MM, Erume, J, Wieland, B, Mutisya, C, Ochieng, L, et al. Caracterización molecular del virus del síndrome respiratorio y reproductivo porcino (PRRSv) identificado a partir de cerdos sacrificados en el norte de Uganda. BMC Vet Res. (2022) 18:176–8. doi: 10.1186/s12917-022-03272-x
Resumen de PubMed | Texto completo de Crossref | Google Académico
34. Mutua, F, y Dione, M. El contexto de la aplicación de la bioseguridad para el control de la peste porcina africana en sistemas porcinos de pequeños productores: brechas actuales y recomendaciones. Front Vet Sci. (2021) 8:1–11. doi: 10.3389/fvets.2021.689811
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Franzo, G, De Villiers, L, De Villiers, M, Ravandi, A, Gyani, K, Van Zyl, L, et al. Epidemiología molecular del parvovirus canino en Namibia: vías de introducción y persistencia local. Prev Vet Med. (2022) 209:105780. doi: 10.1016/j.prevetmed.2022.105780
36. Franzo, G, Dundon, WG, De Villiers, M, De Villiers, L, Coetzee, LM, Khaiseb, S, et al. Reconstrucción filodinámica y filogeográfica de la epidemiología del virus de la enfermedad del pico y las plumas y sus implicaciones para el comercio internacional de aves exóticas. Transbound Emerg Dis. (2022) 69:E2677–87. doi: 10.1111/tbed.14618
Resumen de PubMed | Texto completo de Crossref | Google Académico
37. Franzo, G, Settypalli, TBK, Agusi, ER, Meseko, C, Minoungou, G, Ouoba, BL, et al. Circovirus-2 porcino en África: identificación de conglomerados específicos de continentes y evidencia de introducciones virales independientes desde Europa, América del Norte y Asia. Transbound Emerg Dis. (2022) 69:E1142–52. doi: 10.1111/tbed.14400
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Franzo, G, Zerbo, HL, Ouoba, BL, Dji-Tombo, AD, Kindo, MG, Sawadogo, R, et al. Un análisis filogeográfico del parvovirus porcino 1 en África. Virus. (2023) 15:207. doi: 10.3390/V15010207
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: PRRSV, Namibia, África, epidemiología molecular, filogenética, virus
Cita: Molini U, Coetzee LM, Hemberger MY, Chiwome B, Khaiseb S, Dundon WG y Franzo G (2024) Primera detección y caracterización molecular del virus del síndrome respiratorio y reproductivo porcino en Namibia, África. Frente. Vet. Sci. 10:1323974. doi: 10.3389/fvets.2023.1323974
Recibido: 18 de octubre de 2023; Aceptado: 08 de diciembre de 2023;
Publicado: 15 enero 2024.
Editado por:
Mengmeng Zhao, Universidad de Foshan, China
Revisado por:
Alfonso Rosamilia, Instituto Zooprofiláctico Experimental de Lombardía y Emilia Romagna (IZSLER), Italia
Peter Oba, Instituto Internacional de Investigación Pecuaria, Uganda
†DIRECCIÓN ACTUAL:
Lauren M. Coetzee, Facultad de Medicina Veterinaria, Universidad de Teramo, Teramo, Italia
Derechos de autor © 2024 Molini, Coetzee, Hemberger, Chiwome, Khaiseb, Dundon y Franzo. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Umberto Molini, u.molini76@gmail.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía