Primera visión de la caracterización de vesículas extracelulares y miARN en un modelo in vitro de inflamación de ovejas
Primera visión de la caracterización de vesículas extracelulares y miARN en un modelo in vitro de inflamación de ovejas
 Maria Giovanna Ciliberti1*
Maria Giovanna Ciliberti1*  Antonella Santillo1
Antonella Santillo1  Agostino Sevi1
Agostino Sevi1  Marzia Albenzio1
Marzia Albenzio1  Vincenzo De Leo2
Vincenzo De Leo2  Chiara Ingrosso3
Chiara Ingrosso3  Lucía Catucci2
Lucía Catucci2  Mariangela Caroprese1
Mariangela Caroprese1- 1Departamento de Agricultura, Alimentación, Recursos Naturales e Ingeniería (DAFNE), Universidad de Foggia, Foggia, Italia
- 2Departamento de Química, Universidad de Bari Aldo Moro, Bari, Italia
- 3Departamento de Química, Instituto de Procesos Químicos y Físicos del Consejo Nacional de Investigación (CNR-IPCF), Universidad de Bari, Bari, Italia
Las vesículas extracelulares (VE) y sus cargas de microARN (miARN) han atraído la atención en el campo veterinario por su papel regulador en diversos procesos biológicos. Este estudio tuvo como objetivo (i) evaluar dos técnicas de aislamiento de EV a partir de sobrenadantes de células mononucleares de sangre periférica (PBMC) de ovejas utilizando los métodos de ultracentrifugación (CU) y reactivo (REA) y (ii) caracterizar los perfiles de EV-miRNA después de un ambiente inflamatorio in vitro mediado por lipopolisacáridos (LPS). Se recolectó sangre periférica de ovejas y se separaron las PBMC mediante un reactivo de gradiente de densidad. Posteriormente, las PBMC se cultivaron a 37 °C durante 24 h (5% CO2), y se colectaron los sobrenadantes para realizar el aislamiento del VE. Se determinó la presencia del marcador de vesícula extracelular CD81 y se calculó la pureza de los VE aislados como un cociente entre el número de VE aislados y la concentración de proteínas. Además, la caracterización morfológica reveló principalmente estructuras de forma redonda con tamaños promedio de 211 nm para los VE aislados por el método UC y 99 nm para los VE aislados por el método REA. Se utilizó la secuenciación de Illumina NextSeq en modo de un solo extremo para caracterizar el perfil de miARN, y los miARN expresados diferencialmente (DE) se analizaron utilizando una combinación de herramientas bioinformáticas. Los resultados revelaron que el método REA es fiable para el aislamiento de EV de sobrenadantes de ovejas. Se consideró una mejora de la tasa de recuperación y pureza de los EV con el aumento del número y los niveles de expresión de los miRNAs caracterizados. Los VE aislados por el método de UC después de un desafío con LPS mostraron 11 miRNAs de DE, entre los cuales ocho miRNAs estaban regulados al alza y tres a la baja. Por otro lado, el método REA reveló una carga de EV en la que ocho miRNAs de DE estaban regulados al alza y 21 miRNAs de DE estaban regulados a la baja. Los miRNAs maestros reguladores del proceso biológico se identificaron mediante la realización del análisis de la red MIRNA-mRNA, demostrando que, entre los miRNAs de mayor representatividad en función de la centralidad y la intermediación, el miR-26a-5p podría tener un papel crucial en la resolución de la inflamación. Además, la identificación de la familia de miARN let-7 en todos los VE mostró genes potenciales dirigidos que regulan la inflamación y las respuestas inmunitarias.+
1 Introducción
Estudios recientes en ciencias veterinarias han mostrado un creciente interés en las vesículas extracelulares (VE) y sus cargas moleculares relacionadas, en particular los microARN (miARN), que se consideran potenciales biomarcadores de enfermedades y dianas terapéuticas (1-4), dado su papel regulador en diversos procesos biológicos (5). Las VE se definen como nanopartículas liberadas por múltiples tipos de células, y sus vías de biogénesis incluyen el origen del endosoma, con la liberación de exosomas, y el origen derivado de la membrana plasmática, con la liberación de micropartículas/microvesículas (6). Los vehículos eléctricos se clasifican en función de su tamaño como vehículos eléctricos pequeños (<200 nm) o vehículos eléctricos medianos-grandes (>200 nm) (6). En una revisión reciente de Moccia et al. (7), se discute el uso potencial de las VE como marcadores para el diagnóstico de enfermedades o como posibles transportadores naturales de terapias o vacunas en estudios veterinarios. De hecho, los VE están involucrados en gran medida en la comunicación inmunitaria y célula-célula y facilitan la transferencia de ADN, ARNm, microARN y lípidos a las células receptoras cercanas y distantes en relación con la aparición y el desarrollo de muchas enfermedades (8, 9).
Los miRNAs se describen como pequeñas moléculas de ARN no codificante (ncRNA) de aproximadamente 22 nucleótidos de longitud que tienen un papel principal en el proceso postranscripcional como represor de genes o destructor del ARNm objetivo al unirse con el par de bases complementarias en 3′UTR, 5′UTR, o regiones de siembra y codificación de ARNm objetivo en plantas. animales y virus (10, 11). Un gran número de genes codificadores de proteínas están bajo el control del miARN, que está estrechamente regulado (10). En particular, se ha encontrado que las especies de miARN son significativamente más abundantes en los VE que en la célula de origen (12). Durante la invasión de patógenos o bajo lesiones, los miARN pueden desempeñar un papel fundamental en la activación de procesos inmunológicos y fisiológicos que son importantes para la eliminación de patógenos y el mantenimiento de la homeostasis. Curiosamente, los miARN tienen la capacidad de regular la secreción de mediadores inmunitarios por parte de las células inmunitarias junto con la maduración y diferenciación de las células dendríticas, macrófagos, granulocitos y otras células inmunitarias. Además, durante la inflamación de la médula ósea, los miRNAs también desempeñan un papel en los procesos de desarrollo (13). De hecho, durante la inflamación y la activación del sistema inmunitario, una clase de miARN puede funcionar como importantes bucles de retroalimentación negativa en el sistema inmunitario, mientras que otros miARN pueden ser cruciales para la amplificación de la respuesta del sistema inmunitario a través de la represión de los inhibidores de la respuesta (13).
Cabe destacar que la identificación de la técnica óptima para aislar los VE es esencial en la búsqueda de un nuevo biomarcador. Sin embargo, uno de los principales desafíos para avanzar en nuestro conocimiento de las funciones de los VE es la falta de una estrategia de aislamiento eficiente y estandarizada para aislar subpoblaciones específicas y la ausencia de un método estándar de oro para aislar los VE (14). Por lo tanto, la Sociedad Internacional de Vesículas Extracelulares (ISEV, por sus siglas en inglés) propuso en 2014 las directrices de Información Mínima para Estudios de EV (MISEV, por sus siglas en inglés), actualizadas en 14, para mejorar la calidad de la investigación sobre EV, recomendando que no existe un único método de separación óptimo; Sin embargo, la elección depende en última instancia de la aplicación posterior específica y de la cuestión científica. Chen et al. (2018) revisaron las técnicas comunes de separación de EV, demostrando que cada estrategia de separación tiene ventajas y desventajas. Como método más común, la ultracentrifugación es una tecnología madura que se puede utilizar para separar la mayoría de las muestras con bajos gastos operativos. Sin embargo, se considera que requiere mucho tiempo, con una repetibilidad deficiente o inestable, y puede conducir a una posible copurificación con agregados de proteínas, lo que puede afectar los resultados de la espectrometría de masas posterior o la cuantificación de proteínas (15-16). Además, la centrifugación a alta velocidad puede causar daños a los vehículos eléctricos y reducir su actividad biológica (18). Por el contrario, el método de aislamiento basado en kits es un procedimiento «fácil y rápido» que puede sufrir de coaislamiento entre los vehículos eléctricos; sin embargo, puede considerarse una opción ideal para la identificación de biomarcadores de enfermedades relacionadas con exosomas (16).
En el presente estudio, basado en la suposición de que los métodos de aislamiento podrían afectar la carga de EV, se seleccionaron dos métodos para el aislamiento de EV a partir de sobrenadantes de células mononucleares de sangre periférica (PBMC) de ovejas para comparar el perfil miRNomic después de un desafío inflamatorio in vitro mediado por LPS.
Por lo tanto, el presente estudio tuvo como objetivo (i) aislar EVs de sobrenadantes PBMC de ovejas mediante el uso de métodos de ultracentrifugación (UC) y reactivo de aislamiento de exosomas totales (REA) y (ii) caracterizar el perfil miRNomic después de un desafío inflamatorio in vitro, como un modelo de inflamación en ovejas. Hasta donde sabemos, este es el primer estudio que aísla los VE del sobrenadante PBMC de ovejas para analizar el perfil miRNómico. Nuestra hipótesis se centró en una posible conexión entre los miRNAs, la inflamación y las respuestas inmunes en ovejas para definir nuevos biomarcadores predictivos asociados a fenotipos de salud o producción.
2 Materiales y métodos
2.1 Animales y tratamientos experimentales
En este estudio se utilizó la raza ovina lechera Gentile di Puglia criada en el sistema de pastoreo (n = 3). Todos los procedimientos se llevaron a cabo de acuerdo con las directrices de la Directiva de la UE 2010/63/UE (2010) sobre la protección de los animales utilizados para experimentación y otros fines científicos. Los animales fueron examinados cuidadosamente por veterinarios durante todo el ensayo para descartar la presencia de cualquier signo de enfermedad (N. 12,917 del 20220303 2022-UNFGCLE-0012917).
2.2 Aislamiento de PBMC periférica
Las muestras de sangre (15 mL) se recolectaron en tubos de vacío de Na-heparinizado de la vena yugular de las ovejas a través de una aguja de 21G, y las PBMC se aislaron mediante gradiente de densidad Histopaque-1077® (Sigma Aldrich, Milán, Italia) según Wattegedera et al. (20) y se modificaron como se informó previamente por Ciliberti et al. (21). Brevemente, se centrifugó la sangre entera diluida en una proporción de 1:1 con PBS frío, y los anillos de glóbulos blancos recuperados después de la centrifugación se diluyeron en la solución salina equilibrada de Hanks (HBSS, Thermo Fisher Scientific, Waltham, Estados Unidos) y se colocaron lentamente en capas sobre la solución Histopaque-1077® (10 ml, Sigma Aldrich, Milán, Italia). Los tubos se centrifugaron a 400 × g durante 30 min a 20°C, y se recuperó la capa buffy que contenía los PBMC colocados en capas sobre la capa superior de Ficoll-Pacè. La suspensión de PBMC se lavó tres veces con tampón de lavado HBSS que contenía un 2% de suero fetal bovino (FBS) empobrecido en exosomas (Thermo Fisher, Waltham, Estados Unidos) y antibióticos de mezcla de penicilina y estreptomicina (Biowest, Riverside, Estados Unidos). Finalmente, las PBMC se resuspendieron en medio RPMI 1640 sin calcio ni magnesio (Sigma Aldrich, Milán, Italia), que contenía 10% de FBS, antibióticos penicilina/estreptomicina y solución de L-glutamina. Las PBMC se contaron utilizando el método de exclusión de azul de tripano en un contador celular automatizado Countess™ II (Thermo Fisher Scientific, Waltham, Estados Unidos). La viabilidad de las células obtenidas tras el aislamiento de PBMC fue superior al 98%. Una concentración final de 1 × 106 Las células/mL se sembraron en seis placas de pocillos y se cultivaron durante 24 h a 37 °C. Las PBMC se estimularon con lipopolisacáridos (LPS) de Escherichia coli (1 μg/mL) o se dejaron sin estimular (CON).
2.3 Aislamiento de vesículas extracelulares a partir de sobrenadantes de PBMC
Después de la recolección de células libres sobrenadantes de PBMC, el aislamiento de EV se realizó utilizando dos métodos diferentes, como se muestra en el diagrama de flujo del experimento (Figura 1).
 Figura 1. Diagrama de flujo que representa los dos métodos (UC, Ultracentrifugación; REA, Reactivo) utilizado para el aislamiento de EV del sobrenadante de PBMC de ovejas.
Figura 1. Diagrama de flujo que representa los dos métodos (UC, Ultracentrifugación; REA, Reactivo) utilizado para el aislamiento de EV del sobrenadante de PBMC de ovejas.
El método CU se realizó según Baharlooi et al. (22) con algunas modificaciones. Brevemente, los sobrenadantes recolectados se centrifugaron a 300 g durante 5 min para evitar la contaminación celular o celular. Posteriormente, el sobrenadante se filtró con un filtro de 0,22 μm y se centrifugó a 17.000 g durante 30 min para eliminar las microvesículas. A continuación, el sobrenadante recogido se transfirió a un tubo de ultracentrífuga de polipropileno y se sometió a una primera ultracentrifugación a 100.000 × g durante 120 min a 4 °C con una ultracentrífuga Optima LE-80 K (Beckman Coulter, Estados Unidos). Posteriormente, se retiró el sobrenadante y el gránulo EV se suspendió en PBS. Además, la muestra se ultracentrifugó a 100.000 × g durante 60 min. Todos los pasos de centrifugación se realizaron a 4 °C. Finalmente, se desechó el sobrenadante y el gránulo que contenía los VE se suspendió en PBS y se almacenó a -20 °C para su posterior análisis. El método REA se realizó de acuerdo con las instrucciones del fabricante del reactivo de aislamiento total de exosomas (de medios de cultivo celular, Thermo Fisher, Waltham, Estados Unidos), y la relación de aislamiento se basó en unir la molécula de agua y forzar las moléculas menos solubles (EV) fuera de la solución, lo que resultó en una concentración de EV intactos y evitó el procedimiento de ultracentrifugación que requiere mucho tiempo.
El número de VE aislados se determinó mediante la detección de CD81 del ExoELISA-ULTRA Complete Kit (SBI System Bioscience, Embarcadero Way Palo Alto, CA, Estados Unidos), y se generó una curva estándar para determinar el número de VE positivos a tetraspanina CD81 (CD81) siguiendo las recomendaciones de los fabricantes.+
Tanto el método REA como el kit Elisa utilizado para contar el número final de vehículos eléctricos aislados se calibraron en el instrumento NanoSight LM10 según lo informado en las instrucciones del fabricante respectivo. Los EV aislados se analizaron para determinar el contenido de proteína después de la lisis en tampón RIPA siguiendo el procedimiento descrito por Subedi et al. (23). La concentración de proteína se determinó utilizando el kit BCA Protein Assay (Thermo Scientific, Waltham, Estados Unidos) de acuerdo con el manual de instrucciones, calibrado contra la albúmina sérica bovina como estándar.
2.4 Análisis de potencial zeta y microscopía electrónica de transmisión
El potencial zeta de los VE se midió mediante electroforesis láser Doppler (LDE) con un Nanosizer ZS (Malvern Instruments, Malvern, Reino Unido), como informaron previamente De Leo et al. (24).
Los análisis de microscopía electrónica de transmisión se realizaron con un microscopio Jeol Jem-1011 (JEOL USA, Inc., Pleasanton, CA, Estados Unidos) que operaba a 100 kV, equipado con una lente objetivo de alto contraste y un filamento W como fuente de electrones, con una resolución de punto final de 0,34 nm. Las imágenes fueron adquiridas por una cámara Quemesa Olympus CCD de 11 MP. Las muestras se prepararon fundiendo 3 μL de las dispersiones acuosas de los VE aislados en 300 rejillas de Cu amorfo recubiertas de carbono de malla y luego dejando que el disolvente se evaporara a temperatura ambiente. Después de la deposición en la rejilla, las muestras se tiñeron con una solución acuosa al 2% de hidrato de ácido fosfotúngstico. El análisis estadístico del tamaño (tamaño promedio del EV y distribución del tamaño) de cada muestra (CON EVs aislados por los métodos UC y REA, y LPS EVs aislados usando los métodos UC y REA) se realizó en 100 nanoestructuras utilizando un programa gratuito de análisis Image J (National Institutes of Health, Estados Unidos). Los resultados se presentan como media ± desviación estándar.
2.5 Preparación de la biblioteca y secuenciación de nueva generación
Para la preparación de la biblioteca se utilizó el pequeño kit RNA-Seq [Bioo Scientific NextflexTM (v2, v3)] siguiendo las instrucciones del fabricante. A continuación, las bibliotecas se secuenciaron en modo de un solo extremo en el Illumina NextSeq. Antes de un análisis adicional, se realizó una verificación de calidad de los datos de secuenciación sin procesar utilizando la herramienta FastQC disponible en http://www.bioinformatics.babraham.ac.uk/projects/fastqc para datos de secuenciación de alto rendimiento. El recorte de calidad y la retirada del adaptador se realizaron mediante la herramienta bioinformática seleccionada sRNAbench, siguiendo el procedimiento recomendado por Aparicio-Puerta et al. (25).
2.6 Identificación de miRNAs y análisis de expresión diferencial
Las secuencias detectadas se analizaron utilizando sRNAtoolbox (25), para identificar perfiles de expresión de miRNA utilizando un conjunto de datos de especies ovis aries contenido en miRBase (versión 22) (26). Los archivos de expresión generados con sRNAbench enumeraban todas las copias de miARN. Se utilizó el método NOISeq-Sim para determinar los miRNAs expresados de manera diferente de CON frente a LPS de los dos métodos de aislamiento de EV, y la probabilidad de expresarse de manera diferente se calculó y se estableció en >0,85.
2.7 Análisis estadístico y bioinformática
Los datos sobre el marcador EV CD81 y la cuantificación de proteínas se verificaron mediante pruebas de normalidad y se analizaron con un ANOVA de un factor de SAS (27). La significancia de las diferencias se evaluó mediante la prueba post hoc de Tukey para comparaciones múltiples y se consideró estadísticamente significativo un valor de p inferior a 0,05. Los datos se presentaron como media ± SEM.+
Se utilizó R para crear una matriz de todas las transcripciones expresadas en todas las muestras con los correspondientes recuentos de lectura, y se utilizó el paquete de bioconductores NOISeq (28, 29) para normalizar los datos utilizando el método RSEM y luego realizar el análisis de expresión diferencial utilizando NOISeq-Sim (28, 29).
Los datos sobre miRNAs expresados diferencialmente regulados a la baja y regulados al alza se analizaron utilizando una herramienta de análisis de enriquecimiento funcional (FunRich, http://funrich.org/index.html) alimentada con homólogos de miRNAs humanos recuperados a través de miRbase (versión 22.2, https://mirbase.org). La herramienta FunRich realiza análisis estadísticos para diferentes listas de proteínas, análisis de péptidos, agrupamiento o análisis proteómico más complejo (30). El punto de corte de los análisis de enriquecimiento se fijó en <0,05. El análisis de la red de interacción miRNAs-gen diana se realizó y visualizó utilizando la plataforma basada en la web miRNet (versión 2.0, https://www.mirnet.ca/miRNet/home.xhtml), y el análisis de enriquecimiento funcional KEGG se realizó basado en el análisis de la red miRNet de los EVs-miRNAs regulados a la baja y al alza.
3 Resultados
3.1 Cuantificación y caracterización de los VE
Para calcular el número de partículas aisladas se utilizó la presencia de tetraspanina CD81, localizada en la membrana del EV, mostrando una diferencia significativa entre tratamientos (p < 0,001, Figura 2A); de hecho, el aislamiento de EV realizado mediante el método REA resultó en una mayor cantidad de aislamiento de EV, además de la estimulación de PBMC con LPS. Adicionalmente, para calcular la pureza de los VE aislados, se calculó la relación entre el número de VE y la concentración de proteínas, según lo sugerido por Tang et al. (31), y se encontraron diferencias significativas entre las dos técnicas de aislamiento, resultando en una mayor relación en el método REA que en el método UC (p = 0,006, Figura 2B). Mediante el análisis TEM, se identificaron claramente numerosas estructuras de forma redonda en los dos tipos de muestras, aunque se observó una ligera polidispersión en la distribución del tamaño de las partículas. Además, se observó una ligera diferencia en el tamaño medio de las muestras de EV (Figura 3). El análisis estadístico arrojó un valor de diámetro promedio de 211 ± 26 nm para los VE aislados con UC de la muestra CON y de 99 ± 40 nm para los EV aislados con REA de la muestra CON (Figuras 3A, B). La estimulación con LPS no produjo cambios significativos en las dimensiones observadas, y el diámetro medio fue de 260 ± 29 nm para los VE aislados por el método UC y de 67 ± 28 nm para los VE aislados por el método REA (Figuras 3C,D). Los valores de potencial zeta de todas las preparaciones de VE fueron negativos, como se observó previamente para los VE aislados de fluidos biológicos (32). En particular, los valores medidos fueron − 12,3 ± 3,26 y − 27,2 ± 1,9 mV para los EV de muestra de las muestras UC y REA CON, respectivamente. Los valores de potencial zeta permanecieron negativos después de la estimulación con LPS, es decir, −19,3 ± 6,8 mV para los EV de las muestras de UC y 24,7 ± 0,78 mV para los EV de las muestras de REA, lo que sugiere una buena estabilidad coloidal.+
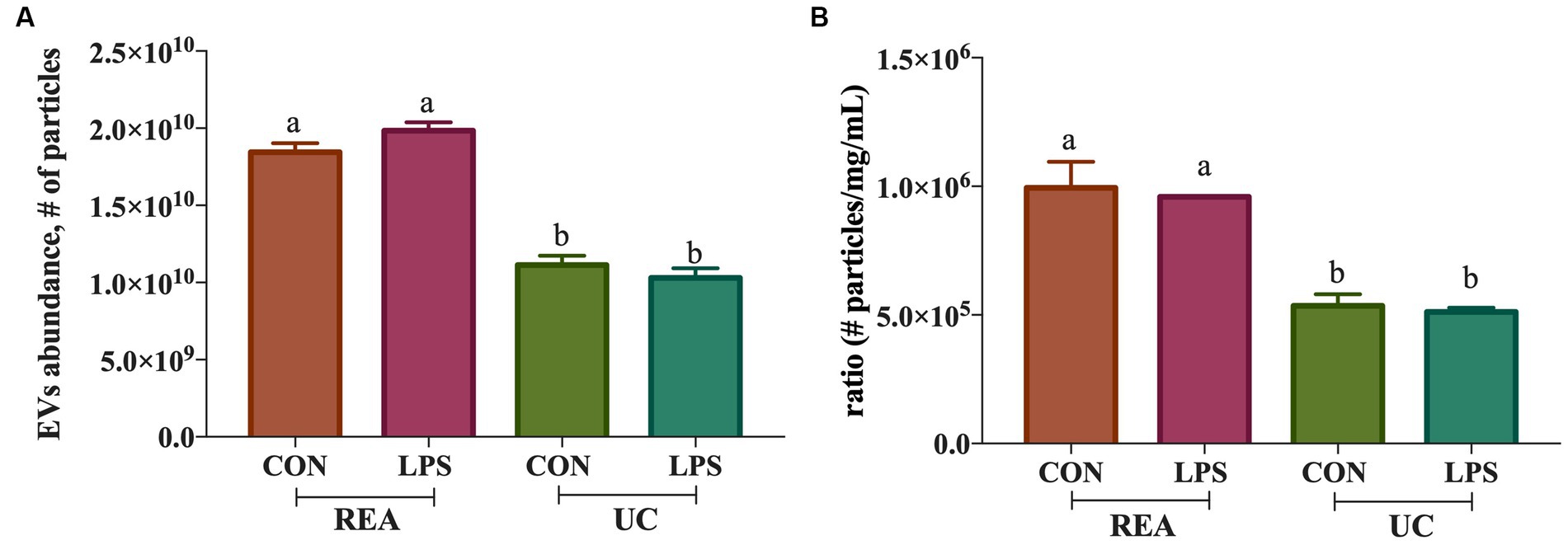 Figura 2. (A) Abundancia de vesículas extracelulares (# de vesículas extracelulares aisladas ± SEM) utilizando los dos métodos (UC, Ultracentrifugación; y REA, Reactivo) en presencia y ausencia de estímulo LPS de inflamación; (B) Relación entre el número de vesículas extracelulares aisladas/concentración de proteínas (mg/mL) de los métodos de ultracentrifugación (CU) y reactivo (REA) en presencia y ausencia de estímulo LPS de inflamación. Los datos se presentaron como medios ± EE.
Figura 2. (A) Abundancia de vesículas extracelulares (# de vesículas extracelulares aisladas ± SEM) utilizando los dos métodos (UC, Ultracentrifugación; y REA, Reactivo) en presencia y ausencia de estímulo LPS de inflamación; (B) Relación entre el número de vesículas extracelulares aisladas/concentración de proteínas (mg/mL) de los métodos de ultracentrifugación (CU) y reactivo (REA) en presencia y ausencia de estímulo LPS de inflamación. Los datos se presentaron como medios ± EE.
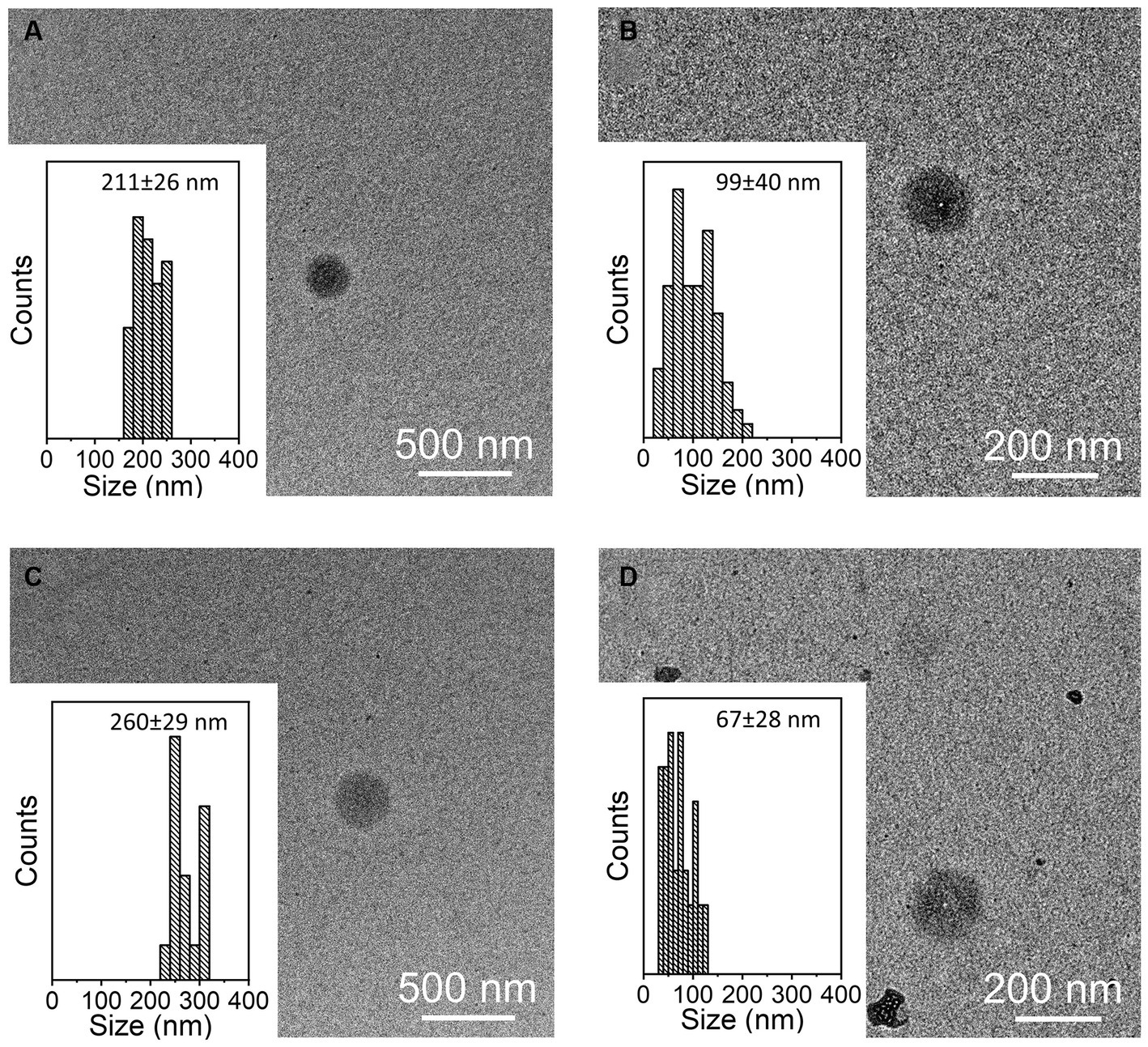 Figura 3. Imágenes TEM representativas de muestras de vesículas extracelulares (VE) y distribución de tamaño medidas en 100 VE para cada muestra. (A) CON EV aislados por el método de ultracentrifugación (UC); (B) CON EV aislados por el método del reactivo (REA); (C) LPS EVs aislados por el método UC; y (D) LPS EVs aislados por el método REA.
Figura 3. Imágenes TEM representativas de muestras de vesículas extracelulares (VE) y distribución de tamaño medidas en 100 VE para cada muestra. (A) CON EV aislados por el método de ultracentrifugación (UC); (B) CON EV aislados por el método del reactivo (REA); (C) LPS EVs aislados por el método UC; y (D) LPS EVs aislados por el método REA.
3.2 MiRNAs expresados diferencialmente entre métodos de aislamiento
Para comparar la superposición de miRNAs entre las cargas de EV-miRNA de las técnicas de aislamiento de CU y REA, independientemente de la presencia de un estimulante, se construyó un diagrama de Venn (Figura 4). Se identificaron un total de cinco miRNAs para CON CU y 12 miRNAs para LPS CU; los miRNAs únicos encontrados en REA CON y REA LPS fueron 42 y 24, respectivamente. El nivel de expresión de cada miARN maduro caracterizado en todas las muestras se informó en la Figura Suplementaria S1.
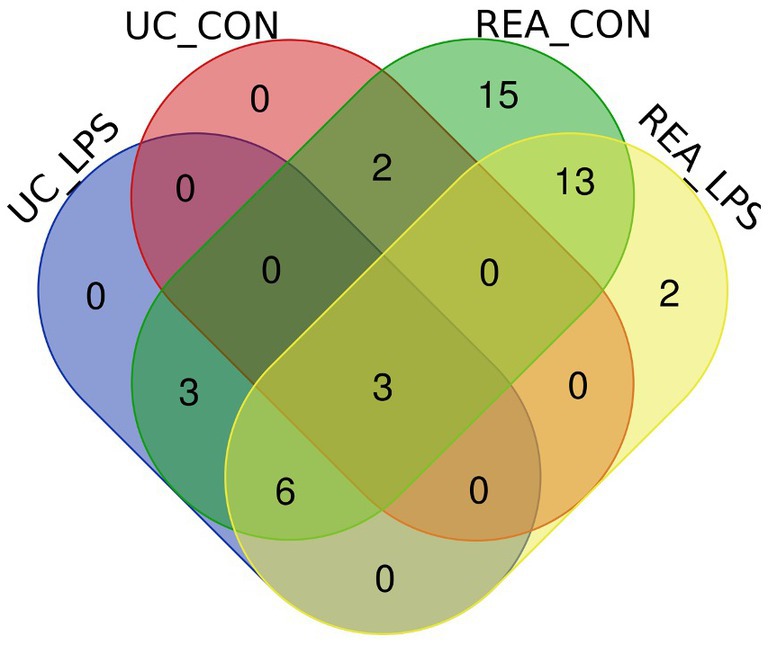 Figura 4. Diagrama de Venn de vesículas extracelulares-miRNAs caracterizadas en sobrenadantes PBMC por métodos de CU y REA y en presencia y ausencia de estímulo LPS de inflamación utilizando la herramienta de Funrich.
Figura 4. Diagrama de Venn de vesículas extracelulares-miRNAs caracterizadas en sobrenadantes PBMC por métodos de CU y REA y en presencia y ausencia de estímulo LPS de inflamación utilizando la herramienta de Funrich.
Los miRNAs significativos expresados diferencialmente (DE) de los EVs de PBMCs aislados por el método UC (CON vs. LPS) mostraron 11 miRNAs de DE en total, entre los cuales ocho miRNAs estaban regulados al alza (oar-miR-26b, oar-miR-23a, oar-let-7i, oar-let-7f, oar-miR-16b, oar-miR-30c, oar-miR-21 y oar-miR-27a), con cambio de pliegue (FC) > 1,5 y probabilidad > 0,85, y tres estaban regulados a la baja (oar-miR-125b, oar-miR-143 y oar-let-7a), con FC < 1,5 y probabilidad > 0,85 (Figura 5A). Por el contrario, el uso del método REA (CON vs. LPS) mostró que entre los miRNAs DE, ocho estaban regulados al alza (oar-miR-369-3p, oar-miR-199a-3p, oar-miR-194, oar-miR-10b, remo-let-7b, remo-let-7d, remo-let-7i y remo-let-7a), con FC > 1.5 y la probabilidad > 0,85 (Figura 5B), y 21 miRNAs se regularon a la baja (oar-miR-27a, oar-miR-10a, oar-miR-152, oar-miR-181a, oar-miR-17-5p, remo-miR-30c, remo-miR-19b, remo-miR-99a, remo-miR-374a, remo-miR-106b, remo-miR-200c, remo-miR-221, remo-miR-25, oar-miR-30d, oar-miR-150, remo-miR-29b, remo-miR-191, remo-miR-26b, remo-miR-29a, remo-miR-23a y remo-miR-148a), con FC < 1,5 y probabilidad > 0,85 (Figura 5B). Entre los miRNAs de DE, los métodos REA y UC tenían en común seis miRNAs entre los cuales oar-miR-26b, oar-miR-30c, oar-miR-27a y oar-miR-23a estaban regulados al alza en la CU y a la baja en la REA, el miARN oar-let-7i estaba regulado al alza en los métodos de UC y REA, y oar-let-7a estaba regulado al alza en el método REA y a la baja en el método UC.
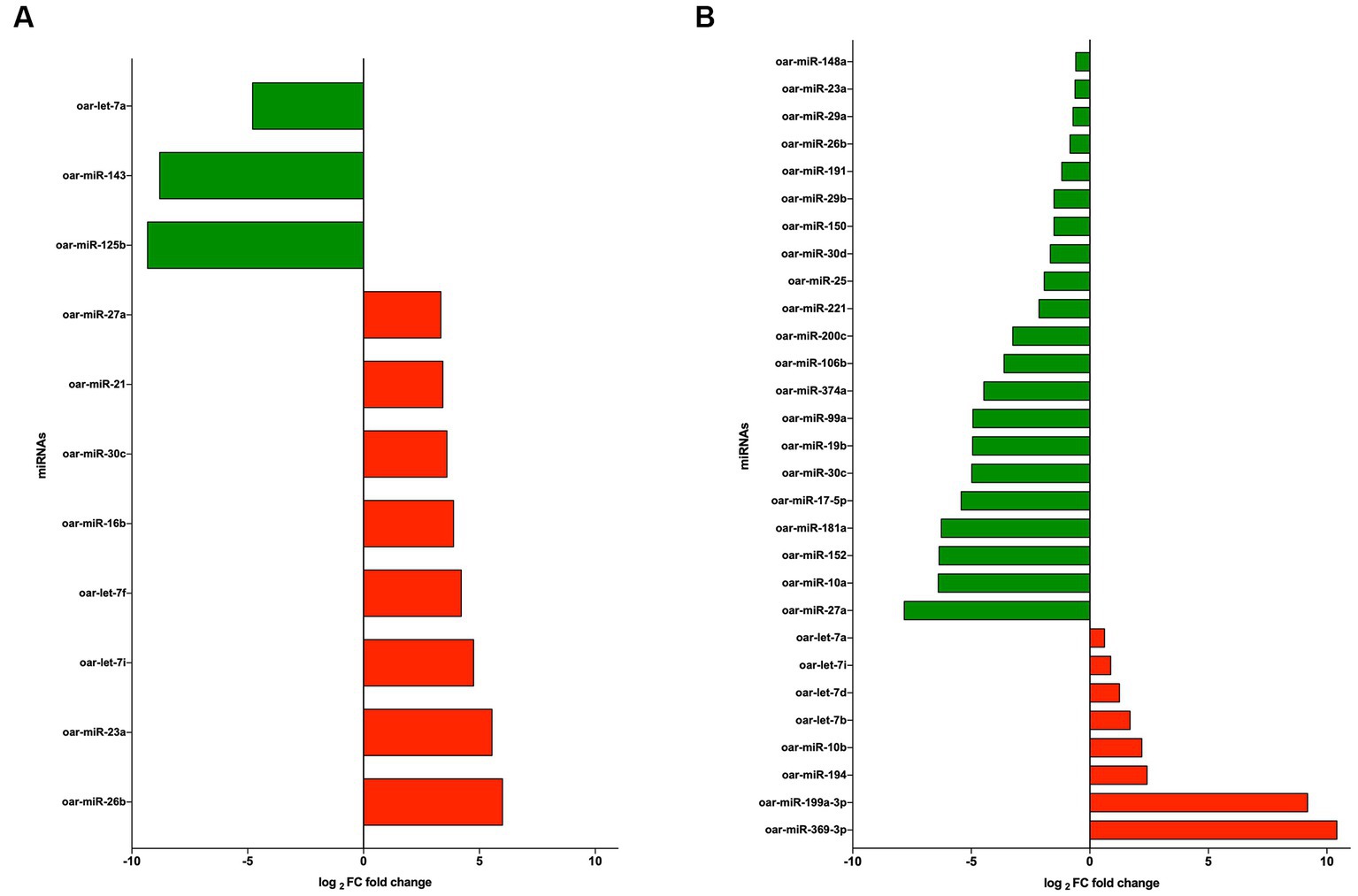 Figura 5. (A) registro2 cambio de pliegue de miRNAs regulados al alza (verde) y regulados a la baja (rojo) de vesículas extracelulares (EVs) de CON vs. LPS aislados por el método de ultracentrifugación (UC). (B) bitácora2 cambio de pliegue de miRNAs regulados al alza (verde) y regulados a la baja (rojo) de EVs CON vs. LPS aislados por el método de reactivo (REA).
Figura 5. (A) registro2 cambio de pliegue de miRNAs regulados al alza (verde) y regulados a la baja (rojo) de vesículas extracelulares (EVs) de CON vs. LPS aislados por el método de ultracentrifugación (UC). (B) bitácora2 cambio de pliegue de miRNAs regulados al alza (verde) y regulados a la baja (rojo) de EVs CON vs. LPS aislados por el método de reactivo (REA).
3.3 Análisis funcional y de enriquecimiento de vías para genes diana de miARN identificados
3.3.1 Método de ultracentrifugación
Para obtener una mejor comprensión de la función y el mecanismo de los miARN, analizamos los datos sobre los miARN de DE regulados a la baja y al alza. Para llevar a cabo este análisis se utilizó la herramienta de enriquecimiento en línea Funrich, y con esta herramienta se analizaron los procesos biológicos (BPs), las vías biológicas (BPath) y las funciones moleculares (MFs). Después del desafío de LPS, los miRNAs regulados a la baja no dieron lugar a ningún gen significativamente enriquecido en el término BP (Figura 6A). Por el contrario, los miRNAs regulados al alza dieron lugar a genes enriquecidos en el término BP relacionados con la regulación del metabolismo de nucleobases, nucleósidos, nucleótidos y ácidos nucleicos (Figura 6B). Con respecto al término BPath, los genes significativos enriquecidos para los miRNAs regulados a la baja (Figura 6C) estuvieron implicados en la vía p53 (7,3%, p = 0,004), los efectores directos de p53 (5,9%, p = 0,007) y la señalización del receptor de estrógeno de la membrana plasmática (28,8%, p = 0,038). Los miRNAs regulados al alza (Figura 6D) dieron como resultado los seis genes principales representativos significativamente enriquecidos relacionados con la señalización del receptor de estrógeno de la membrana plasmática (28,8%, p = 0,005), la red de señalización del receptor ErbB (29%, p = 0,005), los eventos de señalización mediados por VEGFR1 y VEGFR2 (28,8%, p = 0,004), la vía de señalización TRAIL (29,4%, p = 0,004), la vía de la esfingosina 1-fosfato (S1P) (29,2%, p = 0,003), y la red de señalización VEGF y VEGFR (29,2%, p = 0,002).
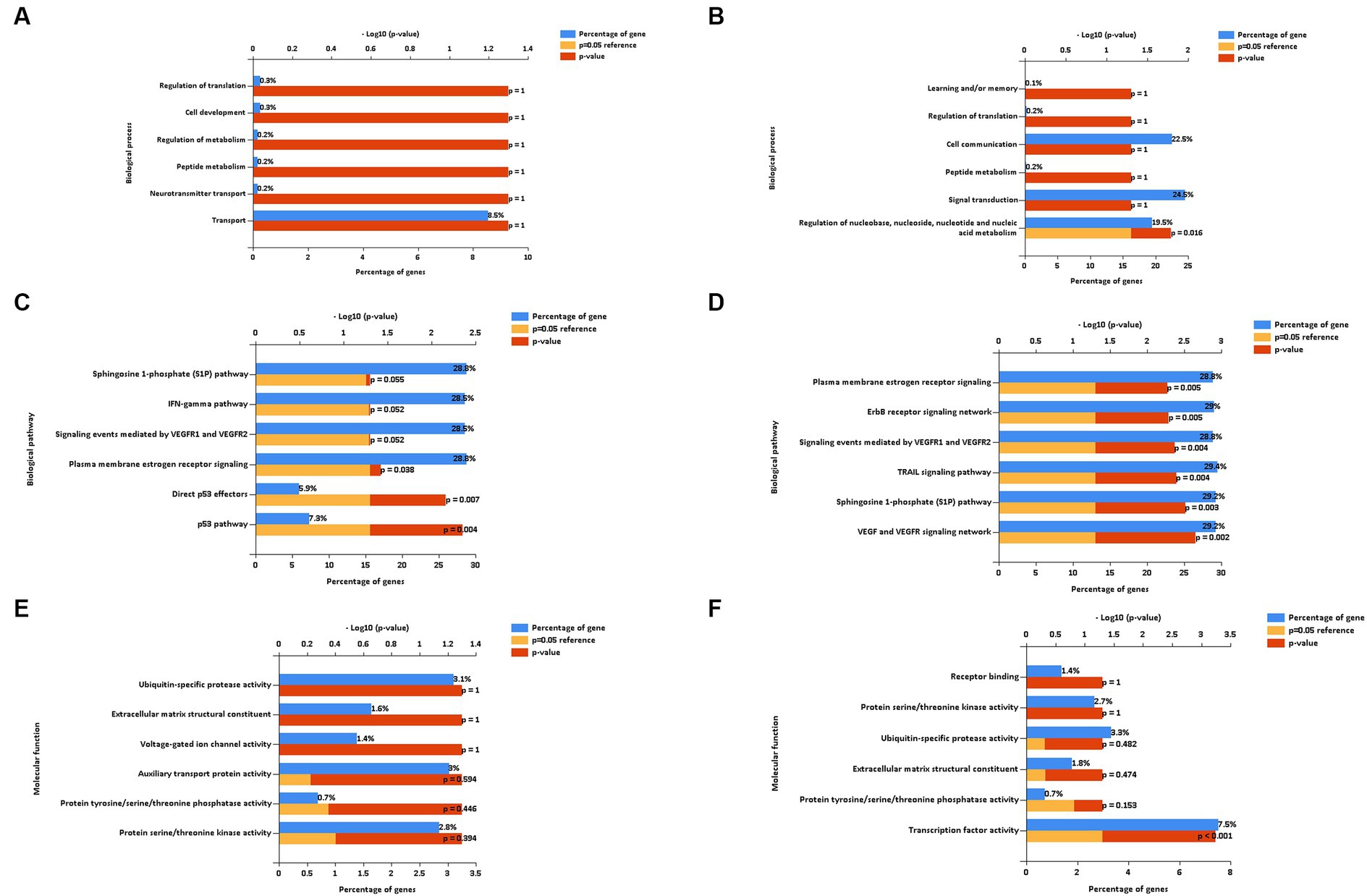 Figura 6. Proceso biológico (A,B), vías biológicas (C,D) y función molecular (E,F) de miRNAs de vesículas extracelulares reguladas hacia abajo y hacia arriba aisladas de sobrenadantes PBMC mediante el método de ultracentrifugación (UC).
Figura 6. Proceso biológico (A,B), vías biológicas (C,D) y función molecular (E,F) de miRNAs de vesículas extracelulares reguladas hacia abajo y hacia arriba aisladas de sobrenadantes PBMC mediante el método de ultracentrifugación (UC).
Con respecto al término MF, los miRNAs regulados a la baja no dieron lugar a ningún gen significativamente enriquecido (Figura 6E), mientras que los miRNAs regulados al alza dieron lugar a los genes enriquecidos implicados en la actividad significativa del factor de transcripción (7,5%, p < 0,001, Figura 6F).
3.3.2 Método del reactivo
Los principales genes enriquecidos implicados en los miRNAs regulados a la baja (Figura 7A) y regulados al alza (Figura 7B) de los términos de BP caracterizados en los EV aislados mediante el método REA (CON vs. LPS) estaban implicados en la comunicación celular y la transducción de señales. Además, los genes enriquecidos de los miRNAs regulados a la baja también estuvieron implicados en la regulación del metabolismo de nucleobases, nucleósidos, nucleótidos y ácidos nucleicos del término BP (20%, p < 0,001).
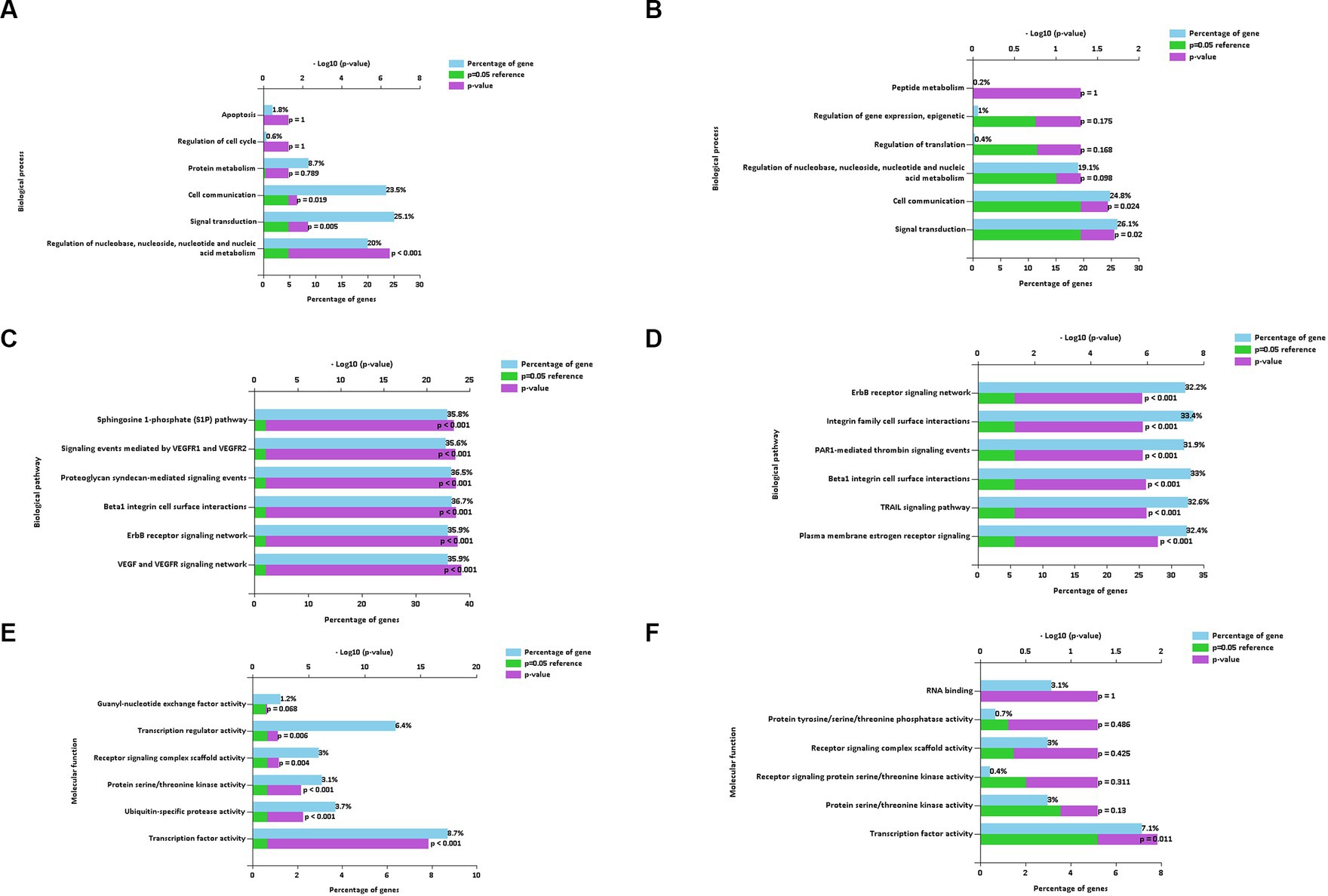 Figura 7. Proceso biológico (A,B), vías biológicas (C,D) y función molecular (E,F) de miRNAs de vesículas extracelulares reguladas hacia abajo y hacia arriba aisladas de sobrenadantes de PBMC mediante el método de reactivos (REA).
Figura 7. Proceso biológico (A,B), vías biológicas (C,D) y función molecular (E,F) de miRNAs de vesículas extracelulares reguladas hacia abajo y hacia arriba aisladas de sobrenadantes de PBMC mediante el método de reactivos (REA).
Los genes enriquecidos de los términos BPath implicados tanto en los miRNAs regulados a la baja (Figura 7C) como en los regulados al alza (Figura 7D) fueron todos significativos (p < 0,001). En particular, los genes enriquecidos con los porcentajes más altos en miRNAs regulados a la baja y regulados al alza estuvieron implicados en las interacciones de la superficie celular de la integrina beta1 del término BPath (33,7 y 33%, respectivamente).
Los genes significativamente enriquecidos del término principal de MF se relacionaron con la actividad del factor de transcripción tanto para los miRNAs regulados a la baja (8,7%, p < 0,001, Figura 7F) como para los regulados al alza (7,1%, p = 0,011, Figura 7E). Además, los genes enriquecidos de los miRNAs regulados a la baja también estuvieron implicados en la actividad reguladora de la transcripción significativa (6,4%, p = 0,006), seguida de la actividad proteasa específica de ubiquitina ( 3,7%, p < 0,001).
3.4 Identificación de miRNAs de vesículas extracelulares a partir de PBMC
3.4.1 Redes de interacción miARN-gen diana y vías KEGG
La red de interacción entre los miRNAs y sus potenciales genes diana es crucial para identificar la relevancia biológica de los miRNAs. En consecuencia, en el método UC, la Figura 8A muestra el ForceAtlas de los miRNAs homólogos humanos EV regulados a la baja en los que has-let-7a-5p tenía una mayor centralidad e intermediación que has-miR-125b-5p y has-miR-143-3p. Entre los EVs-miRNAs regulados al alza de la CU, el homólogo humano has-miR-26a-5p mostró una mayor centralidad e intermediación que has-let-7i-5p (Figura 8B).
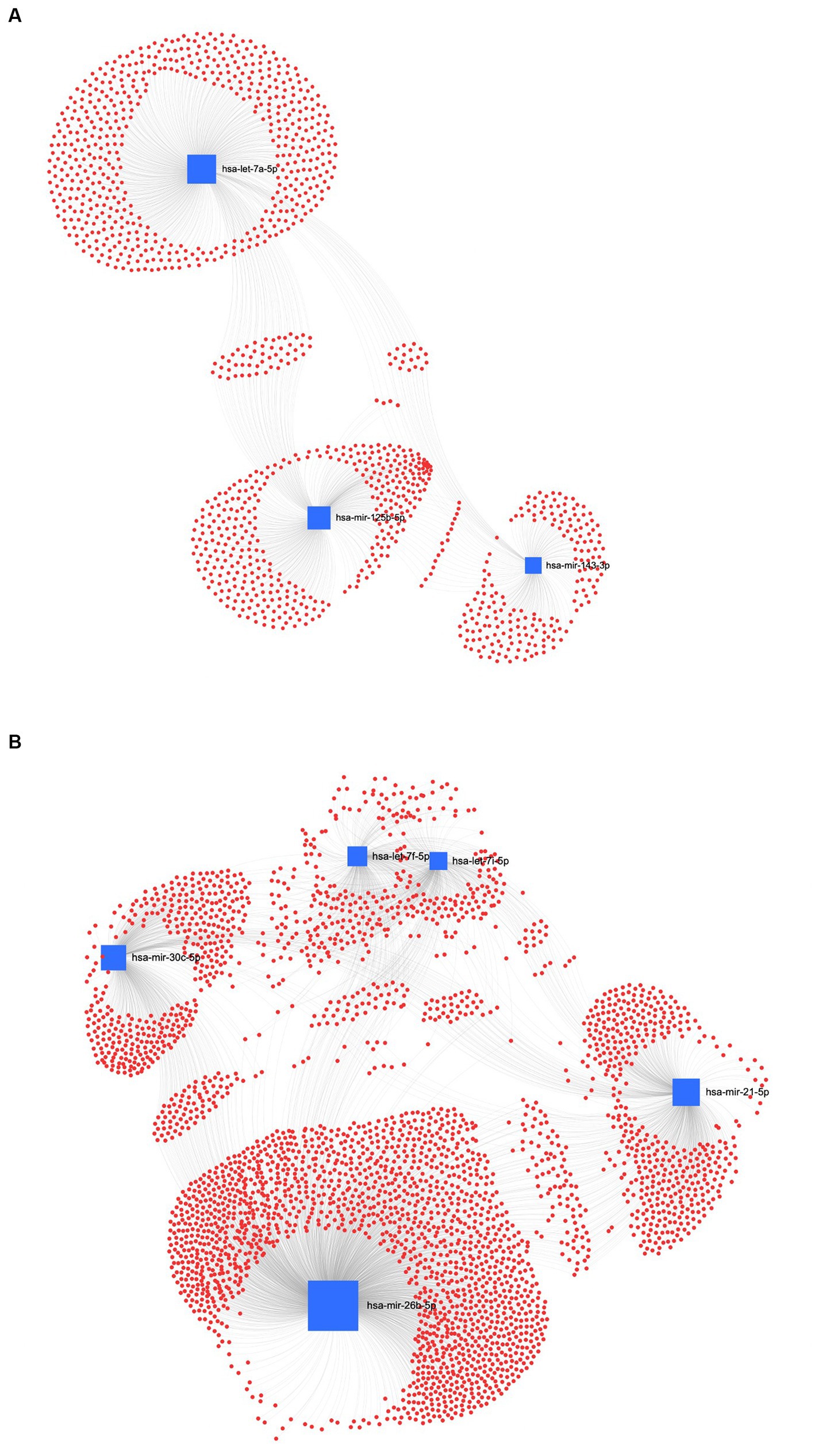 Figura 8. Análisis de la red diana de miARN-gen. La red diana céntrica de genes diana regulados al alza (A) y regulados a la baja (B) en muestras de ultracentrifugación (UC) de VE mediante el software miRNet. Los cuadros representan miRNAs (azul) con el tamaño resultante de la intermediación de los miRNAs en la construcción de la red. Los nodos representan los genes (rojo).
Figura 8. Análisis de la red diana de miARN-gen. La red diana céntrica de genes diana regulados al alza (A) y regulados a la baja (B) en muestras de ultracentrifugación (UC) de VE mediante el software miRNet. Los cuadros representan miRNAs (azul) con el tamaño resultante de la intermediación de los miRNAs en la construcción de la red. Los nodos representan los genes (rojo).
En el método REA, los EVs-miRNAs regulados a la baja de los homólogos humanos, has-miR-26a-5p y has-miR-17-5p, tuvieron la mayor centralidad e intermediación (Figura 9A). Además, entre los miRNAs regulados al alza, has-let-7b-5p, has-let-7a-5p y has-let-7i-5p dieron lugar a una mayor centralidad e intermediación que los otros miRNAs regulados al alza (Figura 9B). Para explorar más a fondo la función biológica de los genes diana predichos, se informó del análisis de la vía KEGG de los miARN significativamente regulados a la baja y al alza en muestras de UC y REA (Figuras suplementarias S2, S3).
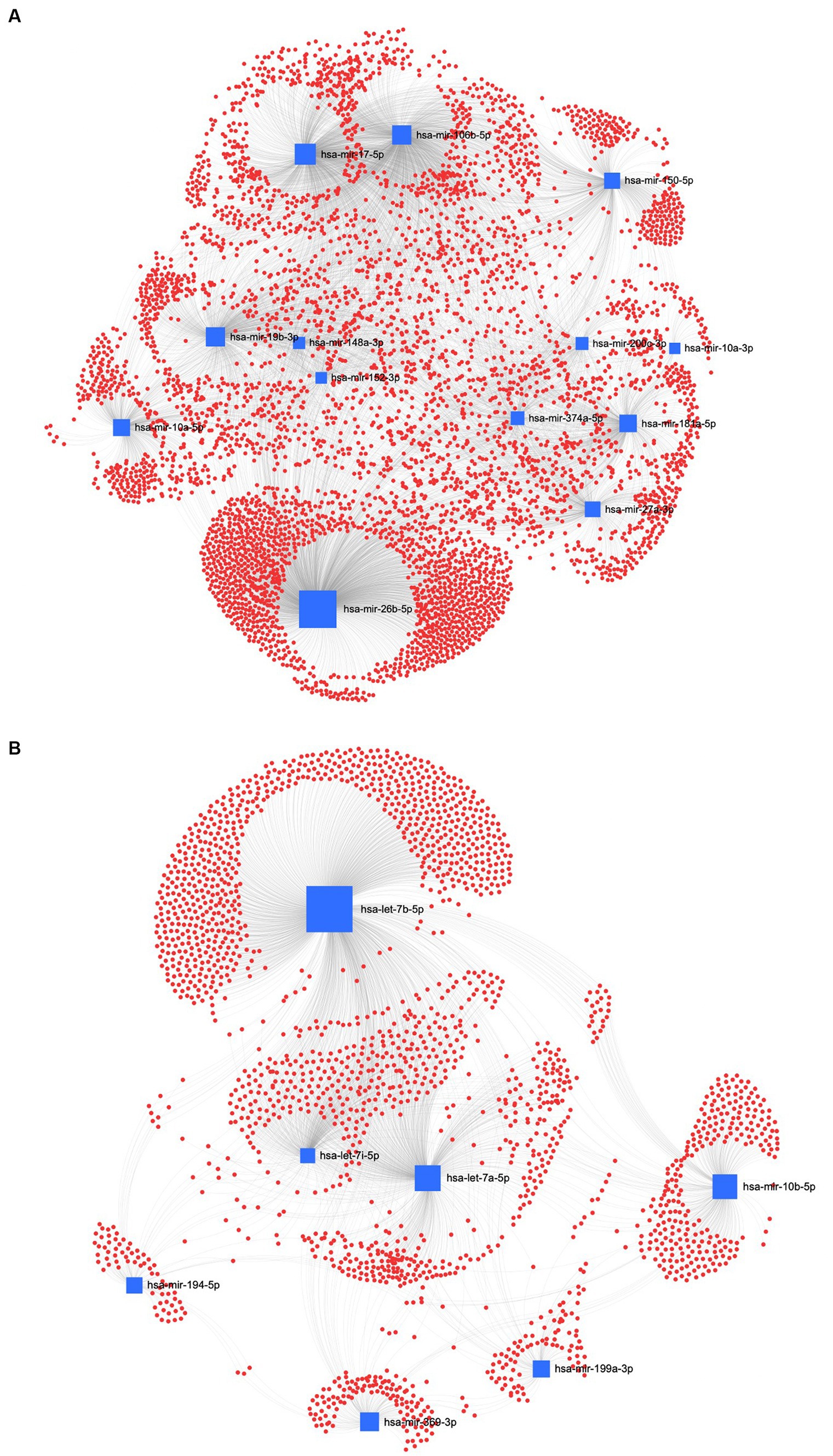 Figura 9. Análisis de la red diana de miARN-gen. La red diana céntrica de genes diana regulados al alza (A) y regulados a la baja (B) en muestras de reactivos (REA) de VE mediante el software miRNet. Las cajas representan los miRNAs (azul) con el tamaño resultante de la intermediación de los miRNAs en la construcción de la red. Los nodos representan los genes (rojo).
Figura 9. Análisis de la red diana de miARN-gen. La red diana céntrica de genes diana regulados al alza (A) y regulados a la baja (B) en muestras de reactivos (REA) de VE mediante el software miRNet. Las cajas representan los miRNAs (azul) con el tamaño resultante de la intermediación de los miRNAs en la construcción de la red. Los nodos representan los genes (rojo).
4 Discusión
En los últimos años, se ha explorado el papel putativo de los miRNAs como biomarcadores en enfermedades de los animales de granja y como enfoque terapéutico basado en su papel clave regulador en la enfermedad (33). Hasta donde sabemos, este es el primer estudio en el que se ha investigado la caracterización del perfil de miRNA de VE aislados de sobrenadantes de PBMC de ovejas, desafiados in vitro con LPS. Uno de los retos científicos más importantes hasta la fecha es el aislamiento y la caracterización de los vehículos eléctricos. Comprender la importancia de los EVs-miRNAs es un aspecto crucial de esta investigación (14, 34). Por lo tanto, la identificación de la técnica óptima para aislar los VE es crucial cuando el objetivo principal es la caracterización de los miRNAs para futuros descubrimientos de biomarcadores. En particular, en el presente estudio, nos centramos en dos métodos de aislamiento de EV a partir de sobrenadantes de cultivos celulares. El aislamiento de EV mediante el método UC es una técnica tradicional, pero requiere un entrenamiento previo y puede ser bastante tedioso y llevar mucho tiempo. Además, los resultados pueden ser sensibles a la técnica utilizada (35). Nuestros resultados demostraron que el método UC disminuye la tasa de recuperación de los EV CD81, como también lo confirma la baja pureza calculada como la relación entre el número de partículas y la concentración de proteínas. De acuerdo con los estudios realizados por Helwa et al. (35) y Lane et al. (36), el método de aislamiento de CU resultó en una menor recuperación de VE en comparación con los kits disponibles comercialmente. Este resultado puede deberse a las diferentes fuerzas de centrifugación a lo largo de múltiples ciclos, útiles para eliminar los restos celulares y otros contaminantes, lo que también puede causar una pérdida de EV de la muestra, lo que conduce a un rendimiento de EV más bajo y variable (37). Según el estudio realizado por Tang et al. (31), el número de VE aislados mediante el método UC fue menor en comparación con el obtenido utilizando dos kits comerciales diseñados específicamente para el aislamiento de EV (ExoQuick y Total Exosomes Isolation Reagent_TEI). Sin embargo, incluso si el método de CU resultó en un menor número de EV, se obtuvieron marcadores de EV más enriquecidos mediante el uso de Western blot (31). En contraste con estos hallazgos, el uso de una centrífuga a altas velocidades puede afectar las propiedades físicas de los VE y la sensibilidad del análisis proteómico (38, 39). En consecuencia, en el presente estudio, la caracterización de EV-miRNA reveló que el método UC reduce las cargas de EV-miRNA, en términos del número de miRNAs identificados, como se muestra en el diagrama de Venn. Este resultado es consistente con la menor tasa de recuperación de EV y pureza debido al método de aislamiento UC. Hasta la fecha, este estudio es el primero sobre la caracterización de EV a partir de sobrenadantes de PBMC de ovejas; por lo tanto, será un paso crucial confirmar nuestros datos con una caracterización adicional de la proteína EV basada en los métodos informados en las MISEV2018 directrices.+
Se reconoce que los miRNAs desempeñan un papel clave en la regulación de procesos genéticos y biológicos como la proliferación celular, la apoptosis, la diferenciación y la tumorigénesis (40, 41). Los miARN maduros regulan la expresión del gen diana reconociendo y complementando completa o incompletamente las secuencias de ARNm diana a través de 5-8 nucleótidos en su extremo 5′ y luego induciendo la degradación o inhibición traduccional del ARNm diana; por lo tanto, los miRNAs maduros regulan la expresión del gen diana (42).
En estudios veterinarios, se ha demostrado que los miARN median la inmunidad celular, la apoptosis, la transducción de señales y la diferenciación celular (43, 44). En particular, el inicio de estímulos inflamatorios desencadena tanto miARN como genes codificadores de proteínas. Por un lado, la expresión de muchos miRNAs está regulada por silenciadores, potenciadores y modificaciones epigenéticas como la hipometilación e hipermetilación en los factores promotores y de transcripción de miRNAs (45, 46). De estos factores de transcripción, el factor nuclear-κB (NF-κB) es el que regula la expresión de varios miRNAs asociados a la inflamación (miR-146a y miR-155) (47-49) y funciona como una fuerte señal proinflamatoria (47), induciendo la expresión de genes proinflamatorios, incluyendo citoquinas y quimiocinas (50). En las vacas lecheras, los miRNAs pueden ejercer una regulación postranscripcional de las reacciones endocrinas, metabólicas e inmunitarias activadas por el desequilibrio energético que se produce durante la transición a la lactancia (51, 52). En particular, en los perfiles de miARN circulantes de vacas lecheras en transición, se ha identificado una selección de subconjuntos de miARN como biomarcadores putativos de trastornos metabólicos, siendo estos biomarcadores las principales vías afectadas relacionadas con la adaptación metabólica e inmunitaria (52). En el presente estudio, los EVs-miRNAs, caracterizados utilizando ambos métodos y analizados considerando el desafío in vitro de LPS, mostraron un subconjunto diferente a los miRNAs regulados al alza y a la baja. En particular, los métodos REA y UC tenían en común, entre los DEM, el miR-26b, que estaba regulado al alza en el método UC y a la baja en el método REA. Se ha reconocido que la familia miR-26 se dirige a los reguladores que controlan el desarrollo (53), el tipo de célula y la diferenciación de los tumores (54). Zhu et al. (55) informaron que, tanto en condiciones fisiológicas como patológicas, miR-26b se expresa concomitantemente con los genes de su huésped, cooperando para bloquear la transición de la fase G1/S de la proliferación a través de la activación de la proteína pRb (55). La sobreexpresión de miR-26b se relacionó directamente con la composición de los ácidos grasos de la leche en un estudio in vitro en células epiteliales mamarias de cabras (56). Además, el papel de miR-26b se asoció con numerosas enfermedades metabólicas, desempeñando un papel crucial en el desarrollo de los adipocitos (56) y la lipogénesis (57). Se descubrió que el bta-miR-26b sintético regula a la baja tres genes relacionados con el sistema inmunitario en las células epiteliales endometriales (EEC) primarias bovinas; por lo tanto, bta-miR-26b ha sido un miRNA candidato entre los miRNAs potenciales que regulan el sistema inmune durante los períodos periimplantarios (58).
El efecto de miR-17-5p sobre la inflamación mediada por LPS se probó en un estudio in vitro en células de RPMI2650 realizado por Huang et al. (59), quienes encontraron que la sobreexpresión de miR-17-5p intensifica la inflamación a través de la regulación negativa de la expresión de la proteína Smad7, que ejerce un papel protector por la inactivación de las vías NF-κB y Wnt/β-catenina. Además, miR-17-5p actúa como un regulador clave de los reguladores negativos de desacoplamiento del ciclo celular G1/S de la cascada de señalización MAPK (60). En el presente estudio, el análisis de KEGG reveló las vías más enriquecidas de los genes diana de los miRNAs regulados a la baja y al alza, la implicación de la vía de señalización MAPK. La familia MAPK tiene un papel central en muchos procesos fisiológicos como la apoptosis y la inflamación, con regulación directa o indirecta de la actividad NF-κB, que es el factor de transcripción clave para promover la expresión de citoquinas inflamatorias (61). El reclutamiento de las vías MAPK se activa por patrones moleculares asociados a patógenos (PAMPs), porciones moleculares presentes en patógenos invasores, y por patrones moleculares asociados al daño (DAMPs), moléculas producidas endógenamente en momentos de estrés fisiológico, que apoyan una respuesta inflamatoria robusta que incluye la producción de citoquinas y la activación de leucocitos (62). En el presente estudio, la implicación de la vía MAPK de los genes diana de los miRNAs regulados al alza podría establecer la relación entre la información postranscripcional impulsada por los miRNAs EV y la respuesta a la inflamación mediada por LPS. Estudios previos afirmaron que la lesión celular inducida por LPS puede estar mediada por la acción moduladora de los miRNAs (63). Además, los resultados del presente estudio, a partir de la red de interacción entre los miRNAs y sus potenciales genes diana, sugieren que tanto el miR-26b como el miR-17-5p podrían considerarse entre los reguladores maestros de los procesos biológicos activados por el LPS. No se han realizado estudios previos sobre la caracterización del miARN sobrenadante de PBMC en ovejas; sin embargo, se podría plantear la hipótesis de que miR-26b y miR-17-5p pueden tener un papel crucial en el proceso de inflamación mediado por LPS en las células sanguíneas y en su resolución, aunque se necesitan más estudios para dilucidar mejor esta hipótesis.
A partir del análisis de datos de la red de interacción entre los miRNAs y sus potenciales genes diana, se encontró que existe un alto número de miRNAs de la familia let-7 presentes, entre los cuales oar-let-7a, remo-let-7b, remo-let-7d y oar-let-7i emergieron como los más destacados. En el estudio de Izumi et al. (64), se encontró que el miRNA let-7a se expresa en gran medida en los exosomas de la leche cruda bovina (MEV) y también está presente en los exosomas de la leche humana y porcina (65, 66). Los miRNAs altamente expresados de los exosomas de leche de varias especies exhibieron funciones comunes, como la regulación de la función inmune y la maduración intestinal (64). De hecho, se demostró el papel de los microARN en el desarrollo, la salud y la función de las glándulas mamarias del ganado vacuno, caprino y ovino (67). Además, los enfoques metabolómicos y transcriptómicos aplicados a los MEV de vacas, burros y cabras revelaron que numerosas vías metabólicas implicadas en la inmunomodulación (68) y las dianas de miARN con términos enriquecidos relacionados con la modulación de la inmunidad, la síntesis de proteínas y la regulación del ciclo celular eran comunes en las especies de vacas, burros y cabras (69).
Los miRNAs let-7f y let-7b están altamente expresados tanto en la leche de vaca como en la de oveja con funciones inmunológicas establecidas (70). El let-7b se dirige a los receptores tipo toll (TLR), que regulan la activación de NF-κB, y sus genes posteriores se relacionaron con la inflamación y las respuestas inmunitarias durante una infección in vitro por Helicobacter pylori de células epiteliales gástricas humanas (71). El perfil de miRNA de las ovejas que respondieron a la infección de lentivirus de pequeños rumiantes mostró que DE miR-21, miR-148a y let-7f regulaban las ovejas seronegativas e infectadas, lo que demuestra sus posibles implicaciones para la interacción huésped-virus (72). Además, el miARN let-7a se dirige al gen IL-6, lo que resulta en una regulación negativa de los tumores relacionados con un modelo celular in vitro de inflamación (73, 74). En el presente estudio, la familia de miRNAs let-7 fue implicada bajo el desafío inflamatorio LPS, mostrando miRNAs regulados tanto al alza como a la baja, demostrando así su función biológica reguladora durante la inflamación en PBMC de ovejas. Además, una implicación prometedora del gen IL-6 puede revelar una posible relación cruzada entre las vías inflamatorias y los miARN, como ha revisado recientemente Chatterjee et al. (75), que debe explorarse más a fondo. De hecho, la IL-6 es bien conocida como un biomarcador potencial de inflamación tanto en estudios en humanos como en rumiantes (21, 76, 77).
Del análisis de KEGG surgió la implicación de la vía de señalización p53 a partir de la diana génica de todos los miRNAs de DE caracterizados. Se ha sugerido que el equilibrio adecuado entre las vías de señalización p53 y MAPK es necesario después de aplicar un estímulo estresante a las células, ya que esto determinará si las células sobreviven o mueren. La participación de la vía de señalización p53 en este equilibrio se ha propuesto como un vínculo potencial entre las dos vías (78). De hecho, la activación de p53 provoca la detención del crecimiento y la apoptosis a través de la supresión de una serie de genes que pueden favorecer la supervivencia celular (78). MiR-143 se clasifica como un supresor tumoral al detener las células de la fase G0/G1 y promover la apoptosis de la caspasa-3 (79). Además, un mecanismo específico de apoptosis activado por miR-143 está implicado en la diana de MDM2 e, indirectamente, en la activación de la vía p53, que a su vez activa la transcripción de miR-143, generando un bucle de retroalimentación corto miR-143-MDM2-p53 (18, 80, 81). La evidencia previa podría explicar la regulación a la baja caracterizada de oar-miR-143 en la muestra de CU y el aumento concomitante de la vía biológica p-53.
Además, el análisis de KEGG mostró que las vías más enriquecidas de los genes diana de los miRNAs de DE regulados al alza y a la baja de los métodos UC y REA después de la estimulación de LPS se asociaron con la vía de señalización JAK/STAT. En el estudio de Yang et al. (82), se encontró que miR-203a-3p induce la polarización del macrófago-M2 mediante la regulación negativa del supresor de la expresión de la señalización de citoquinas 3 (SOCS3) y la activación de la señalización JAK/STAT3 (82). Es bien sabido que SOCS es una familia de proteínas de ocho miembros (SOCS1-7 y CIS) que inhiben la activación de STAT actuando sobre los receptores activadores de JAK/STAT. Un estudio anterior destacó que los miRNAs están involucrados en la regulación de las respuestas inmunes e inflamatorias innatas (83). En estudios con rumiantes, el nexo entre los miRNAs circulantes y los perfiles de citoquinas podría considerarse un factor clave en el estudio de su susceptibilidad a la enfermedad. En un estudio realizado por Naylor et al. (84) en corderos desafiados con LPS a nivel sistemático, se observó que había un aumento simultáneo de los biomarcadores inmunológicos y de estrés. Algunos de los biomarcadores que mostraron un aumento fueron la IL-6, el cortisol y ciertos miARN, incluidos miR-145, miR-1246 y miR-223. Además, se demostró que, tras la inflamación mediada por LPS en una línea celular dendrítica murina derivada de la médula ósea, miR-369-3p puede suprimir la inflamación reduciendo la secreción de moléculas proinflamatorias (TNFα, IL-6, IL-12, IL-1α e IL-1β) al tiempo que aumenta los niveles de IL-10 e IL-1RA, con funciones antiinflamatorias (85). En el presente estudio, miR-369-3p fue regulado al alza en REA probablemente como resultado de su acción supresora en la inflamación mediada por LPS. En un estudio realizado por Gholami et al. (86), algunos miRNAs estuvieron implicados en el inicio de la respuesta inflamatoria mediante la modulación de muchas respuestas inmunitarias, incluyendo la secreción de citoquinas y quimiocinas como miR-26a. En consecuencia, para seleccionar posibles miRNAs como nuevos biomarcadores en ovejas, será fundamental comprender las funciones específicas y los mecanismos de interacción entre los miRNAs y otras biomoléculas implicadas en la patogénesis de la enfermedad, que incluyen citoquinas proinflamatorias y proteínas plasmáticas (87).
5 Conclusión
En el presente estudio, se utilizaron dos métodos de aislamiento diferentes para comparar el EV-miRNome de PBMC sobrenadantes de ovejas. Los datos demostraron que el método REA es fiable para el aislamiento de VE, mejorando la tasa de recuperación y la pureza de los VE CD81, y mejorando el número de miARN caracterizados. Sin embargo, dado que este estudio es el primero sobre la caracterización del EV sobrenadante de ovejas, en el futuro se realizarán métodos adicionales para una caracterización más profunda de las proteínas, tal y como se informa en las directrices de MISEV2018.+
Entre los miRNAs de DE, los EVs de PBMC aislados mediante el método UC mostraron ocho miRNAs regulados al alza y tres miRNAs regulados a la baja, mientras que el método de aislamiento de REA EV se caracterizó por ocho miRNAs regulados al alza y 21 miRNAs regulados a la baja. Además, los métodos de aislamiento probados de los miRNAs de DE tenían en común la presencia de miR-26b y miR-17-5p, que podrían desempeñar un papel crucial en la resolución de la inflamación mediada por LPS, así como la familia de miRNAs let-7 caracterizada en todos los genes diana de EV que regulan la inflamación y las respuestas inmunitarias.
Los datos del presente estudio pueden proporcionar información sobre el papel putativo de los EV-miRNAs en un modelo de inflamación de ovejas mediado por un desafío de LPS.
Declaración de disponibilidad de datos
Los conjuntos de datos generados para este estudio están disponibles previa solicitud al autor correspondiente.
Declaración ética
El estudio en animales fue aprobado por el Comité de Ética Científica de la Universidad de Foggia. El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
MCi, MCa y AgS contribuyeron a la concepción y diseño del estudio. MCi supervisó el proyecto en general y realizó el análisis estadístico. MCi, VL, CI y LC realizaron el análisis. MCi y AnS organizaron la base de datos. MCi, MA, AnS y MCa escribieron el primer borrador del manuscrito. VL, CI y LC contribuyeron a la revisión del manuscrito. Todos los autores contribuyeron al artículo y aprobaron la versión enviada.
Reconocimientos
Los autores agradecen a la región de Apulia por el apoyo en el proyecto «Cambiamenti climatici e indagine predittiva di malattie degli animali da reddito mediante micrornas (microcippare)» (código de proyecto UNIFG288-CUP D74I19003340002), como parte de la acción regional «Investigación para la innovación» en cooperación con la estrategia europea 2020. VL es investigadora en la Universidad de Bari dentro del programa de la Unión Europea «FSE-REACT-EU, PON Research and Innovation 2014-2020», DM1062/2021.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1186989/full#supplementary-material
Referencias
1. Ailawadi, S, Wang, X, Gu, H, and Fan, GC. Pathologic function and therapeutic potential of exosomes in cardiovascular disease. Biochim Biophys Acta Bioenerg. (2015) 1852:1–11. doi: 10.1016/j.bbadis.2014.10.008
2. Ojo, OE, and Kreuzer-Redmer, S. MicroRNAs in ruminants and their potential role in nutrition and physiology. Vet Sci. (2023) 10:57. doi: 10.3390/vetsci10010057
3. Yáñez-Mó, M, Siljander, PR, Andreu, Z, Bedina Zavec, A, Borràs, FE, Buzas, EI, et al. Biological properties of extracellular vesicles and their physiological functions. J Extracell Vesicles. (2015) 4:27066. doi: 10.3402/jev.v4.27066
4. Zhao, L, Liu, W, Xiao, J, and Cao, B. The role of exosomes and «exosomal shuttle microRNA» in tumorigenesis and drug resistance. Cancer Lett. (2015) 356:339–46. doi: 10.1016/j.canlet.2014.10.027
5. Colitti, M, Sgorlon, S, and Stefanon, B. Exosome cargo in milk as a potential marker of cow health. J Dairy Res. (2020) 87:79–83. doi: 10.1017/S0022029920000485
6. Théry, C, Witwer, KW, Aikawa, E, Alcaraz, MJ, Anderson, JD, Andriantsitohaina, R, et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. J Extracell Vesicles. (2018) 7:1535750. doi: 10.1080/20013078.2018.1535750
7. Moccia, V, Sammarco, A, Cavicchioli, L, Castagnaro, M, Bongiovanni, L, and Zappulli, V. Extracellular vesicles in veterinary medicine. Animals. (2022) 12:2716. doi: 10.3390/ani12192716
8. Théry, C, Zitvogel, L, and Amigorena, S. Exosomes: composition, biogenesis and function. Nat Rev Immunol. (2002) 2:569–79. doi: 10.1038/nri855
9. Zamboni, C, Zamarian, V, Stefanello, D, Ferrari, R, Auletta, L, Milanesi, S, et al. Plasma small extracellular vesicles from dogs affected by cutaneous mast cell tumors deliver high levels of miR-21-5p. Front Vet Sci. (2023) 9:1083174. doi: 10.3389/fvets.2022.1083174
10. Almeida, MI, Reis, RM, and Calin, GA. MicroRNA history: discovery, recent applications, and next frontiers. Mutat Res Fund Mol Mechanism. (2011) 717:1–8. doi: 10.1016/j.mrfmmm.2011.03.009
11. Bartel, DP. MicroRNAs: genomics, biogenesis, mechanism, and function. Cells. (2004) 16:281–97. doi: 10.1016/S0092-8674(04)00045-5
12. Van Balkom, BWM, Eisele, AS, Pegtel, DM, Bervoets, S, and Verhaar, MC. Quantitative and qualitative analysis of small RNAs in human endothelial cells and exosomes provides insights into localized RNA processing, degradation and sorting. J Extracell Vesicles. (2015) 4:26760. doi: 10.3402/jev.v4.26760
13. Contreras, J, and Rao, DS. MicroRNAs in inflammation and immune responses. Leukemia. (2012) 26:404–13. doi: 10.1038/leu.2011.356
14. Lötvall, J, Hill, AF, Hochberg, F, Buzás, EI, di Vizio, D, Gardiner, C, et al. Minimal experimental requirements for definition of extracellular vesicles and their functions: a position statement from the International Society for Extracellular Vesicles. J Extracell Vesicles. (2014) 3:26913. doi: 10.3402/jev.v3.26913
15. Chen, J, Li, P, Zhang, T, Xu, Z, Huang, X, Wang, R, et al. Review on strategies and technologies for exosome isolation and purification. Front Bioeng Biotechnol. (2022) 9:811971. doi: 10.3389/fbioe.2021.811971
16. Jeppesen, DK, Hvam, ML, Primdahl-Bengtson, B, Boysen, AT, Whitehead, B, Dyrskjøt, L, et al. Comparative analysis of discrete exosome fractions obtained by differential centrifugation. J Extracell Vesicles. (2014) 3:25011. doi: 10.3402/jev.v3.25011
17. Tauro, BJ, Greening, DW, Mathias, RA, Ji, H, Mathivanan, S, Scott, AM, et al. Comparison of ultracentrifugation, density gradient separation, and immunoaffinity capture methods for isolating human colon cancer cell line LIM1863-derived exosomes. Methods. (2012) 56:293–304. doi: 10.1016/j.ymeth.2012.01.002
18. Zhang, M, Jin, K, Gao, L, Zhang, Z, Li, F, Zhou, F, et al. Methods and technologies for exosome isolation and characterization. Small Methods. (2018) 2:1800021. doi: 10.1002/smtd.201800021
19. Kalra, H, Drummen, GP, and Mathivanan, S. Focus on extracellular vesicles: introducing the next small big thing. Int J Mol Sci. (2016) 17:170. doi: 10.3390/ijms17020170
20. Wattegedera, S, Sills, K, Howard, CJ, Hope, JC, McInnes, CJ, and Entrican, G. Variability in cytokine production and cell proliferation by mitogen-activated ovine peripheral blood mononuclear cells: modulation by interleukin (IL)-10 and IL-12. Vet Immunol Immunopathol. (2004) 102:67–76. doi: 10.1016/j.vetimm.2004.06.006
21. Ciliberti, MG, Albenzio, M, Inghese, C, Santillo, A, Marino, R, Sevi, A, et al. Peripheral blood mononuclear cell proliferation and cytokine production in sheep as affected by cortisol level and duration of stress. J Dairy Sci. (2017) 100:750–6. doi: 10.3168/jds.2016-11688
22. Baharlooi, H, Nouraei, Z, Azimi, M, Moghadasi, AN, Tavassolifar, MJ, Moradi, B, et al. Umbilical cord mesenchymal stem cells as well as their released exosomes suppress proliferation of activated PBMCs in multiple sclerosis. Scand J Immunol. (2021) 93:e13013. doi: 10.1111/sji.13013
23. Subedi, P, Schneider, M, Philipp, J, Azimzadeh, O, Metzger, F, Moertl, S, et al. Comparison of methods to isolate proteins from extracellular vesicles for mass spectrometry-based proteomic analyses. Anal Biochem. (2019) 584:113390. doi: 10.1016/j.ab.2019.113390
24. De Leo, V, Maurelli, AM, Ingrosso, C, Lupone, F, and Catucci, L. Easy preparation of liposome@ PDA microspheres for fast and highly efficient removal of methylene blue from water. Int J Mol Sci. (2021) 22:11916. doi: 10.3390/ijms222111916
25. Aparicio-Puerta, E, Lebrón, R, Rueda, A, Gómez-Martín, C, Giannoukakos, S, Jaspez, D, et al. sRNAbench and sRNAtoolbox 2019: perfil intuitivo rápido de ARN pequeño y expresión diferencial. Ácidos nucleicos Res. (2019) 47:W530–5. doi: 10.1093/nar/gkz415
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Kozomara, A, y Griffiths-Jones, S. miRBase: anotación de microARN de alta confianza utilizando datos de secuenciación profunda. Ácidos nucleicos Res. (2013) 42:D68-73. doi: 10.1093/nar/gkt1181
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Instituto SAS (2013). Guía de SAS Enterprise: estadísticas. Versión 6.1. SAS Inst Inc, Cary, Carolina del Norte.
28. Tarazona, S, Furio-Tari, P, Turra, D, Pietro, AD, y Nueda, MJ. Ferrer a y Conesa un análisis de la calidad de los datos de la expresión diferencial en RNA-seq con el paquete NOISeq R/bioc. Ácidos nucleicos Res. (2015) 43:E140. doi: 10.1093/nar/gkv711
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Tarazona, S, García-Alcalde, F, Dopazo, J, Ferrer, A, y Conesa, A. Expresión diferencial en RNA-seq: una cuestión de profundidad. Genoma Res. (2011) 21:2213–23. doi: 10.1101/gr.124321.111
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Benito-Martin, A, y Peinado, H. Análisis de software de proteómica FunRich, ¡que comience la diversión! Proteómica. (2015) 15:2555–6. doi: 10.1002/pmic.201500260
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Tang, YT, Huang, Y, Zheng, L, Qin, SH, Xu, XP, An, TX, et al. Comparación de métodos de aislamiento de exosomas y ARN exosomal a partir de medio de cultivo celular y suero. Int J Mol Med. (2017) 40:834–44. doi: 10.3892/ijmm.2017.3080
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Soares Martins, T, Catita, J, Martins Rosa, I, AB da Cruz e Silva, O, y Henriques, AG. Aislamiento de exosomas a partir de distintos biofluidos utilizando enfoques basados en precipitación y columnas. PLoS Uno. (2018) 13:e0198820. doi: 10.1371/journal.pone.0198820
33. Do, DN, Dudemaine, PL, Mathur, M, Suravajhala, P, Zhao, X, e Ibeagha-Awemu, EM. funciones reguladoras de miRNA en enfermedades de animales de granja, y potenciales de biomarcadores para terapias efectivas. Int J Mol Sci. (2021) 22:3080. doi: 10.3390/ijms22063080
34. Szatanek, R, Baran, J, Siedlar, M, y Baj-Krzyworzeka, M. Aislamiento de vesículas extracelulares: determinación del enfoque correcto. Int J Mol Med. (2015) 36:11–7. doi: 10.3892/ijmm.2015.2194
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Helwa, I, Cai, J, Drewry, MD, Zimmerman, A, Dinkins, MB, Khaled, ML, et al. Estudio comparativo del aislamiento de exosomas séricos mediante ultracentrifugación diferencial y tres reactivos comerciales. PLoS Uno. (2017) 12:e0170628. doi: 10.1371/journal.pone.0170628
Resumen de PubMed | Texto completo de CrossRef | Google Académico
36. Lane, RE, Korbie, D, Anderson, W, Vaidyanathan, R, y Trau, M. Análisis de métodos de purificación de exosomas utilizando un sistema modelo de liposomas y detección de pulso resistivo sintonizable. Sci Rep. (2015) 5:7639. doi: 10.1038/srep07639
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Álvarez, ML, Khosroheidari, M, Kanchi Ravi, R, y DiStefano, JK. La comparación de proteínas, microARN y ARNm arroja resultados utilizando diferentes métodos de aislamiento de exosomas urinarios para el descubrimiento de biomarcadores de enfermedad renal. Riñón Int. (2012) 82:1024–32. doi: 10.1038/ki.2012.256
38. Linares, R, Tan, S, Gounou, C, Arraud, N y Brisson, AR. La centrifugación de alta velocidad induce la agregación de vesículas extracelulares. J Vesículas extracelulares. (2015) 4:29509. doi: 10.3402/jev.v4.29509
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Rood, IM, Deegens, JK, Merchant, ML, Tamboer, WP, Wilkey, DW, Wetzels, JF, et al. Comparación de tres métodos de aislamiento de microvesículas urinarias para identificar biomarcadores de síndrome nefrótico. Riñón Int. (2010) 78:810–6. doi: 10.1038/ki.2010.262
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Lv, Y, Lu, C, Ji, X, Miao, Z, Long, W, Ding, H, et al. Funciones de los microRNAs en la preeclampsia. J Cell Physiol. (2019) 234:1052–61. doi: 10.1002/jcp.27291
41. Skalis, G, Katsi, V, Miliou, A, Georgiopoulos, G, Papazachou, O, Vamvakou, G, et al. MicroRNAs en la preeclampsia. Microarn. (2019) 8:28–35. doi: 10.2174/2211536607666180813123303
42. Afonso-Grunz, F y Müller, S. Principios de las interacciones miARN-ARNm: más allá de la complementariedad de secuencias. Cell Mol Life Sci. (2015) 72:3127–41. doi: 10.1007/s00018-015-1922-2
43. Tian, F, Luo, J, Zhang, H, Chang, S y Song, J. Firmas de expresión de miARN inducidas por la infección por el virus de la enfermedad de Marek en pollos. Genómica. (2012) 99:152–9. doi: 10.1016/j.ygeno.2011.11.004
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Zhong, B, Zhang, Y, Yan, Y, Wang, Z, Ying, S, Huang, M, et al. Silenciamiento de miostatina mediado por microARN en fibroblastos fetales caprinos. PLoS Uno. (2014) 9:e107071. doi: 10.1371/journal.pone.0107071
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Misiewicz-Krzeminska, I, Krzeminski, P, Corchete, LA, Quwaider, D, Rojas, EA, Herrero, AB, et al. Factores que regulan la expresión y función de los microARN en el mieloma múltiple. ARN no codificante. (2019) 5:9. doi: 10.3390/NCRNA5010009
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Pajares, MJ, Alemany-Cosme, E, Goñi, S, Bandrés, E, Palanca-Ballester, C, y Sandoval, J. Regulación epigenética de los microARN en el cáncer: acortamiento de la distancia desde el laboratorio hasta la cabecera del paciente. Int J Mol Sci. (2021) 22:7350. doi: 10.3390/ijms22147350
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. O’Connell, RM, Taganov, KD, Boldin, MP, Cheng, G y Baltimore, D. El microARN-155 se induce durante la respuesta inflamatoria de los macrófagos. Proc Natl Acad Sci. (2007) 104:1604–9. doi: 10.1073/pnas.061073110
Resumen de PubMed | Texto completo de CrossRef | Google Académico
48. Taganov, KD, Boldin, MP, Chang, KJ y Baltimore, D. Inducción dependiente de NF-κB del microARN miR-146, un inhibidor dirigido a las proteínas de señalización de las respuestas inmunitarias innatas. Proc Natl Acad Sci. (2006) 103:12481–6. doi: 10.1073/pnas.0605298103
Resumen de PubMed | Texto completo de CrossRef | Google Académico
49. Thai, TH, Calado, DP, Casola, S, Ansel, KM, Xiao, C, Xue, Y, et al. Regulación de la respuesta del centro germinal por microRNA-155. Ciencia. (2007) 316:604–8. doi: 10.1126/science.1141229
Resumen de PubMed | Texto completo de CrossRef | Google Académico
50. Breving, K, y Esquela-Kerscher, AA. Las complejidades de la regulación de los microARN: deambulando alrededor de las reglas. Int J Biochem Cell Biol. (2010) 42:1316–29. doi: 10.1016/j.biocel.2009.09.016
Resumen de PubMed | Texto completo de CrossRef | Google Académico
51. Fátima, A, Lynn, DJ, O’Boyle, P, Seoighe, C, y Morris, D. El miRNAoma del hígado de vaca lechera posparto en balance energético negativo. BMC Genómica. (2014) 15:1–8. doi: 10.1186/1471-2164-15-279
Resumen de PubMed | Texto completo de CrossRef | Google Académico
52. Veshkini, A, Hammon, HM, Lazzari, B, Vogel, L, Gnott, M, Tröscher, A, et al. Investigación del miARN circulante en vacas lecheras en transición: lo que dice miRNAomics sobre la adaptación metabólica. Frente Genet. (2022) 13:946211. doi: 10.3389/fgene.2022.946211
Resumen de PubMed | Texto completo de CrossRef | Google Académico
53. Fu, X, Meng, Z, Liang, W, Tian, Y, Wang, X, Han, W, et al. miR-26a mejora la biogénesis del miARN al dirigirse a Lin28B y Zcchc11 para suprimir el crecimiento tumoral y la metástasis. Oncogén. (2014) 33:4296–306. doi: 10.1038/onc.2013.385
Resumen de PubMed | Texto completo de CrossRef | Google Académico
54. Dill, H, Linder, B, Fehr, A y Fischer, U. El miR-26b intrónico controla la diferenciación neuronal reprimiendo su transcripción huésped, ctdsp2. Genes Dev. (2012) 26:25–30. doi: 10.1101/gad.177774.111
Resumen de PubMed | Texto completo de CrossRef | Google Académico
55. Zhu, Y, Lu, Y, Zhang, Q, Liu, JJ, Li, TJ, Yang, JR, et al. El microARN-26a/b y sus genes del huésped cooperan para inhibir la transición G1/S mediante la activación de la proteína pRb. Ácidos nucleicos Res. (2012) 40:4615–25. doi: 10.1093/nar/gkr1278
56. Wang, H, Luo, J, He, Q, Yao, D, Wu, J y Loor, JJ. El análisis del promotor miR-26b revela mecanismos reguladores por factores de transcripción relacionados con lípidos en células epiteliales mamarias de cabras. J Lácteos Sci. (2017) 100:5837–49. doi: 10.3168/jds.2016-12440
Resumen de PubMed | Texto completo de CrossRef | Google Académico
57. Ma, J, Lin, Y, Zhu, J y Huang, K. Wang Y MiR-26b-5p regula la diferenciación de preadipocitos dirigiéndose al FGF 21 en cabras. In Vitro Cell Dev Biol Anim. (2021) 57:257–63. doi: 10.1007/s11626-020-00493-y
Resumen de PubMed | Texto completo de CrossRef | Google Académico
58. Nakamura, K, Kusama, K, Hori, M, e Imakawa, K. Efecto de bta-miR-26b en vesículas extracelulares intrauterinas sobre el sistema inmune materno durante el período de implantación. Biochem Biophys Res Commun. (2021) 573:100–6. doi: 10.1016/j.bbrc.2021.08.019
Resumen de PubMed | Texto completo de CrossRef | Google Académico
59. Huang, N, Li, W, Wang, X y Qi, S. MicroRNA-17-5p agrava la lesión inducida por lipopolisacáridos en las células epiteliales nasales al dirigirse a Smad7. BMC Cell Biol. (2018) 19:1–12. doi: 10.1186/s12860-018-0152-5
60. Cloonan, N, Brown, MK, Steptoe, AL, Wani, S, Chan, WL, Forrest, AR, et al. El microARN miR-17-5p es un regulador clave de la transición del ciclo celular de fase G1/S. Genoma Biol. (2008) 9:R127. doi: 10.1186/gb-2008-9-8-r127
Resumen de PubMed | Texto completo de CrossRef | Google Académico
61. Cuadrado, A, y Nebreda, AR. Mecanismos y funciones de la señalización p38 MAPK. Biochem J. (2010) 429:403–17. doi: 10.1042/BJ20100323
62. Kyriakis, JM y Avruch, J. Vías de transducción de señales MAPK de mamíferos activadas por el estrés y la inflamación: una actualización de 10 años. Physiol Rev. (2012) 92:689–737. doi: 10.1152/physrev.00028.2011
63. Li, Y. La regulación positiva de miR-146a contribuye a la supresión de las respuestas inflamatorias en la lesión pulmonar aguda inducida por LPS. Exp Lung Res. (2013) 39:275–82. doi: 10.3109/01902148.2013.808285
64. Izumi, H, Tsuda, M, Sato, Y, Kosaka, N, Ochiya, T, Iwamoto, H, et al. Los exosomas de la leche bovina contienen microARN y ARNm y son absorbidos por los macrófagos humanos. J Lácteos Sci. (2015) 98:2920–33. doi: 10.3168/jds.2014-9076
Resumen de PubMed | Texto completo de CrossRef | Google Académico
65. Gu, Y, Li, M, Wang, T, Liang, Y, Zhong, Z, Wang, X, et al. Perfiles de expresión de microARN relacionados con la lactancia de exosomas de leche materna porcina. PLoS Uno. (2012) 7:e43691. doi: 10.1371/journal.pone.0043691
Resumen de PubMed | Texto completo de CrossRef | Google Académico
66. Zhou, Q, Li, M, Wang, X, Li, Q, Wang, T, Zhu, Q, et al. Los microARN relacionados con el sistema inmunitario son abundantes en los exosomas de la leche materna. Int J Biol Sci. (2012) 8:118–23. doi: 10.7150/ijbs.8.118
Resumen de PubMed | Texto completo de CrossRef | Google Académico
67. Dysin, AP, y Barkova, OY. Pozovnikova M V El papel de los microARN en el desarrollo, la salud y la función de la glándula mamaria del ganado vacuno, caprino y ovino. ARN no codificante. (2021) 7:78. doi: 10.3390/ncrna7040078
Resumen de PubMed | Texto completo de CrossRef | Google Académico
68. Mecocci, S, Gevi, F, Pietrucci, D, Cavinato, L, Luly, FR, Pascucci, L, et al. Potencial antiinflamatorio de las vesículas extracelulares de leche de vaca, burra y cabra según lo revelado por el perfil metabolómico. Nutrientes. (2020) 12:2908. doi: 10.3390/nu12102908
Resumen de PubMed | Texto completo de CrossRef | Google Académico
69. Mecocci, S, Pietrucci, D, Milanesi, M, Pascucci, L, Filippi, S, Rosato, V, et al. La caracterización transcriptómica de vesículas extracelulares de leche de vaca, burra y cabra revela su potencial antiinflamatorio e inmunomodulador. Int J Mol Sci. (2021) 22:12759. doi: 10.3390/ijms222312759
Resumen de PubMed | Texto completo de CrossRef | Google Académico
70. Quan, S, Nan, X, Wang, K, Jiang, L, Yao, J y Xiong, B. Caracterización del miARN de vesículas extracelulares de la leche de oveja mediante secuenciación y comparación con la leche de vaca. Animales. (2020) 10:331. doi: 10.3390/ani10020331
Resumen de PubMed | Texto completo de CrossRef | Google Académico
71. Teng, GG, Wang, WH, Dai, Y, Wang, SJ, Chu, YX y Li, J. Let-7b está involucrado en la inflamación y las respuestas inmunes asociadas con la infección por Helicobacter pylori al dirigirse al receptor tipo toll 4. PLoS Uno. (2013) 8:e56709. doi: 10.1371/journal.pone.0056709
Resumen de PubMed | Texto completo de CrossRef | Google Académico
72. Bilbao-Arribas, M, Abendaño, N, Varela-Martínez, E, Reina, R, y De Andrés, D. Análisis de la expresión de Jugo B M de miRNAs pulmonares que responden a la infección por el virus de la VM ovina por RNA-seq. BMC Genómica. (2019) 20:1–13. doi: 10.1186/s12864-018-5416-0
73. Iliopoulos, D, Hirsch, HA, y Struhl, K. Un interruptor epigenético que involucra NF-kappaB, Lin28, Let-7 MicroRNA e IL6 vincula la inflamación con la transformación celular. Células. (2009) 139:693–706. doi: 10.1016/j.cell.2009.10.014
74. Li, D, Jia, H, Zhang, H, Lv, M, Liu, J, Zhang, Y, et al. La señalización de TLR4 induce la liberación de micropartículas por parte de las células tumorales que regulan la citoquina inflamatoria IL-6 de los macrófagos a través del microARN let-7b. Onco apunta a Ther. (2012) 1:687–93. doi: 10.4161/onci.19854
Resumen de PubMed | Texto completo de CrossRef | Google Académico
75. Chatterjee, B, Sarkar, M, Bose, S, Alam, MT, Chaudhary, AA, Dixit, AK, et al. MicroRNAs: moduladores clave de las enfermedades asociadas a la inflamación. Semin Cell Dev Biol. (2023) 154:364–73. doi: 10.1016/j.semcdb.2023.01.009
76. Paquette, SG, Banner, D, Zhao, Z, Fang, Y, Huang, S, Leόn, A, et al. La interleucina-6 es un biomarcador potencial de la infección grave por influenza pandémica H1N1. PLoS Uno. (2012) 7:e38214. doi: 10.1371/journal.pone.0038214
Resumen de PubMed | Texto completo de CrossRef | Google Académico
77. Tripathy, AS, Vishwakarma, S, Trimbake, D, Gurav, YK, Potdar, VA, Mokashi, ND, et al. Proinflamatorios CXCL-10, TNF-α, IL-1β e IL-6: biomarcadores de infección por SARS-CoV-2. Arch Virol. (2021) 166:3301–10. DOI: 10.1007/S00705-021-05247-Z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
78. Wu, GS. Las interacciones funcionales entre las vías de señalización MAPK y p53. Cáncer Biol Ther. (2004) 3:156–61. doi: 10.4161/cbt.3.2.614
79. Huang, C, Huang, J, Ma, P y Yu, G. El microARN-143 actúa como un supresor del crecimiento del hemangioma al dirigirse a Bcl-2. Gen. (2017) 628:211–7. doi: 10.1016/j.gene.2017.07.046
Resumen de PubMed | Texto completo de CrossRef | Google Académico
80. Dong, P, Xiong, Y, Hanley, SJ, Yue, J y Watari, H. Musashi-2, una nueva oncoproteína que promueve el crecimiento y la invasión de células de cáncer de cuello uterino, está regulada negativamente por la activación de miR-53 y miR-143 inducida por p107. J Exp Clin Cáncer Res. (2017) 36:150–12. doi: 10.1186/s13046-017-0617-y
Resumen de PubMed | Texto completo de CrossRef | Google Académico
81. Zhou, P, Tu, L, Lin, X, Hao, X, Zheng, Q, Zeng, W, et al. Cfa-mir-143 promueve la apoptosis a través de la vía p53 en células infectadas por el virus de la influenza canina h3n2. Virus. (2017) 9:360. doi: 10.3390/v9120360
Resumen de PubMed | Texto completo de CrossRef | Google Académico
82. Yang, H, Xu, H, Wang, Z, Li, X, Wang, P, Cao, X, et al. Análisis de la inducción mediada por miR-203a-3p/SOCS3 de la polarización de macrófagos M2 para promover la cicatrización de heridas diabéticas basada en exosomas derivados de células madre epidérmicas. Diabetes Res Clin Pract. (2023) 197:110573. doi: 10.1016/j.diabres.2023.110573
83. Nejad, C, y Stunden, HJ. Gantier M P una guía de miRNAs en la inflamación y las respuestas inmunes innatas. FEBS J. (2018) 285:3695–716. doi: 10.1111/febs.14482
Resumen de PubMed | Texto completo de CrossRef | Google Académico
84. Naylor, D, Sharma, A, Li, Z, Monteith, G, Sullivan, T, Cánovas, A, et al. Caracterización de biomarcadores de estrés sérico ovino durante la endotoxemia. J Lácteos Sci. (2020) 103:5501–8. doi: 10.3168/jds.2019-17718
Resumen de PubMed | Texto completo de CrossRef | Google Académico
85. Scalavino, V, Liso, M, Cavalcanti, E, Gigante, I, Lippolis, A, Mastronardi, M, et al. miR-369-3p modulates inducible nitric oxide synthase and is involved in regulation of chronic inflammatory response. Sci Rep. (2020) 10:15942. doi: 10.1038/s41598-020-72991-8
86. Gholami, M, Larijani, B, Zahedi, Z, Mahmoudian, F, Bahrami, S, Omran, S, et al. Inflammation related miRNAs as an important player between obesity and cancers. J Diabetes Metab Disord. (2019) 18:675–92. doi: 10.1007/s40200-019-00459-2
87. Chemonges, S, Tung, JP, and Fraser, JF. Proteogenomics of selective susceptibility to endotoxin using circulating acute phase biomarkers and bioassay development in sheep: a review. Proteome Sci. (2014) 12:12. doi: 10.1186/1477-5956-12-12
Keywords: biomarkers, LPS, cell model, livestock, immune system, small ruminant
Cita: Ciliberti MG, Santillo A, Sevi A, Albenzio M, De Leo V, Ingrosso C, Catucci L y Caroprese M (2023) Primera visión de la caracterización de vesículas extracelulares-miARN en un modelo in vitro de inflamación de ovejas. Frente. Vet. Sci. 10:1186989. doi: 10.3389/fvets.2023.1186989
Recibido: 16 de marzo de 2023; Aceptado: 16 de octubre de 2023;
Publicado: 22 Noviembre 2023.
Editado por:
Josipa Kuleš, Universidad de Zagreb, Croacia
Revisado por:
Michael J. Myers, Administración de Alimentos y Medicamentos de los Estados Unidos, Estados
Unidos Katia Cappelli, Universidad de Perugia, Italia
Copyright © 2023 Ciliberti, Santillo, Sevi, Albenzio, De Leo, Ingrosso, Catucci y Caroprese. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Maria Giovanna Ciliberti, maria.ciliberti@unifg.it
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía


