Rasgos genómicos de aislados enterotoxigénicos de Escherichia coli multirresistentes de cerdos diarreicos

Rasgos genómicos de aislados enterotoxigénicos de Escherichia coli multirresistentes de cerdos diarreicos
 Jiameng Hu1
Jiameng Hu1  Junlin Li1
Junlin Li1  Xiaobo Huang1,2,3
Xiaobo Huang1,2,3  Jing Xia1,2,3
Jing Xia1,2,3  Min Cui1,2,3
Min Cui1,2,3  Yong uang1,2,3
Yong uang1,2,3  Yiping Wen1,2,3
Yiping Wen1,2,3  Yue Xie1,2,3
Yue Xie1,2,3  Qin Zhao1,2,3
Qin Zhao1,2,3  Sanjie Cao1,2,3
Sanjie Cao1,2,3  Likou Zou4*
Likou Zou4*  Xinfeng Han1,2,3*
Xinfeng Han1,2,3*- 1Facultad de Medicina Veterinaria, Universidad Agrícola de Sichuan, Chengdu, China
- 2Centro de Investigación de Ingeniería de Tecnología de Prevención y Control de Enfermedades Animales del Suroeste, Ministerio de Educación de China, Chengdu, China
- 3Laboratorio clave de enfermedades animales y salud humana de la provincia de Sichuan, Chengdu, China
- 4Facultad de Recursos, Universidad Agrícola de Sichuan, Chengdu, China
La diarrea causada por infecciones enterotoxigénicas por Escherichia coli (ETEC) plantea un desafío significativo en la cría mundial de cerdos. Para abordar este problema, el estudio se realizó para identificar y caracterizar 19 aislados de ETEC de muestras fecales de cerdos diarreicos procedentes de granjas a gran escala en la provincia de Sichuan, China. La secuenciación del genoma completo y el análisis bioinformático se utilizaron para la identificación y caracterización. Los aislados mostraron una resistencia sustancial a cefotaxima, ceftriaxona, cloranfenicol, ciprofloxacina, gentamicina, ampicilina, tetraciclina, florfenicol y sulfadiazina, pero fueron altamente susceptibles a amikacina, imipenem y cefoxitina. Se observó diversidad genética entre los aislados, siendo dominantes los serotipos O22:H10, O163orOX21:H4 y O105:H8. Un análisis posterior reveló 53 genes de resistencia y 13 categorías de 195 factores de virulencia. De preocupación fue la presencia de tet(X4) en algunos aislados, lo que indica posibles riesgos para la salud pública. Los aislados de ETEC demostraron la capacidad de producir enterotoxina (ST) termoestable sola o enterotoxina termolábil (LT) y ST simultáneamente, involucrando varios genes de virulencia. En particular, las STa se relacionaron con enfermedades humanas. Además, se identificó la presencia de 4 aislados híbridos ETEC / STEC que albergan factores de virulencia relacionados con la toxina similar a Shiga, a saber, stx2a, stx2b y stx2e-ONT-2771. Predominaron los plásmidos IncF portadores de múltiples genes de resistencia antimicrobiana, y se detectó un plásmido híbrido ETEC/STEC, destacando el papel de los plásmidos en la emergencia del patotipo híbrido. Estos hallazgos enfatizaron la resistencia a múltiples fármacos y la patogenicidad de las cepas ETEC de origen porcino y el riesgo potencial de epidemias a través de la transmisión horizontal de la resistencia a los medicamentos, que es crucial para las estrategias e intervenciones de control efectivas para mitigar el impacto en la salud animal y humana.
1. Introducción
La Escherichia coli enterotoxigénica (ETEC) es una causa importante de diarrea tanto en humanos como en animales (Francis, 2002; Esfandiari et al., 2017). La diarrea post-destete (PCD) generalmente se manifiesta dentro de las 2 semanas posteriores al destete, y se caracteriza por signos clínicos que incluyen diarrea, deshidratación y mortalidad. ETEC es el patógeno aislado con mayor frecuencia de brotes de PCD (Fairbrother et al., 2005), y se distingue por su producción de fimbrias y enterotoxinas. Se puede propagar a través de las heces, el agua y los alimentos. Debido a sus altas tasas de morbilidad y mortalidad, afecta significativamente el rendimiento de los lechones y causa pérdidas económicas significativas en la industria porcina a nivel mundial (Luppi, 2017; García-Meniño et al., 2021).
Aunque los agentes antimicrobianos pueden proporcionar un alivio temporal y ayudar a controlar la carga de morbilidad en los lechones, su uso profiláctico y terapéutico puede conducir al establecimiento de bacterias resistentes a los antimicrobianos en la microbiota intestinal, lo que representa una amenaza tanto para el medio ambiente como para la salud pública. La PCD es una de las razones más comunes para el uso de agentes antimicrobianos en la industria porcina en todo el mundo y contribuye significativamente al problema del desarrollo de la resistencia a los antimicrobianos (RAM) (Luppi, 2017; Rhouma y otros, 2017; Luise et al., 2019; Laird et al., 2021). Los cromosomas y plásmidos desempeñan un papel crucial en el campo de la salud pública, particularmente cuando se trata de resistencia a los antibióticos y la presencia de genes de virulencia. Los genes responsables de la virulencia en las cepas de E. coli pueden transmitirse con frecuencia entre ellas, residiendo en cromosomas, plásmidos o fagos. La aparición de bacterias resistentes a los antibióticos puede atribuirse a mutaciones genéticas dentro de los cromosomas o a la adquisición de material genético, como los plásmidos de resistencia (Bakkeren et al., 2019). Genes AMR como mcr, tet(X4), genes ESBL como blaCTX-M y BLATEM, y genes asociados a Enterobacteriaceae resistentes a carbapenem (CRE) como blaKPC y BLANDM presentan amenazas sustanciales para la salud pública (Faccone et al., 2019; Nguyet et al., 2022; Wang y otros, 2022; Zhang et al., 2022).
La ETEC de los cerdos produce dos enterotoxinas, a saber, la enterotoxina termolábil (LT) y la enterotoxina termoestable (STa y STb), ambas codificadas por plásmidos (Johnson y Nolan, 2009). Existen claras disparidades en las enterotoxinas LT y ST producidas por cepas ETEC porcinas y humanas. A pesar de un alto grado de similitud de secuencia de nucleótidos entre los genes eltAB (pLT y hLT) y los genes estA (pSTa y hSTa), la ausencia de toxinas hLT y hSTa en cepas ETEC porcinas que causan diarrea es notable. Además, existen variaciones en los epítopos y antigenicidades entre las toxinas de origen porcino y humano (Zhang et al., 2008). Los patotipos ETEC en cerdos se caracterizan por adhesinas fimbriales específicas que permiten la colonización bacteriana de la superficie mucosa intestinal. ETEC utiliza fimbrias para adherirse a receptores específicos en las células huésped objetivo y posteriormente coloniza las células epiteliales del intestino delgado de los lechones (Smith y Linggood, 1971). Una vez establecido en el intestino, ETEC secreta enterotoxinas que interrumpen la homeostasis de los líquidos, lo que resulta en diarrea acuosa al aumentar la secreción e inhibir la absorción en el intestino (Dubreuil et al., 2016; Sun y Kim, 2017). Las cepas ETEC que causan diarrea porcina incluyen cinco subtipos fimbriales diferentes, a saber, K88 (F4), K99 (F5), F41, F18 y 987P (F6) (Isaacson, 1977; Isaacson y Richter, 1981; De Graaf y Roorda, 1982; Bakker y otros, 1991; Nagy y otros, 1997). En comparación con las cepas ETEC que infectan a los humanos, las adhesinas predominantes que se encuentran en las cepas ETEC aisladas de cerdos con PWD son las fimbrias F4 y F18 (García et al., 2020). En contraste, las fimbrias F5, F6 y F41 se asocian típicamente con diarrea neonatal inducida por ETEC en lechones, y tienen poca correlación con PWD (Frydendahl, 2002; Zhang y otros, 2007). Las fimbrias de ETEC tienen propiedades inmunogénicas y antigénicas y son el objetivo principal para el desarrollo de vacunas contra este patógeno (Ntakiyisumba et al., 2022). Sin embargo, debido a la prevalencia de infecciones mixtas y la diversidad de tipos de pilus de ETEC, sigue siendo difícil determinar los tipos específicos de pili transportados por individuos infectados, lo que presenta un obstáculo significativo para la prevención y el control efectivos de la enfermedad.
Las cepas de Escherichia coli productoras de toxina Shiga (STEC) son reconocidas mundialmente como causas importantes de diarrea tanto en humanos como en animales (Lee et al., 2023). Estas cepas son conocidas por su capacidad para producir uno o dos tipos distintos de toxina Shiga (Stx), que sirven como el principal factor de virulencia. Las toxinas Shiga, clasificadas como Stx1 y Stx2, juegan un papel crucial en la patogenicidad de STEC (Melton-Celsa, 2014). Estudios previos han demostrado que las cepas ETEC, capaces de producir toxinas Stx, a menudo se clasifican como cepas híbridas ETEC/STEC (Fairbrother et al., 2005).
La secuenciación del genoma completo (WGS) se ha vuelto cada vez más accesible para los laboratorios clínicos y microbiológicos en las últimas décadas debido a una disminución en el costo. Esta tecnología es una herramienta poderosa para analizar genomas bacterianos rápidamente (Kwong et al., 2015). En este estudio, la tecnología WGS se empleó junto con un sofisticado análisis bioinformático para investigar exhaustivamente una multitud de aspectos fundamentales relacionados con los aislados de ETEC. El alcance de la investigación abarcó una exploración en profundidad de la prevalencia de serotipos, fenotipos de resistencia a los antimicrobianos, genes de resistencia a los antimicrobianos (ARG), factores de virulencia (FV) y tipos de incompatibilidad de plásmidos. Además, se realizó un análisis comparativo para dilucidar la relación filogenética entre las cepas ETEC bajo investigación y las cepas ETEC originarias de diversas regiones geográficas en todo el mundo.
Para mejorar la comprensión de la ETEC porcina, es crucial monitorear todo su genoma. Sin embargo, el conocimiento actual se ve obstaculizado por algunas lagunas y limitaciones. Las brechas incluyen un conocimiento limitado de la diversidad genómica, la detección de diferentes serotipos en diferentes regiones, la representación geográfica en bases de datos públicas y el análisis comparativo con cepas ETEC humanas. Esto dificulta la capacidad de evaluar con precisión la diversidad genética global, representar varios linajes y variaciones regionales, y puede dar lugar a un sesgo geográfico en los genomas disponibles. El objetivo principal de este estudio fue lograr una caracterización genómica completa de las cepas ETEC aisladas en China, aportando así datos de referencia que puedan informar significativamente el desarrollo y la implementación de estrategias específicas de control de enfermedades.
2. Materiales y métodos
2.1. Aislamiento bacteriano e identificación de Escherichia coli a partir de muestras fecales
Las muestras de heces diarreicas se recolectaron utilizando hisopos de granjas porcinas a gran escala en la provincia de Sichuan entre 2020 y 2021, que es reconocida como uno de los distritos de cría de cerdos más grandes de China. Se obtuvieron muestras de más de 10 granjas porcinas a gran escala ubicadas en varias regiones de la provincia de Sichuan, China, incluida la ciudad de Wansheng (ciudad de Chengdu), el condado de Dayi (ciudad de Chengdu), el condado de Hongya (ciudad de Meishan) y la ciudad de Qinjia (ciudad de Ya’an). Las muestras recolectadas se transfirieron a tubos de centrífuga de 10 ml llenos de PBS estéril. A cada muestra se le asignó un número único y se almacenó cuidadosamente en cajas de espuma aisladas a lo largo de bolsas de hielo. En 24 h, las muestras se transportaron de vuelta al laboratorio en condiciones de baja temperatura para facilitar el procesamiento posterior. Posteriormente, los hisopos recolectados fueron sometidos a incubación en agar MacConkey, y la presencia de Escherichia coli fue confirmada consultando el Manual de Identificación Bacteriana de Bergey (Holt et al., 1994). La confirmación involucró la realización de múltiples ensayos bioquímicos, incluyendo tinción de gramo, pruebas de oxidasa y catalasa, y evaluaciones metabólicas en diferentes medios como rojo de metilo y caldo Vogues-Proskauer (MR-VP), triple azúcar hierro (TSI), motilidad de sulfuro indol (SIM), citrato de Simmons y agar azul de metileno (EMB). Las colonias que exhiben morfología característica fueron identificadas como Escherichia coli (E. coli). La extracción de ADN genómico de aislados de E. coli se realizó utilizando el kit de ADN de bacterias TIANamp (Tiangen Biotech) para su posterior identificación.
2.2. Identificación molecular de ETEC
Con el fin de identificar ETEC fuera de las colonias de E. coli, se diseñaron cebadores específicos dirigidos a secuencias LT y ST de acuerdo con el informe anterior (Tabla 1) (Zhang et al., 2007). La mezcla de reacción por PCR contenía 12,5 μL de Premix Taq™, 1 μL de cada cebador aguas arriba y aguas abajo, 2 μL de muestras de ADN extraídas y 8,5 μL de ddH.2O, para un volumen total de 25 μL. El programa de amplificación por PCR consistió en una desnaturalización inicial a 94°C durante 5 min, seguida de 30 ciclos de desnaturalización a 95°C durante 30 s, recocido durante 30 s y extensión a 72 s°C durante 30 s. Los productos de PCR se separaron en un gel de agarosa al 1% y se visualizaron utilizando un generador de imágenes en gel. Las cepas ETEC identificadas se conservaron a -20 ° C en caldo Luria-Bertani (LB) suplementado con 20% de glicerol.
2.3. Pruebas de sensibilidad antimicrobiana
Para determinar el perfil de susceptibilidad antimicrobiana de los aislados de ETEC, se utilizó el método de dilución de microcaldo siguiendo las pautas del Instituto de Estándares Clínicos y de Laboratorio (CLSI) (CLSI, 2021). Se probó una variedad de agentes antimicrobianos, incluyendo amikacina (AMK), gentamicina (GEN), estreptomicina (STR), cefoxitina (FOX), cefotaxima (CTX), ceftriaxona (CRO), ampicilina (AMP), imipenem (IPM), polimixina B (PMB), tetraciclina (TET), cloranfenicol (CHL), florfenicol (FFC), ciprofloxacina (CIP), ácido nalidíxico (NAL) y sulfasalazina (SSZ), y se registraron sus valores mínimos de concentración inhibitoria (CMI). Los valores de CMI se compararon con los últimos estándares CLSI, con E. coli ATCC25919 sirviendo como cepa de control de calidad. Los resultados fueron categorizados como susceptibles (S), intermedios (I) o resistentes (R). Los aislados que mostraban resistencia a agentes antimicrobianos de tres o más clases se definieron como multirresistentes (MDR).
2.4. Secuenciación del genoma completo y análisis bioinformático
El ADN genómico se envió a Shanghai Ling’en Biotechnology Co. Ltd. (Shanghai, China) para su secuenciación, y su calidad y concentración se evaluaron utilizando un sistema NanoDrop2000 y un fluorómetro Qubit 4. El NEBNext Ultra™ II DNA Library Prep Kit se utilizó para construir bibliotecas utilizando ADN calificado. Estas bibliotecas se secuenciaron en una plataforma Illumina NovaSeq 6000 utilizando una secuenciación de 150 pb por protocolo de síntesis. Para garantizar la calidad de los datos, se excluyeron las lecturas sin procesar de baja calidad en función de criterios predefinidos. El ensamblaje de datos siguió a la eliminación de la contaminación del adaptador y al filtrado de datos mediante AdapterRemoval y SOAPec. Las lecturas de alta calidad se ensamblaron en contigs del genoma utilizando SPAdesv 3.13.0.
Para predecir y analizar la resistencia a los antibióticos, la Base de Datos Integral de Genes de Resistencia a los Antibióticos (CARD)1 y ResFinder 4.12 estaban empleados. Para la predicción y análisis de genes de virulencia, la Base de Datos de Factores de Virulencia de Bacterias Patógenas (VFDB)3 y VirulenceFinder 2.04 se utilizaron. Durante el proceso de búsqueda en ResFinder 4.1, los umbrales para la identidad génica y la longitud mínima se establecieron en 90% y 60%, respectivamente. Del mismo modo, la base de datos VirulenceFinder empleó umbrales del 95% para la identidad génica y del 60% para la cobertura de alineación durante el proceso de análisis. La plataforma de análisis en línea PlasmidFinder 2.15 se utilizó para determinar los tipos de incompatibilidad de plásmidos (Carattoli et al., 2014). La base de datos PlasmidFinder utilizó una cobertura de alineación mínima del ≥60% y requirió un porcentaje de identidad del ≥95% a la secuencia de referencia.
2.5. Serotipo, ST y análisis filogenético
La predicción del serotipo se realizó utilizando la plataforma de análisis en línea EnteroBase.6 El MLST 2.0 – Software del Centro de Epidemiología Genómica7 se utilizó para analizar los tipos de secuencia (ST) de todos los aislados mediante la aplicación de la tipificación de secuencias multilocus (MLST). El análisis de tipificación de secuencia multilocus del genoma central (cgMLST) se realizó utilizando el esquema 2513 locus cgMLST V1 + HierCC V1 proporcionado por EnteroBase. La construcción del árbol cgMLST utilizó el algoritmo MSTree V2 del esquema cgMLST V1 + HierCC V1. Para facilitar las comparaciones con las cepas aisladas en este estudio, las secuencias probadas derivadas de genomas ETEC disponibles en la base de datos Enterobase se incluyeron en el análisis para generar el árbol cgMLST. Un total de 102 genomas ETEC fueron cuidadosamente seleccionados de 13 países, incluidos los Estados Unidos, el Reino Unido, Canadá, Francia, Alemania y Dinamarca, etc. Estos genomas de bacterias se obtuvieron de diversos orígenes, incluidos los humanos, la vida silvestre, el ganado, el medio ambiente, las aves de corral y los alimentos. El árbol de expansión mínimo se visualizó utilizando el software GrapeTree (Zhou et al., 2018).
2.6. Análisis de datos
Heml 2.0 se empleó para construir mapas de calor que muestran la distribución de ARG, VF y grupos de incompatibilidad de plásmidos (Ning et al., 2022). El esquema de puntuación empleado en este estudio asignó un valor de 1 o 0 para denotar la presencia o ausencia del gen designado, respectivamente.
3. Resultados
3.1. Identificación molecular de ETEC por PCR
Para identificar posibles colonias de ETEC, se aplicaron cebadores de genes LT y ST específicos de ETEC en la detección de PCR. La Figura 1 muestra los resultados de la electroforesis en gel de agarosa para aislados representativos de E. coli. En este estudio, se confirmaron un total de 19 aislados de ETEC de 412 cepas de E. coli aisladas de muestras fecales de cerdos, lo que resultó en una tasa de recuperación del 4,61%.
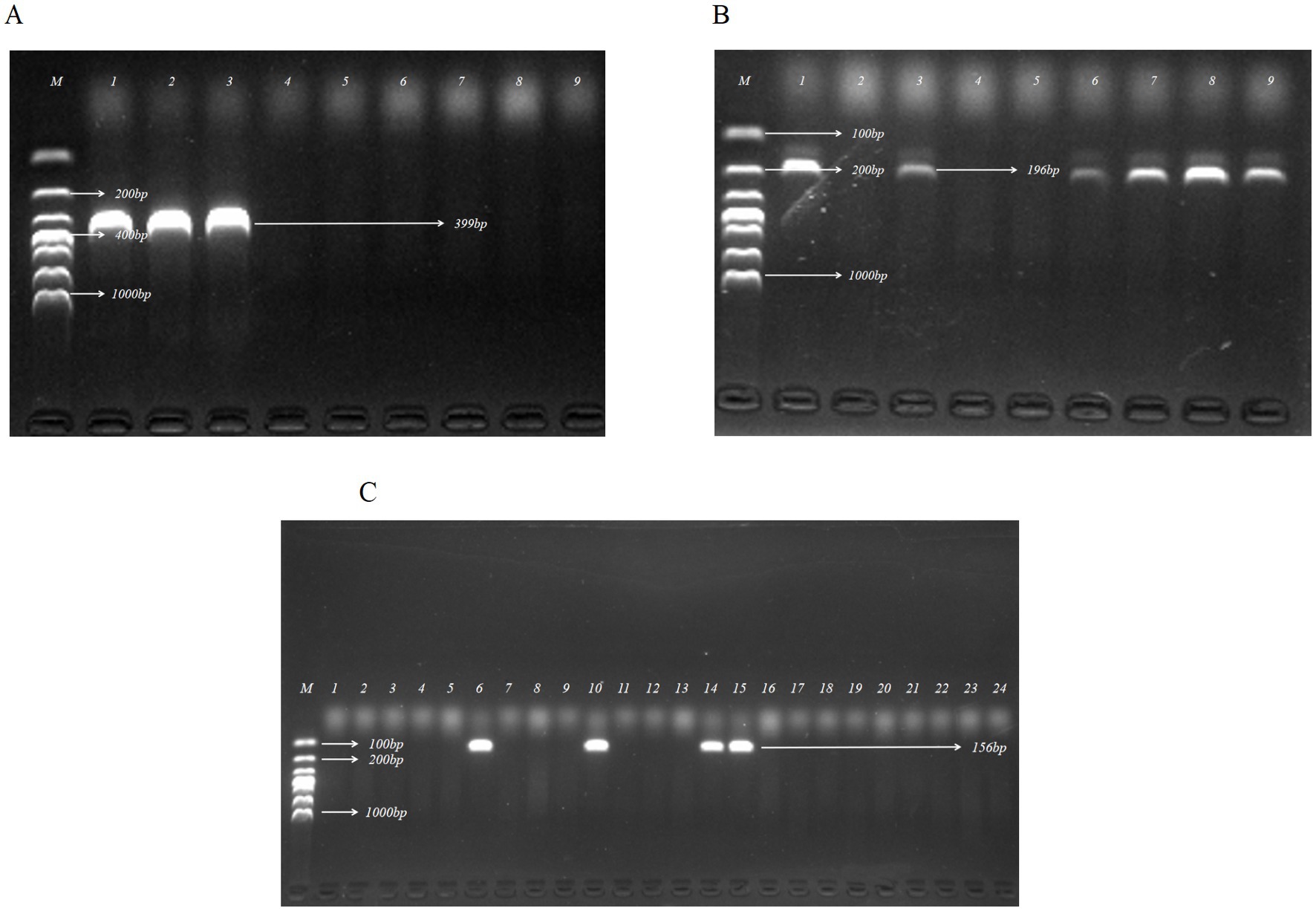 Figura 1. Identificación de ETEC mediante ensayo de PCR dirigido a genes de enterotoxinas de LT, STa y STb. (A) Electroforesis en gel de agarosa de los productos de PCR para la detección de LT en aislados parciales de E. coli. (B) Electroforesis en gel de agarosa de los productos de PCR para la detección de STa en aislados parciales de E. coli. (C) Electroforesis en gel de agarosa de los productos de PCR para la detección de STb en aislados parciales de E. coli. Carriles: A1Un2Un3, B1, B3, B6, B7, B8, B9, C6, C10, C14, C15, aislados que fueron positivos para genes del factor de virulencia; M, marcador de ADN DL 1000 (100, 200, 300, 400, 500, 700, 1.000 pb).
Figura 1. Identificación de ETEC mediante ensayo de PCR dirigido a genes de enterotoxinas de LT, STa y STb. (A) Electroforesis en gel de agarosa de los productos de PCR para la detección de LT en aislados parciales de E. coli. (B) Electroforesis en gel de agarosa de los productos de PCR para la detección de STa en aislados parciales de E. coli. (C) Electroforesis en gel de agarosa de los productos de PCR para la detección de STb en aislados parciales de E. coli. Carriles: A1Un2Un3, B1, B3, B6, B7, B8, B9, C6, C10, C14, C15, aislados que fueron positivos para genes del factor de virulencia; M, marcador de ADN DL 1000 (100, 200, 300, 400, 500, 700, 1.000 pb).
3.2. Fenotipos de resistencia a los antimicrobianos
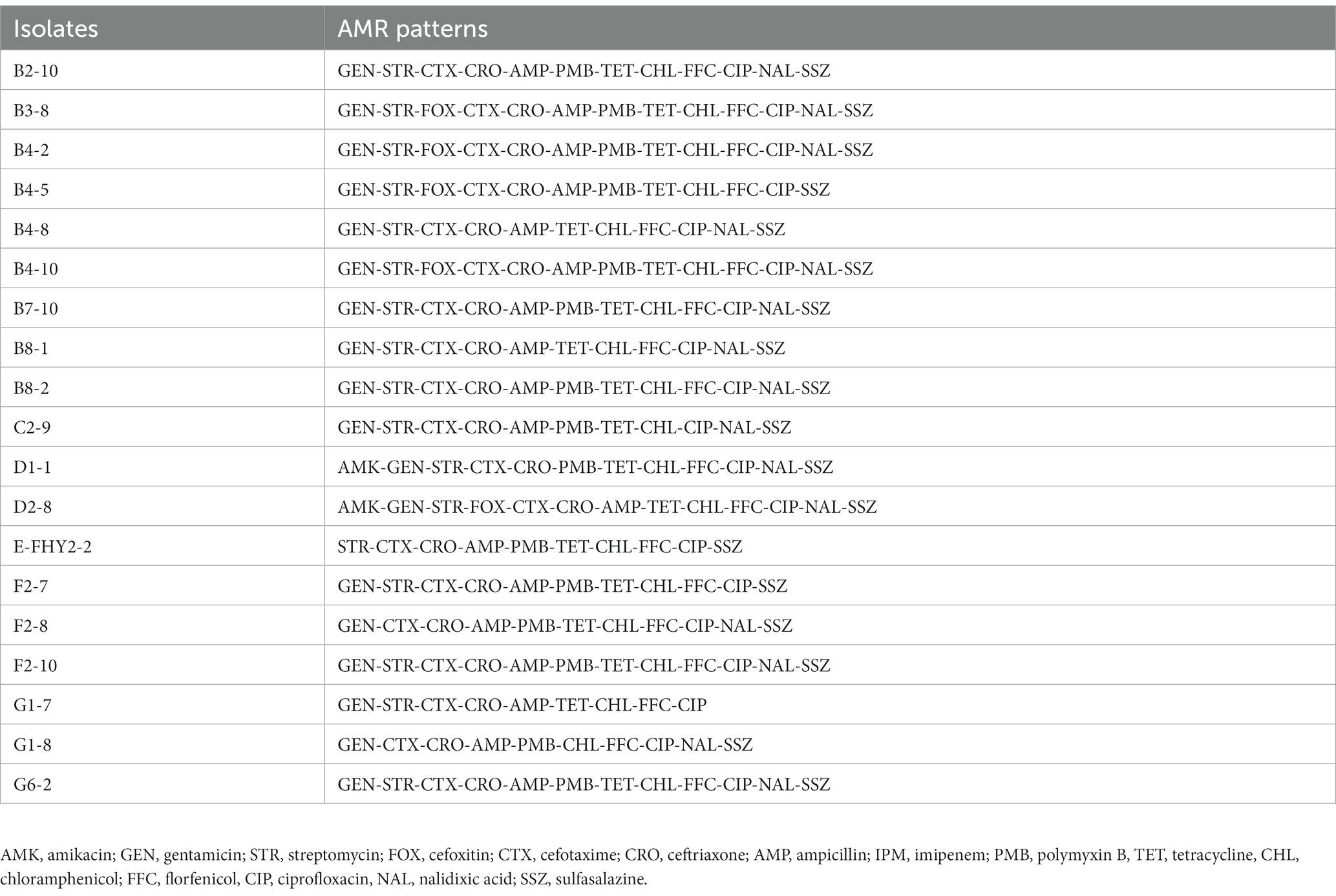
El método de dilución de microcaldo se empleó para evaluar la RAM de los aislados de ETEC, y los resultados demostraron una resistencia significativa a CTX (100%, 19/19), CRO (100%, 19/19), CHL (100%, 19/19), CIP (100%, 19/19) y tasas de resistencia superiores al 94% a GEN, AMP, TET, FFC y SSZ (Figura 2A). En contraste, los aislamientos mostraron altas tasas de susceptibilidad a AMK (84,21%, 16/19), MIP (78,95%, 15/19) y FOX (63,16%, 12/19). Además, se observó una marcada divergencia en los perfiles de RAM entre los aislados (Figura 3), con los perfiles de RAM predominantes de GEN-STR-CTX-CRO-AMP-PMB-TET-CHL-FFC-CIP-NAL-SSZ (26,32%, 5/19), GEN-STR-FOX-CTX-CRO-AMP-PMB-TET-CHL-FFC-CIP-NAL-SSZ (15,79%, 3/19) y GEN-STR-CTX-CRO-AMP-TET-CHL-FFC-CIP-NAL-SSZ (10,53%, 2/19) (Tabla 2). Una evaluación adicional de los aislados de MDR reveló que todas las cepas eran resistentes a un mínimo de 9 agentes antimicrobianos y un máximo de 13 agentes antimicrobianos, con la mayoría (36,84%, 7/19) de los aislados mostraron resistencia a 12 agentes antimicrobianos, y una proporción significativa (26,32%, 5/19) se encontró que era resistente a 11 agentes antimicrobianos (Figura 2B).
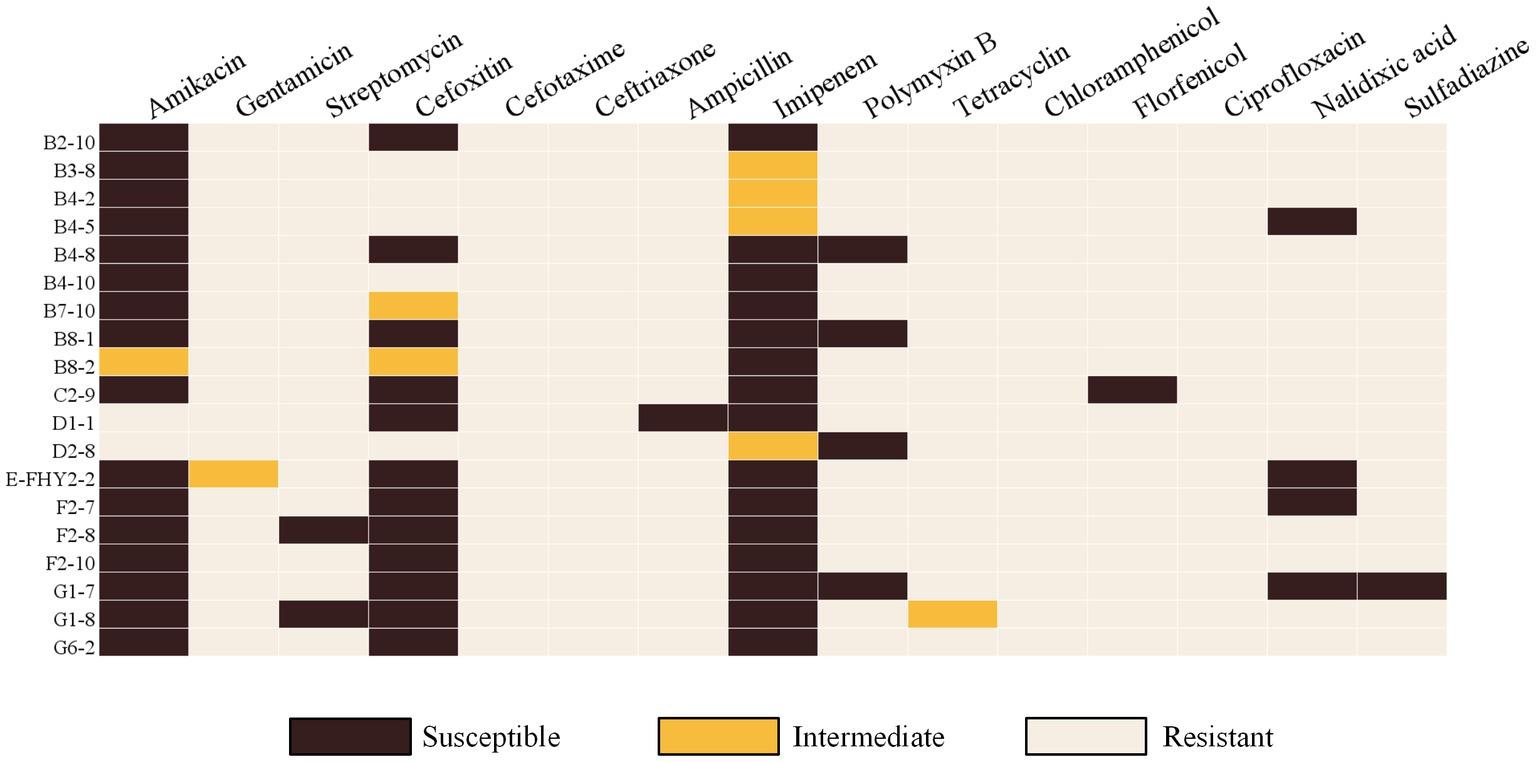 Figura 3. Mapa de calor que muestra los resultados de las pruebas de sensibilidad antimicrobiana de aislados de ETEC de muestras fecales de cerdos con diarrea en granjas porcinas a gran escala en Sichuan, China. El marrón oscuro indica fenotipo susceptible y el marrón claro indica fenotipo resistente.
Figura 3. Mapa de calor que muestra los resultados de las pruebas de sensibilidad antimicrobiana de aislados de ETEC de muestras fecales de cerdos con diarrea en granjas porcinas a gran escala en Sichuan, China. El marrón oscuro indica fenotipo susceptible y el marrón claro indica fenotipo resistente.
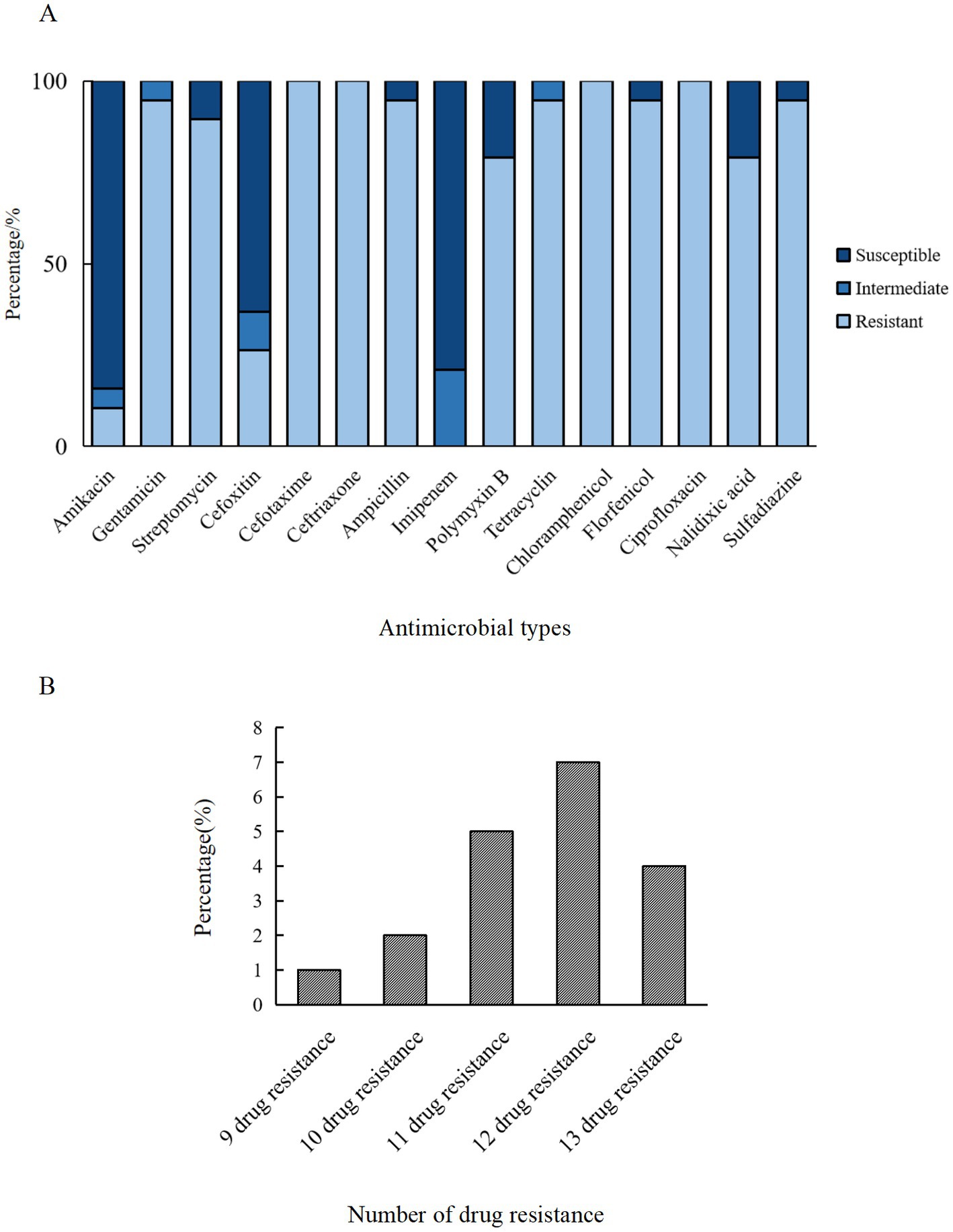 Figura 2. Características de la RAM de aislados de ETEC de muestras fecales de cerdos con diarrea en granjas porcinas a gran escala en Sichuan, China. (A) La proporción de cepas susceptibles, intermedias y resistentes de cada agente antimicrobiano. (B) El porcentaje de aislados de ETEC con cada tipo de cepa MDR. Amikacina (AMK), gentamicina (GEN), estreptomicina (STR), cefoxitina (FOX), cefotaxima (CTX), ceftriaxona (CRO), ampicilina (AMP), imipenem (IPM), polimixina B (PMB), tetraciclina (TET), cloranfenicol (CHL), florfenicol (FFC), ciprofloxacino (CIP), ácido nalidíxico (NAL) y sulfasalazina (SSZ).
Figura 2. Características de la RAM de aislados de ETEC de muestras fecales de cerdos con diarrea en granjas porcinas a gran escala en Sichuan, China. (A) La proporción de cepas susceptibles, intermedias y resistentes de cada agente antimicrobiano. (B) El porcentaje de aislados de ETEC con cada tipo de cepa MDR. Amikacina (AMK), gentamicina (GEN), estreptomicina (STR), cefoxitina (FOX), cefotaxima (CTX), ceftriaxona (CRO), ampicilina (AMP), imipenem (IPM), polimixina B (PMB), tetraciclina (TET), cloranfenicol (CHL), florfenicol (FFC), ciprofloxacino (CIP), ácido nalidíxico (NAL) y sulfasalazina (SSZ).
3.3. Presencia de ARG y mutaciones puntuales cromosómicas en ETEC
En total, se detectaron 53 ARGS en aislados de ETEC. El gen de resistencia detectado con mayor frecuencia fue tet(A) (68,42%, 13/19), que codifica una proteína transportadora de tetraciclina que reduce la concentración de tetraciclina transportándola desde el interior hacia el exterior de la célula. Además, otros ARG, incluyendo floR (63.16%, 12/19), aph(3′)-Ia (52.63%, 10/19), aadA2 (47.37%, 9/19), bleO (47.37%, 9/19), sul3 (47.37%, 9/19), dfrA12 (47.37%, 9/19) y QnrS1 (47.37%, 9/19), proporcionaron resistencia a varios agentes antimicrobianos, como quinolonas, aminoglucósidos, cloranfenicol y sulfonamidas. Es importante tener en cuenta que estos ARG pueden presentar resistencia cruzada a otras clases de antibióticos, lo que reduce la efectividad de otros fármacos. De manera alarmante, se detectó la presencia de tet(X4) en 2 aislamientos (10,53%), lo que indica la presencia del gen asociado con resistencia a tigeciclina de alto nivel. Este hallazgo destacó la aparición de genes de resistencia a la tigeciclina mediados por plásmidos (Sun et al., 2019). La región determinante de la resistencia a las quinolonas (QRDR) es un segmento específico del genoma bacteriano que contiene ciertas regiones de los genes gyrA y parC, que son sitios críticos para la interacción de las quinolonas con las topoisomerasas de ADN bacteriano. Las mutaciones en el QRDR podrían conducir al desarrollo de resistencia a las quinolonas. Se observaron mutaciones puntuales cromosómicas en QRDR en los genes gyrA (42,11%, 8/19) y parC (42,11%, 8/19) en aislados de ETEC. La mutación observada exclusivamente en gyrA fue S83L (100%), mientras que las mutaciones en parC se detectaron comúnmente como S80R (37,5%), A56T (37,5%) y S80I (25%). Es importante destacar que los genotipos de los aislados estaban fundamentalmente alineados con sus fenotipos de resistencia a los antimicrobianos (Figura 4).
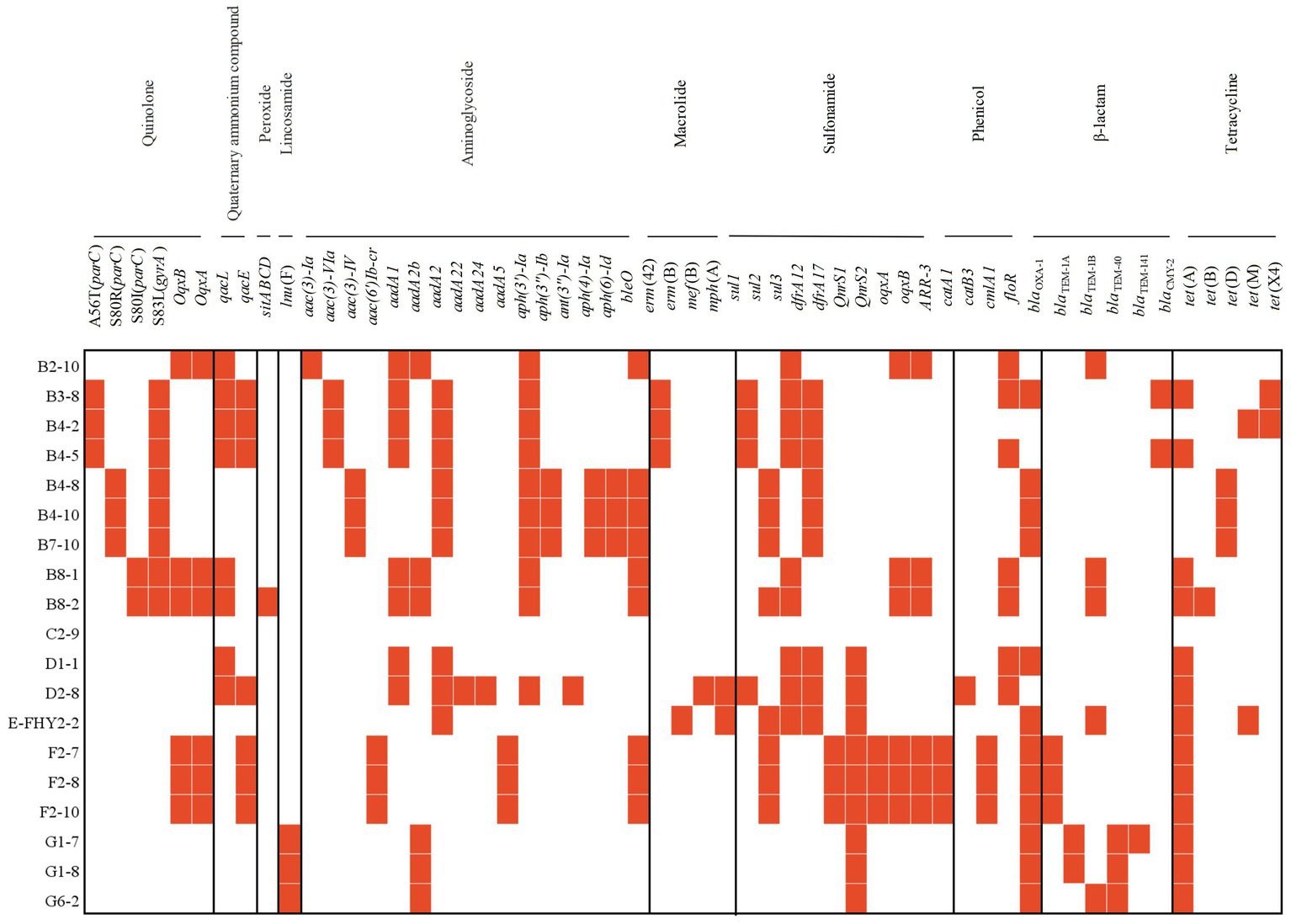 Figura 4. Mapa de calor que muestra los ARG adquiridos o perfiles de mutación puntual cromosómica de aislados de ETEC de muestras fecales de cerdos con diarrea en granjas porcinas a gran escala en Sichuan, China.
Figura 4. Mapa de calor que muestra los ARG adquiridos o perfiles de mutación puntual cromosómica de aislados de ETEC de muestras fecales de cerdos con diarrea en granjas porcinas a gran escala en Sichuan, China.
3.4. Análisis de los determinantes asociados a la virulencia
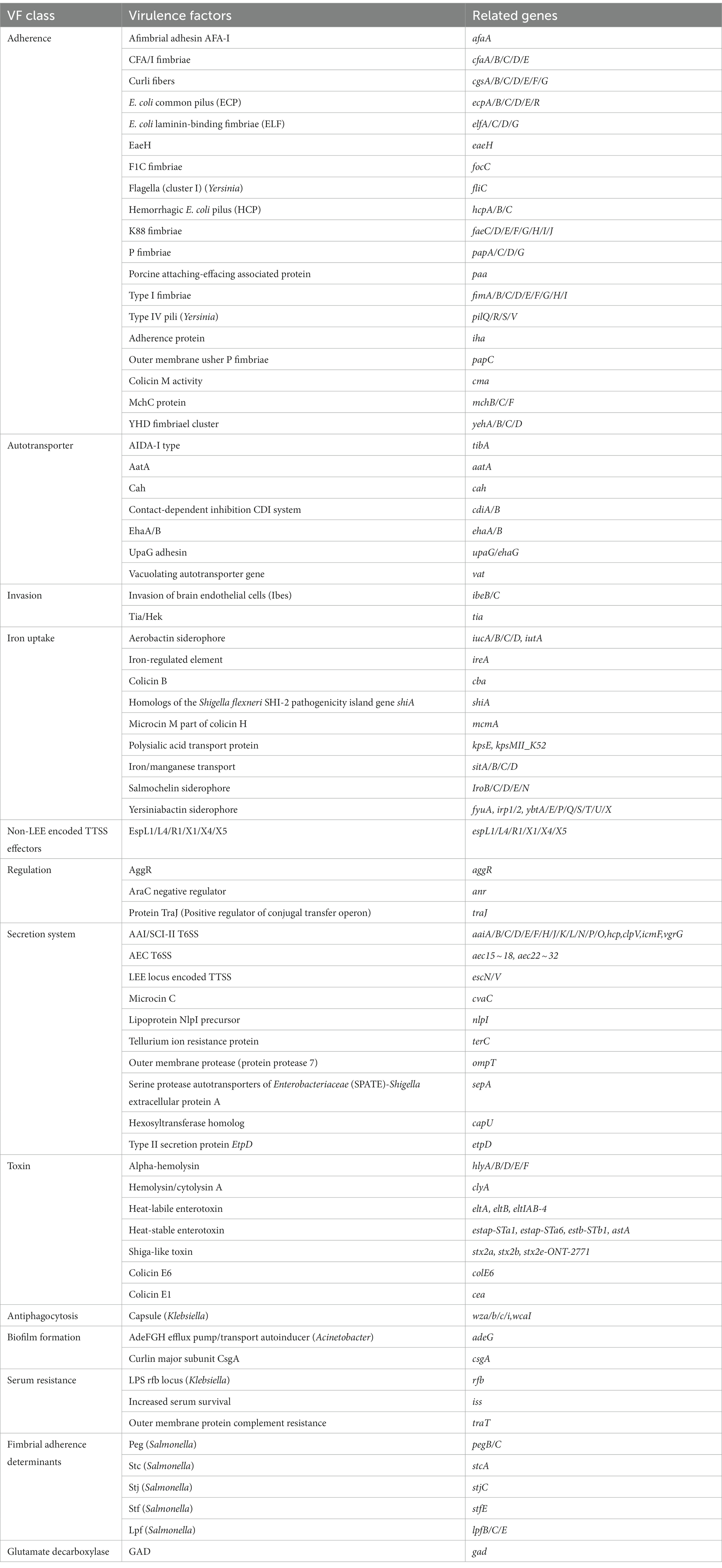
El análisis genético de los determinantes de virulencia en VFDB ha revelado la presencia de un conjunto diverso de genes de virulencia en 19 aislados de ETEC que se dividen en 13 tipos diferentes, totalizando 195 genes (Figura 5), cuyas funciones incluían adherencia, autotransportador, invasión, captación de hierro, efectores TTSS no codificados en LEE, regulación, sistema de secreción, toxina, antifagocitosis, formación de biopelículas, resistencia sérica, determinantes de adherencia fimbrial y glutamato descarboxilasa. Los genes de adherencia constituyeron la categoría más grande, con un total de 65 genes de virulencia identificados en ETEC (Figura 5B). Los genes de adherencia desempeñaron un papel vital en la patogénesis de ETEC, codificando varias adhesinas que facilitan la unión bacteriana a las células huésped y el establecimiento de la infección. Los diversos genes de adherencia, como la adhesina afina afimbrial AFA-I, CFA/I fimbrias, fibras curli, E. coli común pilus (ECP), E. coli laminin-binding fimbriae (ELF), eaeH, F1C fimbriae, hemorrágica E. coli pilus (HCP), K88 fimbriae, P fimbriae, porcino atuning-borring associated protein, type I fimbriae y type IV pili, sugirieron que ETEC ha desarrollado múltiples mecanismos para adherirse a las células huésped y evadir las defensas inmunes del huésped (Tabla 3).
 Figura 5. Mapa de calor que muestra el gen de virulencia en aislados de ETEC de muestras fecales de cerdos con diarrea en granjas porcinas a gran escala en Sichuan, China. (A) Los genes de virulencia asociados con toxinas. (B) Los genes de virulencia asociados con la adherencia. (C) Los genes de virulencia restantes no asociados con toxinas o adherencia.
Figura 5. Mapa de calor que muestra el gen de virulencia en aislados de ETEC de muestras fecales de cerdos con diarrea en granjas porcinas a gran escala en Sichuan, China. (A) Los genes de virulencia asociados con toxinas. (B) Los genes de virulencia asociados con la adherencia. (C) Los genes de virulencia restantes no asociados con toxinas o adherencia.
La investigación reveló la presencia de una amplia gama de genes LT, a saber, eltA, eltB y eltIAB-4, así como genes ST, que abarcan estap-STa1, estap-STa6, estb-STb1 y astA, dentro de los aislados ETEC identificados (Figura 5A). Cabe destacar que la tasa de detección de estb-STb1 exhibió la mayor frecuencia, observada en 18 aislamientos (94,74%), mientras que astA se encontró en 11 aislados (57,89%). En contraste, la presencia de eltB y estap-STa6 fue excepcionalmente baja, con solo un aislado (5,26%) exhibiendo su presencia. STEC se distingue por la presencia de genes stx1 o stx2. Los aislados analizados en este estudio mostraron la detección de genes stx2, incluyendo stx2a (15,79%), stx2b (5,26%) y stx2e-ONT-2771 (15,79%). Los 4 aislados (21,05%), que portaban genes stx, fueron identificados como cepas híbridas.
Además, cabe destacar que los genes de cgsG, elfG, hcpB, hcpC, papC, fimD, ibeB, ibeC, espL1, espL4, espR1, espX1 y espX5 estaban presentes en los 19 aislados de ETEC, lo que indica su papel conservado y esencial en la patogénesis y virulencia de ETEC. Los genes identificados codificaron una variedad de proteínas, incluyendo proteínas de membrana, chaperonas, proteínas relacionadas con la invasión, proteínas efectoras del sistema de secreción tipo III, reguladores transcripcionales y proteínas esenciales para el ensamblaje y secreción de fimbrias. Estos productos génicos funcionan en el transporte de membranas, el plegamiento de proteínas, la invasión y la biogénesis de las fimbrias, entre otros procesos celulares.
3.5. Grupos de incompatibilidad de plásmidos
Los resultados indicaron que se identificó una amplia gama de replicones plásmidos entre los aislados de ETEC, con 17 tipos distintos detectados. En particular, se encontró que los 19 aislamientos albergaban al menos un gen asociado al plásmido replicón, y se detectaron múltiples replicones en el 94,74% (18/19) de los aislados. La distribución de cepas dentro del grupo de incompatibilidad mostró variabilidad, con una cepa que contenía un solo replicón (5,26%), mientras que otras albergaban 3, 4, 5, 6 o 7 replicones, que representaban el 26,32%, 10,53%, 26,32%, 26,32% y 5,26% de los aislados, respectivamente. Estas observaciones sugirieron que la mayoría de los aislados de ETEC contenían al menos dos o más plásmidos, siendo IncFIB (AP001918) un tipo de replicón plásmido presente en todas las cepas bacterianas (100%, 19/19), seguido de los plásmidos IncFII e IncX1, ambos detectados en el 52,63% (10/19) de los aislados (Figura 6).
 Figura 6. Mapa de calor que muestra los perfiles de replicón plásmido para aislados de ETEC de muestras fecales de cerdos con diarrea en granjas porcinas a gran escala en Sichuan, China.
Figura 6. Mapa de calor que muestra los perfiles de replicón plásmido para aislados de ETEC de muestras fecales de cerdos con diarrea en granjas porcinas a gran escala en Sichuan, China.
3.6. Serotipo, ST y análisis filogenético
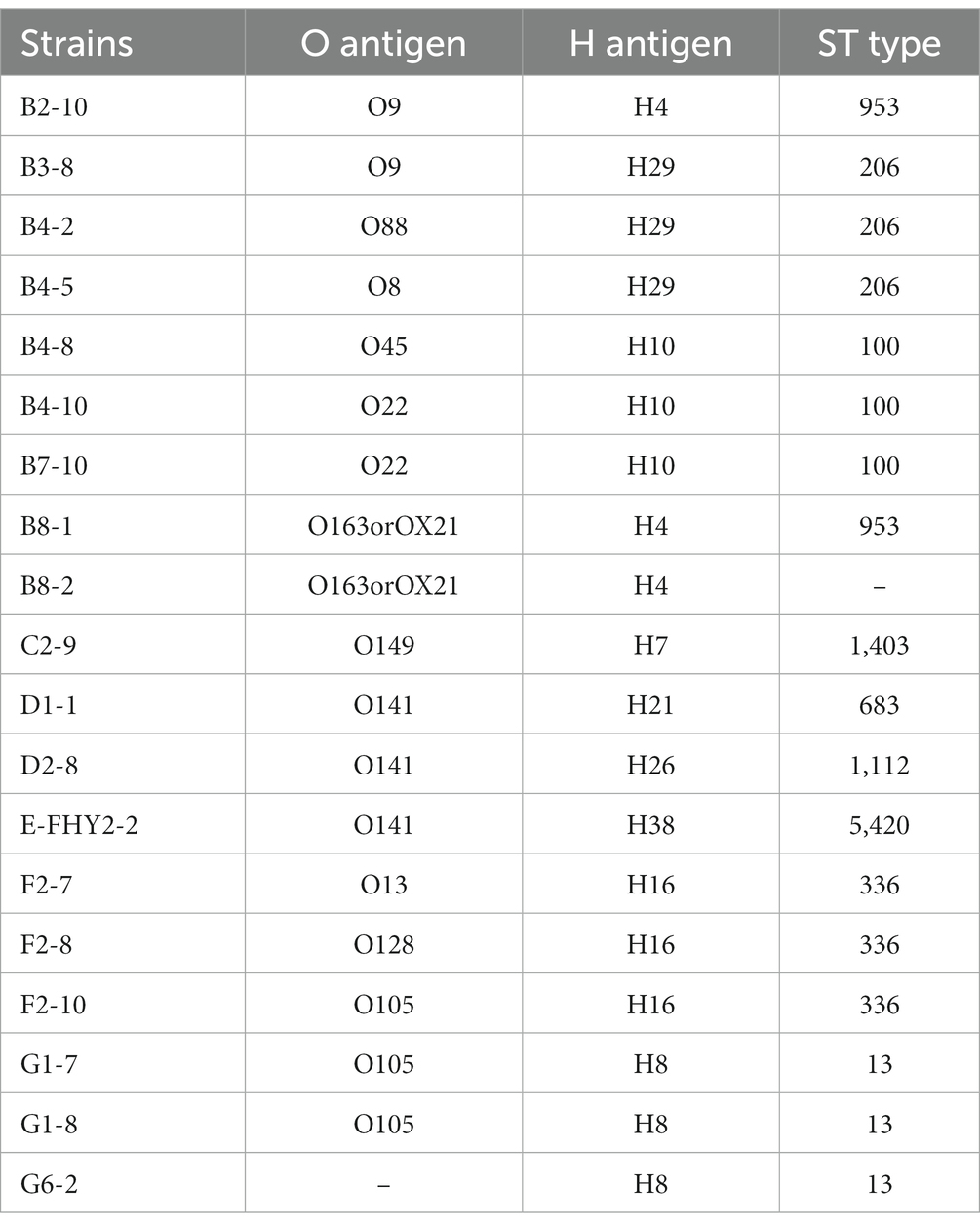
A través de la secuenciación de Illumina, se examinaron 19 cepas MDR ETEC, revelando un grupo diverso de serogrupos O y serogrupos H, que los serotipos O22: H10 (10.53%, 2/19), O163 u OX21: H4 (10.53%, 2/19) y O105: H8 (10.53%, 2/19) fueron dominantes. Los aislados se sometieron a análisis MLST para determinar los ST. El análisis fue exitoso para 18 aislamientos, revelando la presencia de 9 ST diferentes. Sin embargo, no se pudo definir un aislado. Entre las ST identificadas, ST13 (15,79%, 3/19), ST100 (15,79%, 3/19), ST206 (15,79%, 3/19) y ST336 (15,79%, 3/19) fueron las más comunes (Tabla 4).
Para investigar las características filogenéticas de los aislados de ETEC, se construyó un árbol de expansión mínimo utilizando el análisis cgMLST para clasificar 121 aislados de ETEC en 2 grupos (Figura 7). Sin embargo, el árbol de expansión mínimo no reveló ninguna relación obvia entre las ST observadas. Sorprendentemente, diversas fuentes tenían aislados del mismo grupo, lo que indica la diseminación generalizada de ETEC entre animales, alimentos y humanos (Figura 7A). Los resultados sugirieron que el linaje de aislados de ECET no se asoció significativamente con la distribución geográfica, ya que diferentes tipos de ST estaban ampliamente distribuidos en varios países (Figura 7B). Además, no se observó un patrón significativo en la prevalencia de ST en diferentes años (Figura 7C).
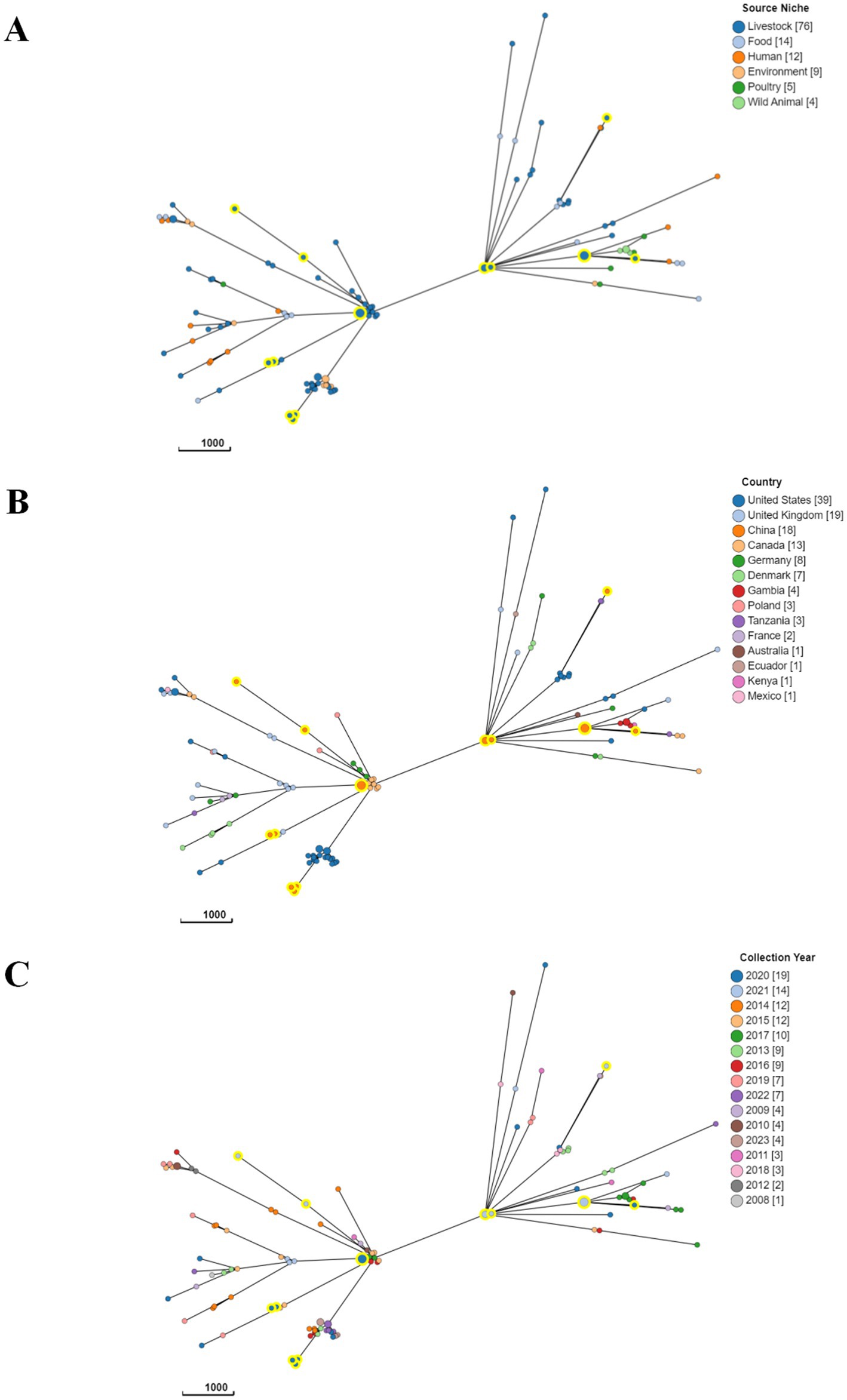 Figura 7. Se generó un árbol de expansión mínima del genoma central para demostrar la relación filogenética de ETEC basada en fuentes (A), país de origen (B) y tiempo de recolección (C). Se generó un árbol de expansión mínimo para 121 aislados ETEC a partir de datos cgMLST del esquema 2513 locus cgMLST V1 + HierCC V1, que está disponible dentro de EnteroBase. Los aislados fueron anotados por sus (A) fuentes, (B) país, (C) año de aislamiento. El árbol MSTree V2 se creó basado en cgMLST, con nodos coloreados de acuerdo con los clústeres definidos en el estudio. Los aislados caracterizados en el estudio fueron rodeados con un gran halo amarillo para facilitar la identificación.
Figura 7. Se generó un árbol de expansión mínima del genoma central para demostrar la relación filogenética de ETEC basada en fuentes (A), país de origen (B) y tiempo de recolección (C). Se generó un árbol de expansión mínimo para 121 aislados ETEC a partir de datos cgMLST del esquema 2513 locus cgMLST V1 + HierCC V1, que está disponible dentro de EnteroBase. Los aislados fueron anotados por sus (A) fuentes, (B) país, (C) año de aislamiento. El árbol MSTree V2 se creó basado en cgMLST, con nodos coloreados de acuerdo con los clústeres definidos en el estudio. Los aislados caracterizados en el estudio fueron rodeados con un gran halo amarillo para facilitar la identificación.
4. Discusión
La diarrea resultante de la infección por ETEC es frecuente entre los cerdos e impone cargas económicas significativas a la industria porcina a nivel mundial. La diarrea resultante en los lechones se atribuye en gran medida a ETEC y puede tener efectos perjudiciales en el crecimiento y el rendimiento de la producción, presentando un obstáculo tecnológico significativo para la producción porcina contemporánea (Gresse et al., 2017). Sin embargo, a pesar de su importancia, las características genómicas y la variación de ETEC de origen porcino han sido poco investigadas. Para abordar esta brecha, se aplicó WGS para identificar aislados de ETEC de muestras de heces de cerdo obtenidas de granjas a gran escala en Sichuan, China. Nuestro estudio tuvo como objetivo aclarar los serotipos, AMR, ARG, VF y tipos de incompatibilidad de plásmidos de ETEC asociados con diarrea en cerdos.
Las cepas ETEC tienen implicaciones significativas para la salud animal y humana. En los cerdos, la colonización por ETEC puede conducir a enfermedades entéricas graves, lo que resulta en impactos adversos en las tasas de crecimiento, la conversión alimenticia y las pérdidas económicas en la industria ganadera. Del mismo modo, en los seres humanos, se sabe que ETEC causa diarrea aguda, que afecta particularmente a las poblaciones vulnerables como bebés, niños pequeños y personas en regiones en desarrollo con acceso limitado a agua potable e instalaciones de saneamiento. En particular, ETEC es un agente causal prominente de la diarrea del viajero, lo que representa una amenaza sustancial para la salud humana (Guiral et al., 2019). Las medidas efectivas para mitigar la transmisión de ETEC abarcan mejores prácticas de saneamiento, acceso a agua limpia y cumplimiento de protocolos de higiene adecuados para las poblaciones animales y humanas. Además, la implementación de programas de vacunación dirigidos que aborden específicamente las cepas de ETEC puede contribuir significativamente a la prevención de infecciones y a la reducción general de la carga de la enfermedad. Por lo tanto, es de suma importancia obtener información sobre los perfiles de RAM y las características genómicas completas de ETEC.
En el presente estudio, los serotipos dominantes previstos en granjas porcinas a gran escala en la provincia china de Sichuan fueron O105, O141, H8, H10, H16 y H29, que diferían de estudios previos realizados en otras regiones de China. Por ejemplo, en granjas porcinas a gran escala en el norte de Jiangsu, los serotipos dominantes de ETEC fueron O8, O101 y O128 (Yang et al., 2020). Del mismo modo, en ciertas áreas de la provincia de Anhui, las granjas porcinas mostraron una prevalencia de O8, O20 y O128 como los serotipos predominantes de ETEC, con niveles generalmente altos de virulencia (Ren et al., 2020). Estos resultados indicaron que ETEC con un grupo diverso de serotipos circulaban en cerdos, con una diversidad significativa en los serotipos observados tanto dentro como entre las regiones. Además, nuestro análisis WGS reveló la distribución de ST100 entre los aislados de ETEC MDR de granjas porcinas, que se consideró una cepa clásica de ETEC porcino (patógeno animal no zoonótico) (Abraham et al., 2015). Los estudios existentes han demostrado que el serogrupo O149 (ST100) es la causa predominante de PWD en cerdos australianos (Abraham et al., 2014). La variedad de serotipos en ETEC es significativa para la gravedad de la enfermedad, la vigilancia y el desarrollo de vacunas. Ayuda a rastrear la propagación de cepas, identificar brotes y seleccionar antígenos relevantes para vacunas dirigidas a serotipos específicos.
El análisis abarcó una evaluación exhaustiva de 121 aislados de ETEC en todo el mundo utilizando el análisis cgMLST, un método altamente preciso para la caracterización de subtipos basado en variaciones genómicas. El árbol de expansión mínimo, derivado de este análisis, proporcionó información sobre la relación genética entre los aislados de ETEC. Sin embargo, el análisis no reveló patrones distintos de relación, ya que diferentes ST se dispersaron en varios grupos. Además, el mismo grupo acomodó aislados procedentes de diversas fuentes, incluidos animales, medio ambiente, alimentos y humanos. Estos resultados indicaron una amplia distribución de ETEC a través de múltiples reservorios y rutas de transmisión, con una asociación limitada entre la diversidad genética de los aislados de ETEC y sus orígenes geográficos o temporales.
La ausencia de patrones visibles en los datos de cgMLST puede atribuirse a una variedad de factores subyacentes, que contribuyen colectivamente a la naturaleza elusiva de las relaciones observadas. Principalmente, la diversidad genética sustancial prevalente entre los aislados de ETEC jugó sin duda un papel fundamental en la intrincada red de asociaciones dentro de sus perfiles genómicos. ETEC muestra una notable diversidad genética que surge de mecanismos como mutaciones y reordenamientos genéticos. En consecuencia, se pueden encontrar distintos conjuntos de genes, incluidos los que codifican VF o AMR, entre las cepas ETEC. La amplia gama de variaciones genéticas planteó desafíos significativos para descifrar las relaciones definitivas entre estos aislados. Además, la ocurrencia recurrente de eventos de transferencia horizontal que involucran elementos genéticos móviles complica aún más la composición genómica de los aislados de ETEC. Estos elementos genéticos móviles tienen la capacidad de moverse a través de cepas bacterianas, lo que lleva a una configuración de genomas similar a un mosaico. Esto facilita el intercambio de material genético, incluidos los FV y los ARG, configurando así la composición genética de las poblaciones de ETEC. Estos eventos de intercambio genético tendieron a desdibujar las demarcaciones entre diferentes linajes, oscureciendo así cualquier asociación directa anticipada. Además, la compleja historia evolutiva de este microorganismo patógeno, esculpida por diversas presiones selectivas, ejerce potencialmente un profundo impacto en los patrones genéticos observados. La adaptación juega un papel clave, impulsando el desarrollo de rasgos que mejoran la supervivencia y la reproducción en entornos específicos. En el caso de ETEC, la adaptación permite la colonización de los ambientes del huésped, la evasión de las respuestas inmunes y la adquisición de nutrientes. La deriva genética, un proceso estocástico, introduce fluctuaciones aleatorias en las frecuencias alélicas a lo largo del tiempo, lo que lleva a la divergencia de linajes y contribuye a la diversidad genética. Los efectos combinados de la adaptación, la deriva genética y otras fuerzas contribuyen en última instancia a los patrones genéticos observados, influyendo en la evolución y diversidad de las poblaciones de ETEC. En consecuencia, la intrincada arquitectura genética resultante planteó un obstáculo significativo para identificar asociaciones inequívocas dentro de los datos de cgMLST.
Las pruebas de sensibilidad antimicrobiana in vitro son una herramienta crucial que proporciona orientación para la práctica clínica veterinaria. La detección de aislados de ETEC MDR de cerdos con diarrea en granjas planteó una situación preocupante de RAM. El estudio demostró que todos los aislados de ETEC eran MDR. En comparación, las tasas de resistencia de las cepas ETEC recolectadas en Bangladesh a la ampicilina (66%), cotrimoxazol (46%), doxiciclina (44%), eritromicina (96%), ácido nalidíxico (83%), estreptomicina (48%) y tetraciclina (42%) difirieron en cierta medida de nuestros hallazgos (Begum et al., 2016). La variación geográfica en el uso de antimicrobianos y los patrones de resistencia es una explicación probable, ya que diferentes regiones pueden tener diferentes niveles de uso de antibióticos en la agricultura, lo que lleva a variaciones en la prevalencia y los tipos de bacterias resistentes. Las prácticas locales de prescripción de antibióticos y el uso de antibióticos veterinarios también contribuyen a estas diferencias. Además, factores como las cepas específicas de ETEC que circulan en diferentes poblaciones, los mecanismos genéticos de resistencia adquiridos por las bacterias y las variaciones en los métodos de vigilancia utilizados en diferentes investigaciones pueden contribuir a las disparidades en las tasas de resistencia observadas. A pesar de la prohibición del uso de CHL en la producción animal en China desde 2002 (Peng et al., 2022), en este estudio se encontraron aislados resistentes a CHL en granjas porcinas en Sichuan, potencialmente atribuidos al uso continuo de FFC, un miembro de la clase cloranfenicol (Yang et al., 2019). En contraste, no se encontraron aislados resistentes al MIP en el estudio, muy probablemente debido a la política oficial que prohíbe el uso de carbapenem en el ganado en China. Aunque la prohibición del uso de AMK en la agricultura en 2017 resultó en un bajo porcentaje (10.53%, 2/19) de aislados resistentes a AMK en granjas porcinas, la alta tasa de resistencia a PMB (78.95%, 15/19) de aislados de ETEC en este estudio indicó que el uso indebido de drogas en la industria porcina sigue siendo una preocupación importante (Shen et al., 2020; Wang et al., 2020).
Afortunadamente, a pesar de que la mayoría de las cepas muestran resistencia a PMB, el gen mcr-1 no se observó en el presente estudio, que se identificó por primera vez en 2016 en aislados bacterianos de alimentos de origen animal (pollos y cerdos) en China (Liu et al., 2016). Este gen de resistencia mediado por plásmidos codifica una enzima que pertenece a la familia de las fosfoetanolamina transferasas y es responsable de sintetizar y conjugar pEtN al lípido A (Ayoub Moubareck, 2020). Otros genes mcr, incluidos mcr-2 y mcr-3, también se han encontrado en plásmidos entre enterobacterias (Zhang et al., 2018). Estos genes de resistencia sugirieron la presencia de varias vías para la transmisión horizontal de la resistencia a la colistina, lo que podría conducir a su alto potencial de diseminación. Con base en nuestros resultados, se sospechó que la resistencia a PMB observada en los aislados de ETEC no puede atribuirse a la transferencia horizontal. Estudios longitudinales en China encontraron que la reducción de las ventas de colistina se asoció con una disminución de la incidencia de E. coli productora de mcr-1 en las heces de cerdo y los intestinos humanos. Las infecciones humanas con E. coli resistente a la colistina también disminuyeron después de 2017, posiblemente debido a la prohibición del gobierno chino de la colistina como promotor del crecimiento animal (Ewers et al., 2022).
Cabe destacar que detectamos la presencia de tet(X4) en 2 (10,53%) aislados de ETEC. Según investigaciones anteriores, las cepas de E. coli portadoras de tet(X4) pueden representar un riesgo de transmisión a los humanos, ya que exhibieron un parecido genético significativo con la E. coli comensal humana. Aunque tet(X4) no se distribuyó ampliamente en todo el país, fue altamente endémica en el noroeste de China (Sun et al., 2020). Entre los 19 aislados de ETEC, tet(A) fue el ARG detectado con mayor frecuencia, representando el 68,42%. Una posible explicación de la prevalencia relativamente baja del gen de resistencia a la tigeciclina fue la presencia de ARG relacionados con TET, como tet (A), tet (B), tet (C) y / o tet (M), que a menudo son transportados por cepas de E. coli de animales (Foong et al., 2020). La propagación mundial de E. coli resistente a la colistina y CRE plantea importantes desafíos de salud pública. En consecuencia, la tigeciclina se ha convertido en un antibiótico crucial de último recurso para tratar ciertas infecciones bacterianas (Zhang et al., 2022). Dada la aparición de resistencia a la tigeciclina mediada por tet(X4), nuestros hallazgos subrayaron la necesidad urgente de una mayor vigilancia y medidas de intervención para prevenir una posible diseminación de estos ARG.
La identificación de los FV de ETEC en cerdos es crucial para comprender su patogenicidad y transmisión, así como para desarrollar estrategias efectivas de prevención y tratamiento. Este estudio identificó varios genes esenciales, incluyendo cgsG, elfG, hcpB, hcpC, papC, fimD, ibeB, ibeC, espL1, espL4, espR1, espX1 y espX5, que desempeñan un papel importante en la patogénesis y la virulencia. La presencia de estos genes en todos los aislados de ETEC demostró su papel crítico en la virulencia de ETEC. Además, la investigación reveló la capacidad de los aislados de ETEC para producir potencialmente ST solo o toxinas LT y ST simultáneamente. Además, esta observación subrayó la importancia de múltiples genes de virulencia, como eltA, eltB, eltIAB-4, estap-STa1, estap-STa6, estb-STb1 y astA, en la síntesis potencial de toxinas. STa se ha implicado en la enfermedad humana, mientras que STb se asocia predominantemente con la infección por ETEC en cerdos, aunque la presencia del gen stb se ha observado en aislados de ETEC humanos. Estudios previos han demostrado que STa4 está frecuentemente relacionado con la diarrea (Leonard et al., 2016). Los plásmidos son el sitio predominante para la presencia de STa (von Mentzer et al., 2014), y es posible la transferencia de plásmidos portadores de genes de toxina ST sta entre diferentes cepas de E. coli (Turner et al., 2006). Dos variantes genéticas distintas de STa, a saber, STp y STh, se identifican en aislados de ETEC humanos. Los hallazgos indican que las cepas productoras de STa3/4 (STh) se correlacionan con la enfermedad en los niños, mientras que las cepas STa5 (STp) están asociadas con la enfermedad en adultos (Joffré et al., 2016). Extensas investigaciones han establecido la presencia del gen astA, que codifica la enterotoxina enteroagregativa termoestable EAST1 en ETEC vinculada a la aparición de enfermedades diarreicas en cerdos (Renzhammer et al., 2020). Sin embargo, el papel preciso de esta toxina en la enfermedad sigue siendo un tema de debate en curso, ya que ni las cepas positivas para EAST1 ni los sobrenadantes de cultivo inducen consistentemente diarrea explícita en modelos animales (Dubreuil, 2019). Cabe destacar que una proporción significativa de las cepas examinadas en este estudio, específicamente el 57,89% (11/19) de los aislados analizados, exhibieron la presencia del gen astA. Este hallazgo destacó la ocurrencia generalizada de este determinante genético crítico entre los aislados de ETEC. Las FV relacionadas con la adhesina, como ECP, ELF y HCP, también contribuyeron a la patogenicidad de ETEC al interactuar con enterotoxinas, mientras que los factores de virulencia relacionados con toxinas similares a Shiga, incluidos stx2a, stx2b y stx2e-ONT-2771, se identificaron con efectos enterotóxicos, citotóxicos y neurotóxicos (Ahsan et al., 2020 ). Además, la aparición de cuatro aislados híbridos se confirmó en este estudio, lo que puede atribuirse a la frecuente incidencia de eventos de transferencia horizontal de genes (HGT). Durante la HGT, los antecedentes genéticos específicos adquieren genes de virulencia que definen el patotipo y que con frecuencia se albergan en elementos genéticos móviles (Leonard et al., 2016). Stx2e, en particular, es una toxina crucial responsable de la enfermedad del edema en lechones, que a menudo acompaña a la diarrea de los lechones (Tseng et al., 2014). Por lo tanto, analizar los síntomas de los lechones por sí solo es inadecuado para diagnosticar la enfermedad con precisión. Una investigación exhaustiva de ETEC VF puede proporcionar un examen en profundidad para el desarrollo de métodos de diagnóstico efectivos y vacunas multivalentes con amplia protección contra este patógeno y otras infecciones bacterianas similares.
La adquisición de ARG se ve facilitada por una amplia gama de elementos genéticos móviles. Entre estos elementos, los plásmidos son particularmente significativos para promover la difusión de ARG (Bennett, 2008). La evaluación de la diversidad de plásmidos en diversas cohortes se puede lograr utilizando grupos de incompatibilidad de plásmidos. El presente estudio reveló diferencias significativas en los grupos de incompatibilidad prevalentes en las granjas porcinas a gran escala en el suroeste de China. La diseminación de plásmidos epidémicos se ha relacionado con genes específicos, y en este estudio, se identificaron replicadores comunes como IncFIB, IncFII e IncX1. Además, ciertos grupos de estos plásmidos, como IncC, IncFIB, IncX1, IncFIIA e IncY, tienen la capacidad de transportar funciones de transferencia, resistencia a múltiples fármacos y virulencia a través de una amplia gama de huéspedes (Johnson y Nolan, 2009). IncFII ha sido reconocido como un plásmido que disemina múltiples resistencia a medicamentos (Wagner et al., 2014), e IncF está implicado en la transmisión de genes de resistencia a medicamentos y virulencia de Enterobacteriaceae (Hopkins et al., 2006). La posibilidad de transmisión de cepas de E. coli MDR, especialmente aquellas que producen β-lactamasas (BLEE) de espectro extendido, de animales a humanos es una preocupación creciente (Jackson et al., 2015). Los tipos de plásmidos IncC e IncX se han reportado recientemente como los más comunes entre E. coli resistente a carbapenem (Kopotsa et al., 2019). Este estudio encontró que la presencia del plásmido IncC era exclusiva de las cepas híbridas, lo que estaba en línea con la existencia del gen tet (X4). Anteriormente se informó que la existencia de un plásmido híbrido ETEC/STEC multivirulencia IncFII/IncX119 de 1 kb (p15ODTXV) que portaba genes de virulencia de patotipos ETEC y STEC desempeñó un papel fundamental en la aparición de patotipos híbridos, enfatizando la importancia de los plásmidos en este mecanismo (Brilhante et al., 2019). En este estudio, se aclaró un plásmido híbrido ETEC/STEC híbrido de múltiples virulencias IncFII/IncX1 comparable en la cepa híbrida, proporcionando evidencia adicional del papel significativo de los plásmidos en la aparición de patotipos híbridos. La transferencia y transmisión de plásmidos desempeñó un papel crítico en la diseminación de genes de virulencia y resistencia a medicamentos en E. coli patógena. La transmisión de plásmidos de animales a humanos puede conducir a la propagación de la RAM, infecciones zoonóticas, aumento de la virulencia y desafíos en el tratamiento de infecciones por ETEC. Por lo tanto, la comprensión de los grupos de incompatibilidad de los plásmidos ETEC porcinos puede informar el desarrollo de estrategias para controlar y prevenir las infecciones por ETEC en cerdos
5. Conclusión
En este estudio, se empleó una investigación para analizar los aislados de ETEC de cerdos con diarrea en China. Los aislados de ETEC exhibieron niveles notables de resistencia a múltiples fármacos, con la aparición de genes tet (X4) móviles en aislados híbridos específicos, lo que planteó un desafío para la industria porcina y la salud pública. Además, el estudio identificó múltiples plásmidos capaces de facilitar la transferencia horizontal de genes de resistencia y virulencia a múltiples fármacos a través de diversos organismos huéspedes, lo que tiene el potencial de exacerbar la diseminación de rasgos de resistencia, lo que representa una amenaza sustancial para la industria porcina. Además, se encontró que las cepas bajo investigación albergan varias FV asociadas con la patogénesis y la gravedad de las enfermedades animales. La ausencia de patrones conspicuos en los datos de cgMLST entre los aislados de ETEC, sugirió su amplia distribución a través de diferentes reservorios y rutas de transmisión. Estos resultados demostraron la necesidad urgente de intensificar las medidas de vigilancia para monitorear eficazmente la aparición, la resistencia y la posible transmisión de ETEC entre los rebaños porcinos en China.
Declaración de disponibilidad de datos
Los conjuntos de datos generados para este estudio se pueden encontrar en el NCBI Bioproject con el número de acceso PRJNA983238.
Contribuciones del autor
JH: investigación, software, curación de datos, redacción de borradores originales y adquisición de fondos. JL: análisis formal. XHu y QZ: metodología. JX, MC, YX y SC: redacción-revisión y edición. YH y YW: visualización. LZ: metodología, supervisión, revisión y edición, y adquisición de fondos. XHa: conceptualización, administración de proyectos, recursos y adquisición de fondos. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Financiación
Este trabajo fue apoyado por la subvención de la Fundación de Ciencias Naturales de Sichuan, China (2023NSFSC0175) y el Programa de Ciencia y Tecnología de Sichuan (2022ZDZX0017).
Conflicto de intereses
Los autores declaran que no tienen intereses financieros o relaciones personales contradictorias conocidos que podrían haber parecido influir en el trabajo reportado en este documento.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fmicb.2023.1244026/full#supplementary-material
Notas
1. ^ https://card.mcmaster.ca/
2. ^ https://cge.food.dtu.dk/services/ResFinder/
3. ^ http://www.mgc.ac.cn/cgi-bin/VFs/v5/main.cgi
4. ^ https://cge.food.dtu.dk/services/VirulenceFinder/
5. ^ https://cge.food.dtu.dk/services/PlasmidFinder/
6. ^ https://enterobase.warwick.ac.uk/
7. ^ https://cge.food.dtu.dk/services/MLST/
Referencias
Abraham, S., Jordan, D., Wong, H. S., Johnson, J. R., Toleman, M. A., Wakeham, D. L., et al. (2015). Primera detección de Escherichia coli resistente a cefalosporinas y fluoroquinolonas de espectro extendido en animales australianos productores de alimentos. J. Glob. Antimicrobiano. Resistir. 3, 273–277. doi: 10.1016/j.jgar.2015.08.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Abraham, S., Trott, D. J., Jordan, D., Gordon, D. M., Groves, M. D., Fairbrother, J. M., et al. (2014). Perspectivas filogenéticas y moleculares sobre la evolución de Escherichia coli enterotoxigénica porcina resistente a múltiples fármacos en Australia. Int. J. Antimicrobiano. Agentes 44, 105–111. doi: 10.1016/j.ijantimicag.2014.04.011
Ahsan, C. R., Begum, K., Kabir, E., Nur-E-Kamal, A. y Talukder, K. A. (2020). Enterotóxico, neurotóxico y citotóxico demostrado por la toxina Shiga (2d) produciendo escherichia coli en modelos experimentales. Bangladesh Med. Toro. 46, 41–47. DOI: 10.3329/bmrcb.v46i1.47468
Ayoub Moubareck, C. (2020). Polimixinas y membranas bacterianas: una revisión de la actividad antibacteriana y los mecanismos de resistencia. Membranas 10:181. DOI: 10.3390/membranas10080181
Bakker, D., Vader, C., Roosendaal, B., Mooi, F., Oudega, B. y De Graaf, F. (1991). Estructura y función de proteínas peripiásmicas tipo chaperona implicadas en la biosíntesis de fimbrias K88 y K99 en Escherichia coli enterotoxigénica. Mol. Microbiol. 5, 875–886. doi: 10.1111/j.1365-2958.1991.tb00761.x
Bakkeren, E., Huisman, J. S., Fattinger, S. A., Hausmann, A., Furter, M., Egli, A., et al. (2019). Los persistentes de Salmonella promueven la propagación de plásmidos de resistencia a los antibióticos en el intestino. Nature 573, 276–280. DOI: 10.1038/S41586-019-1521-8
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Begum, Y. A., Talukder, K., Azmi, I. J., Shahnaij, M., Sheikh, A., Sharmin, S., et al. (2016). Patrón de resistencia y caracterización molecular de cepas enterotoxigénicas de Escherichia coli (ETEC) aisladas en Bangladesh. PLoS One 11:e0157415. doi: 10.1371/journal.pone.0157415
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Bennett, P. M. (2008). Resistencia a antibióticos codificada por plásmidos: adquisición y transferencia de genes de resistencia a antibióticos en bacterias. Fr. J. Pharmacol. 153, S347–S357. doi: 10.1038/sj.bjp.0707607
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Brilhante, M., Perreten, V. y Donà, V. (2019). Plásmidos de resistencia a múltiples fármacos y multivirulencia en Escherichia coli enterotoxigénica e híbrida productora de toxina Shiga/enterotoxigénica aislada de cerdos diarreicos en Suiza. Veterinario J. 244, 60–68. doi: 10.1016/j.tvjl.2018.12.015
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Carattoli, A., Zankari, E., García-Fernández, A., Voldby Larsen, M., Lund, O., Villa, L., et al. (2014). Detección in silico y tipificación de plásmidos mediante buscador de plásmidos y tipificación de secuencias multilocus de plásmidos. Antimicrobiano. Agentes Chemother. 58, 3895–3903. doi: 10.1128/AAC.02412-14
Resumen de PubMed | Texto completo de CrossRef | Google Académico
CLSI (2021). Normas de rendimiento para las pruebas de sensibilidad a los antimicrobianos, 31. Documento CLSI M100. Wayne, PA: Instituto de Estándares Clínicos y de Laboratorio.
De Graaf, F., y Roorda, I. (1982). Producción, purificación y caracterización del antígeno adhesivo fimbrial F41 aislado de la cepa B41M de Escherichia coli enteropatógena de terneros. Infectar. Inmune. 36, 751–758. DOI: 10.1128/IAI.36.2.751-758.1982
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Dubreuil, J. D. (2019). Toxina EAST1: Molécula enigmática asociada con episodios esporádicos de diarrea en humanos y animales. J. Microbiol. 57, 541–549. DOI: 10.1007/S12275-019-8651-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Dubreuil, J. D., Isaacson, R. E. y Schifferli, D. M. (2016). Escherichia coli enterotoxigénica animal. EcoSal Plus 7:10.1128/ecosalplus. ESP-0006-2016. DOI: 10.1128/Ecosalplus. ESP-0006-2016
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Esfandiari, P., Amani, J., Imani Fouladi, A. A., Nazarian, S., Mirhosseini, A. y Moghimi, E. (2017). Ensayo inmunoabsorbente rápido y específico ligado a enzimas de reacción en cadena de la polimerasa para la detección de la toxina LT de Escherichia coli a partir de aislados clínicos. Arch Clin Infect Dis 12:e36261. doi: 10.5812/archcid.36261
Ewers, C., Göpel, L., Prenger-Berninghoff, E., Semmler, T., Kerner, K. y Bauerfeind, R. (2022). Aparición de genes de resistencia a colistina mcr-1 y mcr-2 en aislados de Escherichia coli porcina (2010-2020) y caracterización genómica de E. coli mcr-2-positiva. Frente. Microbiol. 13:1076315. doi: 10.3389/fmicb.2022.1076315
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Faccone, D., Moredo, F. A., Giacoboni, G. I., Albornoz, E., Alarcón, L., Nievas, V. F., et al. (2019). Escherichia coli multirresistente que alberga mcr-1 y blaCTX-M genes aislados de cerdos en Argentina. J. Glob. Antimicrobiano. Resistir. 18, 160–162. doi: 10.1016/j.jgar.2019.03.011
Fairbrother, J. M., Nadeau, É., y Gyles, C. L. (2005). Escherichia coli en la diarrea postdestete en cerdos: una actualización sobre los tipos bacterianos, la patogénesis y las estrategias de prevención. 6, págs. 17–39. doi: 10.1079/AHR2005105
Foong, W. E., Wilhelm, J., Tam, H.-K. y Pos, K. M. (2020). El eflujo de tigeciclina en Acinetobacter baumannii está mediado por TetA en sinergia con transportadores de eflujo de tipo RND. J. Quimioterapia antimicrobiana. 75, 1135–1139. doi: 10.1093/jac/dkaa015
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Francis, D. H. (2002). Infección por Escherichia coli enterotoxigénica en cerdos y su diagnóstico. J. Producto de salud porcina. 10, 171–175. Disponible en: https://www.aasv.org/shap/issues/v10n4/v10n4p171
Frydendahl, K. (2002). Prevalencia de serogrupos y genes de virulencia en Escherichia coli asociados con diarrea postdestete y enfermedad edema en cerdos y comparación de enfoques diagnósticos. Veterinario Microbiol. 85, 169–182. doi: 10.1016/S0378-1135(01)00504-1
Resumen de PubMed | Texto completo de CrossRef | Google Académico
García, V., Gambino, M., Pedersen, K., Haugegaard, S., Olsen, J. E. y Herrero-Fresno, A. (2020). Escherichia coli enterotoxigénica positiva para F4 y F18 aisladas de diarrea de cerdos postdestetados: caracterización genómica. Aplicación. Entorno. Microbiol. 86, E01913–E01920. doi: 10.1128/AEM.01913-20
García-Meniño, I., García, V., Alonso, M. P., Blanco, J. E., Blanco, J. y Mora, A. (2021). Clones de Escherichia coli enterotoxigénica y productora de toxina Shiga implicados en colibacilosis entérica porcina en España y tasas de resistencia a antibióticos. Veterinario Microbiol. 252:108924. doi: 10.1016/j.vetmic.2020.108924
Gresse, R., Chaucheyras-Durand, F., Fleury, M. A., Van de Wiele, T., Forano, E. y Blanquet-Diot, S. (2017). Disbiosis de la microbiota intestinal en lechones postdestetados: comprender las claves de la salud. Tendencias Microbiol. 25, 851–873. doi: 10.1016/j.tim.2017.05.004
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Guiral, E., Goncalves Quiles, M., Muñoz, L., Moreno-Morales, J., Alejo-Cancho, I., Salvador, P., et al. (2019). Aparición de resistencia a quinolonas y antibióticos β-lactámicos en Escherichia coli enteroagregativa y enterotoxigénica causante de diarrea del viajero. Antimicrobiano. Agentes Chemother. 63:01745-01718. DOI: 10.1128/AAC
Holt, J. G., Krieg, N. R., Sneath, P. H., Staley, J. T. y Williams, S. T. (1994). Manual de bacteriología determinada de Bergey. Williams y Wilkins. doi: 10.1001/jama.1958.02990080132031
Hopkins, K. L., Liebana, E., Villa, L., Batchelor, M., Threlfall, E. J. y Carattoli, A. (2006). Tipificación de replicón de plásmidos portadores de CTX-M o CMY β-lactamasas que circulan entre los aislados de Salmonella y Escherichia coli. Antimicrobiano. Agentes Chemother. 50, 3203–3206. doi: 10.1128/AAC.00149-06
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Isaacson, R. (1977). Antígeno de superficie K99 de Escherichia coli: purificación y caracterización parcial. Infectar. Inmune. 15, 272–279. DOI: 10.1128/IAI.15.1.272-279.1977
Isaacson, R. E., y Richter, P. (1981). Escherichia coli 987P pilus: purificación y caracterización parcial. J. Bacteriol. 146, 784–789. DOI: 10.1128/JB.146.2.784-789.1981
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Jackson, C., Davis, J., Frye, J., Barrett, J. y Hiott, L. (2015). Diversidad de plásmidos y genes de resistencia antimicrobiana en Escherichia coli multirresistente aislada de animales de compañía sanos. Zoonosis Salud Pública 62, 479–488. doi: 10.1111/zph.12178
Joffré, E., von Mentzer, A., Svennerholm, A. M. y Sjöling, Å. (2016). Identificación de nuevas variantes del alelo de enterotoxina termoestable (STa) producidas por Escherichia coli enterotoxigénica humana (ETEC). Int. J. Med. Microbiol. 306, 586–594. doi: 10.1016/j.ijmm.2016.05.016
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Johnson, T. J., y Nolan, L. K. (2009). Patogenia de los plásmidos de virulencia de Escherichia coli. Microbiol. Mol. Biol. Rev. 73, 750–774. doi: 10.1128/MMBR.00015-09
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Kopotsa, K., Osei Sekyere, J. y Mbelle, N. M. (2019). Evolución del plásmido en Enterobacteriaceae productoras de carbapenemasas: una revisión. Ann. N. Y. Acad. 1457, 61–91. doi: 10.1111/nyas.14223
Kwong, J. C., McCallum, N., Sintchenko, V. y Howden, B. P. (2015). Secuenciación del genoma completo en microbiología clínica y de salud pública. Patología 47, 199–210. doi: 10.1097/PAT.0000000000000235
Laird, T. J., Abraham, S., Jordan, D., Pluske, J. R., Hampson, D. J., Trott, D. J., et al. (2021). Escherichia coli enterotoxigénica porcina: resistencia antimicrobiana y desarrollo de estrategias de control alternativas basadas en microbios. Veterinario Microbiol. 258:109117. doi: 10.1016/j.vetmic.2021.109117
Lee, W., Kim, M.-H., Sung, S., Kim, E., An, E. S., Kim, S. H., et al. (2023). Caracterización basada en el genoma de cepas híbridas de Escherichia coli productoras de toxina Shiga y enterotoxigénicas (STEC / ETEC) aisladas en Corea del Sur, 2016-2020. Microorganismos 11:1285. doi: 10.3390/microorganismos11051285
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Leonard, S. R., Mammel, M. K., Rasko, D. A. y Lacher, D. W. (2016). La cepa 1 del linaje críptico 7v de Escherichia sp. productora de toxina Shiga híbrida y enterotoxigénica alberga un plásmido híbrido. Aplicación. Entorno. Microbiol. 82, 4309–4319. doi: 10.1128/AEM.01129-16
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Liu, Y.-Y., Wang, Y., Walsh, T. R., Yi, L.-X., Zhang, R., Spencer, J., et al. (2016). Aparición del mecanismo de resistencia a la colistina MCR-1 mediado por plásmidos en animales y seres humanos en China: un estudio microbiológico y biológico molecular. Lancet Infect. Dis. 16, 161–168. doi: 10.1016/S1473-3099(15)00424-7
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Luise, D., Lauridsen, C., Bosi, P. y Trevisi, P. (2019). Metodología y aplicación de modelos de infección por Escherichia coli F4 y F18 en cerdos post-destete. J. Anim. Sci. Biotechnol. 10, 1–20. DOI: 10.1186/S40104-019-0352-7
Luppi, A. (2017). Colibacilosis entérica porcina: diagnóstico, terapia y resistencia antimicrobiana. Gerente de Salud Porcina. 3, 1–18. DOI: 10.1186/S40813-017-0063-4
Melton-Celsa, A. R. (2014). Clasificación, estructura y función de la toxina Shiga (Stx). Microbiol. Espectro. 2:EHEC-0024-2013. DOI: 10.1128/microbiolspec. EHEC-0024-2013
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Nagy, B., Whipp, S., Imberechts, H., Bertschinger, H., Dean-Nystrom, E., Casey, T., et al. (1997). Relación biológica entre las fimbrias F18ab y F18ac de Escherichia colienterotoxigénica y verotoxigénica de cerdos destetados con enfermedad edema o diarrea. Microbio. Pathog. 22, 1–11. DOI: 10.1006/mpat.1996.0085
Nguyet, L. T. Y., Keeratikunakorn, K., Kaeoket, K. y Ngamwongsatit, N. (2022). Escherichia coli resistente a los antibióticos de lechones diarreicos de granjas porcinas en Tailandia que albergan genes MCR resistentes a la colistina. Rep. 12:9083. DOI: 10.1038/S41598-022-13192-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ning, W., Wei, Y., Gao, L., Han, C., Gou, Y., Fu, S., et al. (2022). HemI 2.0: un servicio en línea para la ilustración de mapas de calor. Ácidos nucleicos res. 50, W405–W411. doi: 10.1093/nar/gkac480
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ntakiyisumba, E., Lee, S. y Won, G. (2022). Enfoques basados en la evidencia para determinar antígenos diana efectivos para desarrollar vacunas contra la diarrea post-destete causada por Escherichia coli enterotoxigénica en cerdos: una revisión sistemática y metanálisis en red. Animales 12:2136. DOI: 10.3390/ani12162136
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Peng, Z., Hu, Z., Li, Z., Zhang, X., Jia, C., Li, T., et al. (2022). Resistencia a los antimicrobianos y genómica poblacional de Escherichia coli multirresistente en granjas porcinas en China continental. Nat. Commun. 13:1116. doi: 10.1038/s41467-022-28750-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ren, J., Zhang, P., Yin, H., Huang, H., Yang, D., Zeng, H., et al. (2020). Identificación y características biológicas de aislados de ETEC en granjas porcinas a gran escala en la provincia de Anhui. Anim. Esposo. Vet. Med. 52, 87–91. doi: CNKI:SUN:XMYS.0.2020-05-015
Renzhammer, R., Loncaric, I., Roch, F.-F., Pinior, B., Käsbohrer, A., Spergser, J., et al. (2020). Prevalencia de genes de virulencia y resistencias antimicrobianas en E. coli asociadas con diarrea neonatal, diarrea postdestete y enfermedad de edema en cerdos de Austria. Antibióticos 9:208. doi: 10.3390/antibióticos9040208
Rhouma, M., Fairbrother, J. M., Beaudry, F. y Letellier, A. (2017). Diarrea post destete en cerdos: factores de riesgo y estrategias de control no basadas en colistina. Acta Vet. 59, 1–19. DOI: 10.1186/S13028-017-0299-7
Shen, C., Zhong, L.-L., Yang, Y., Doi, Y., Paterson, D. L., Stoesser, N., et al. (2020). Dinámica de la prevalencia de mcr-1 y Escherichia coli mcr-1 positiva después del cese del uso de colistina como aditivo para piensos para animales en China: un estudio epidemiológico molecular prospectivo transversal y basado en la secuenciación del genoma completo. Microbio de Lancet 1, e34–e43. doi: 10.1016/S2666-5247(20)30005-7
Smith, H. W., y Linggood, M. A. (1971). Observaciones sobre las propiedades patogénicas de los plásmidos K88, Hly y Ent de Escherichia coli con especial referencia a la diarrea porcina. J. Med. Microbiol. 4, 467–485. doi: 10.1099/00222615-4-4-467
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Sun, C., Cui, M., Zhang, S., Liu, D., Fu, B., Li, Z., et al. (2020). La epidemiología genómica de Escherichia coli resistente a la tigeciclina de origen animal en China revela el reciente gen tet (X4) codificado por plásmidos endémicos recientes. Commun. Biol. 3:412. doi: 10.1038/s42003-020-01148-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Sun, C., Cui, M., Zhang, S., Wang, H., Song, L., Zhang, C., et al. (2019). Gen tet(X4) resistente a la tigeciclina mediado por plásmidos en Escherichia coli de animales productores de alimentos, China, 2008-2018. Emerg. Microbio. Infecta. 8, 1524–1527. doi: 10.1080/22221751.2019.1678367
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Sun, Y., y Kim, S. W. (2017). Desafío intestinal con Escherichia coli enterotoxigénica en cerdos, e intervención nutricional para prevenir la diarrea postdestete. Anim. Nutr. 3, 322–330. doi: 10.1016/j.aninu.2017.10.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Tseng, M., Fratamico, P. M., Bagi, L., Delannoy, S., Fach, P., Manning, S. D., et al. (2014). Contenido diverso del gen de virulencia de Escherichia coli productora de toxina Shiga de cerdos de terminación. Aplicación. Entorno. Microbiol. 80, 6395–6402. doi: 10.1128/AEM.01761-14
Turner, S. M., Chaudhuri, R. R., Jiang, Z.-D., DuPont, H., Gyles, C., Penn, C. W., et al. (2006). Las comparaciones filogenéticas revelan múltiples adquisiciones de los genes de la toxina por cepas enterotoxigénicas de Escherichia coli de diferentes linajes evolutivos. J. Clin. Microbiol. 44, 4528–4536. doi: 10.1128/JCM.01474-06
Resumen de PubMed | Texto completo de CrossRef | Google Académico
von Mentzer, A., Connor, T. R., Wieler, L. H., Semmler, T., Iguchi, A., Thomson, N. R., et al. (2014). Identificación de clados enterotoxigénicos de Escherichia coli (ETEC) con distribución global a largo plazo. Nat. Genet. 46, 1321–1326. doi: 10.1038/ng.3145
Wagner, S., Gally, D. L. y Argyle, S. A. (2014). La Escherichia coli multirresistente de infecciones del tracto urinario canino tiende a tener filotipos comensales, menor prevalencia de determinantes de virulencia y replicones ampC. Veterinario Microbiol. 169, 171–178. doi: 10.1016/j.vetmic.2014.01.003
Wang, Q., Lei, C., Cheng, H., Yang, X., Huang, Z., Chen, X., et al. (2022). Diseminación generalizada del gen de resistencia a tigeciclina mediado por plásmidos tet(X4) en Enterobacterales de origen porcino. Microbiol. Espectro. 10, E01615–E01622. doi: 10.1128/spectrum.01615-22
Wang, Y., Xu, C., Zhang, R., Chen, Y., Shen, Y., Hu, F., et al. (2020). Cambios en la resistencia a la colistina y la abundancia de mcr-1 en Escherichia coli de origen animal y humano tras la prohibición de los aditivos positivos para colistina en China: un estudio comparativo epidemiológico. Lancet Infect. Dis. 20, 1161–1171. doi: 10.1016/S1473-3099(20)30149-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Yang, H., Paruch, L., Chen, X., Van Eerde, A., Skomedal, H., Wang, Y., et al. (2019). Aplicación y resistencia a antibióticos en la producción porcina en China: situación actual y perspectivas futuras. Frente. Vet. Sci. 6:136. doi: 10.3389/fvets.2019.00136
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Yang, D., Zhang, P., Yu, P., Ren, J., Li, D., Wang, J., et al. (2020). Aislamiento, identificación y evaluación de la vacuna inactivada de Escherichia coli enterotoxigénica (ETEC) en granjas porcinas a gran escala en el norte de Jiangsu. Acta Microbiol Sin. 60, 261–272. doi: 10.13343/j.cnki.wsxb.20190140
Zhang, J., Chen, L., Wang, J., Yassin, A. K., Butaye, P., Kelly, P., et al. (2018). Detección molecular de genes de resistencia a la colistina (mcr-1, mcr-2 y mcr-3) en hisopos nasales/orofaríngeos y anales/cloacales de cerdos y aves de corral. 8:3705. DOI: 10.1038/S41598-018-22084-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zhang, W., Robertson, D. C., Zhang, C., Bai, W., Zhao, M. y Francis, D. H. (2008). Las construcciones de Escherichia coli que expresan enterotoxinas humanas o porcinas inducen enfermedades diarreicas idénticas en un modelo de infección de lechones. Aplicación. Entorno. Microbiol. 74, 5832–5837. doi: 10.1128/AEM.00893-08
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zhang, S., Wen, J., Wang, Y., Wang, M., Jia, R., Chen, S., et al. (2022). Diseminación y prevalencia del gen de resistencia a tigeciclina de alto nivel mediado por plásmidos tet(X4). Frente. Microbiol. 13:969769. doi: 10.3389/fmicb.2022.969769
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zhang, W., Zhao, M., Ruesch, L., Omot, A. y Francis, D. (2007). Prevalencia de genes de virulencia en cepas de Escherichia coli recientemente aisladas de cerdos jóvenes con diarrea en los Estados Unidos. Veterinario Microbiol. 123, 145–152. doi: 10.1016/j.vetmic.2007.02.018
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zhou, Z., Alikhan, N.-F., Sargento, M. J., Luhmann, N., Vaz, C., Francisco, A. P., et al. (2018). Árbol de uva: visualización de las relaciones genómicas centrales entre 100, 000 patógenos bacterianos. Genoma Res. 28, 1395–1404. doi: 10.1101/gr.232397.117
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: Escherichia coli enterotoxigénica, secuenciación del genoma completo, resistencia antimicrobiana, caracterización genómica, factor de virulencia, cerdo
Cita: Hu J, Li J, Huang X, Xia J, Cui M, Huang Y, Wen Y, Xie Y, Zhao Q, Cao S, Zou L y Han X (2023) Rasgos genómicos de aislados enterotoxigénicos de Escherichia coli resistentes a múltiples fármacos de cerdos diarreicos. Frente. Microbiol. 14:1244026. doi: 10.3389/fmicb.2023.1244026
Recibido: 21 de junio de 2023; Aprobado: 17 de julio de 2023;
Publicado: 03 agosto 2023.
Editado por:
Gururaja Perumal Pazhani, Instituto SRM de Ciencia y Tecnología, India
Revisado por:
Nuvee Prapasarakul, Universidad de Chulalongkorn, Tailandia
Jérôme Ambroise, Université Catholique de Louvain, Bélgica
Derechos de autor © 2023 Hu, Li, Huang, Xia, Cui, Huang, Wen, Xie, Zhao, Cao, Zou y Han. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Likou Zou, zoulikou@sicau.edu.cn; Xinfeng Han, hanxinf@163.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía