Una PCR digital de triple cristal para la detección del virus de la peste porcina africana de genotipos I y II
Una PCR digital de triple cristal para la detección del virus de la peste porcina africana de genotipos I y II
 Kaichuang Shi1,2,3*†
Kaichuang Shi1,2,3*†  Xinxiu Qian2†
Xinxiu Qian2†  Yuwen Shi2
Yuwen Shi2  Haina Wei3
Haina Wei3  Yi Pan1
Yi Pan1  Feng Long3
Feng Long3  Qingan Zhou3
Qingan Zhou3  Shenglan Mo3
Shenglan Mo3  Liping Hu3
Liping Hu3  Zongqiang Li2*
Zongqiang Li2*- 1Facultad de Ciencias Médicas Básicas, Universidad Médica de Youjiang para las Nacionalidades, Baise, China
- 2Facultad de Ciencia y Tecnología Animal, Universidad de Guangxi, Nanning, China
- 3Centro de Guangxi para el Control y la Prevención de Enfermedades Animales, Nanning, China
La peste porcina africana (PPA) es una enfermedad viral altamente contagiosa y letal que causa fiebre hemorrágica grave en los cerdos. Sigue extendiéndose por todo el mundo, lo que supone un grave riesgo socioeconómico y pone en peligro la biodiversidad y la seguridad alimentaria nacional. La PPA brotó por primera vez en China en 2018 y se ha extendido a la mayoría de las provincias de todo el país. Los genotipos I y II del virus de la PPA (PPA) como patógenos etiológicos se han encontrado en China. En este estudio, se diseñaron tres pares de cebadores y sondas específicos dirigidos al gen B646L, el gen F1055L y el gen E183L del virus de la PPA para detectar cepas universales, de genotipo I y de genotipo II, respectivamente. Se estableció una PCR digital de cristal triple (cdPCR) sobre la base de la optimización de diversas condiciones de reacción. El ensayo demostró una sensibilidad notable con límites bajos de detección (LOD) de 5,120, 4,218 y 4,588 copias/reacción para los genes B646L, F1055L y E183L, respectivamente; excelente repetibilidad con coeficientes de variación (CV) intraensayo del 1,24 al 2,01 % y CV entre ensayos del 1,32 al 2,53 %; buena especificidad para la detección solo de los genotipos I y II de PPA, sin reactividad cruzada con PCV2, PRV, SIV, PRRSV, PEDV, FMDV y CSFV. La cdPCR triplex se utilizó para analizar 1.275 muestras clínicas de la provincia china de Guangxi, y las tasas de positividad fueron de 5,05, 3,22 y 1,02% para los genotipos I, II y coinfección de los genotipos I y II, respectivamente. Estas 1.275 muestras clínicas también se detectaron mediante una PCR cuantitativa en tiempo real (qPCR) triplex de referencia reportada, y la concordancia de los resultados de detección entre estos dos métodos fue superior al 98,98%. En conclusión, la cdPCR triplex desarrollada podría utilizarse como un método rápido, sensible y preciso para detectar y diferenciar las cepas de los genotipos I y II del virus de la PPA.
1 Introducción
La peste porcina africana (PPA) es una enfermedad infecciosa altamente contagiosa y letal de los cerdos. El virus de la PPA (PPA), el patógeno etiológico, pertenece al género Asfivirus de la familia Asfiviridae. El virus de la PPA es un virus de ADN lineal de doble cadena, con un genoma de 170 a 193 kb de longitud y contiene entre 151 y 167 marcos de lectura abiertos (ORF) que codifican más de 168 proteínas estructurales y no estructurales (1). La PPA es una enfermedad aguda, altamente infecciosa y mortal para los cerdos domésticos y los jabalíes, con una tasa de mortalidad de hasta el 100% (2). Los síntomas típicos y los cambios patológicos de la PPA se caracterizan por fiebre hemorrágica mortal, dificultad respiratoria, cianosis cutánea, trombocitopenia y sangrado extenso de los riñones, la mucosa gastrointestinal, los ganglios linfáticos y otros órganos (3, 4). La PPA se descubrió por primera vez en Kenia (África) en 1921 y, desde entonces, la PPA se ha diseminado en varios países del África subsahariana y se ha extendido a otros continentes fuera de África (5, 6). El primer brote de PPA en China se descubrió en 2018 en la provincia de Jilin, en el noreste de China, y luego se extendió rápidamente a casi todas las provincias del país en el plazo de un año, lo que provocó un golpe devastador para la industria porcina de China (7-9). Desde 2018, esta enfermedad altamente contagiosa se ha propagado rápidamente a muchos países del sudeste asiático y América (5, 10–12). Hasta la fecha, la PPA se ha descubierto en muchos países, principalmente en África, Asia, Europa y el Caribe americano (13, 14), y ha dañado seriamente la industria porcina en todo el mundo. La historia de la prevención y el control de la PPA en varios países muestra que, ante la ausencia de una vacuna específica y de medidas eficaces, la epidemia de PPA, una vez introducida, es muy difícil de erradicar en poco tiempo (15). En cuanto a China, la PPA es actualmente la enfermedad de máxima prioridad para la prevención y el control.
El virus de la peste porcina africana se puede clasificar en 24 genotipos sobre la base de la secuencia final 3′ del gen B646L del virus de la peste porcina africana (16, 17). Fuera de África, solo se han descubierto cepas de los genotipos I y II del virus de la PPA (6, 17, 18). En China, el virus de la PPA genotipo II se identificó por primera vez en 2018 y ha sido la cepa predominante que ha circulado en el campo a partir de entonces (19, 20). El virus de la peste porcina africana de genotipo I se descubrió por primera vez en 2020 y se ha confirmado en las provincias de Henan, Shandong y Guangxi en China (19, 21–23), pero su prevalencia y daño en todo el país requieren más investigación y evaluación. Recientemente, se descubrió en China la cepa naturalmente recombinante de los genotipos I y II, que demostró ser altamente virulenta y letal para los cerdos domésticos (24). Hoy en día, las cepas recombinantes de genotipo I, genotipo II y genotipo I y II del virus de la PPA prevalecen simultáneamente en China, y se han reportado coinfecciones de genotipo I y II (19, 23). Estas situaciones aumentan la complejidad de las cepas epidémicas y aumentan la dificultad de prevención y control, lo que inflige un daño significativo a la industria porcina de China (8, 9, 25). Por lo tanto, la detección e identificación rápida y precisa del genotipo circulante del virus de la PPA es de suma importancia para implementar medidas efectivas de prevención y control en la etapa temprana de la infección.
La PCR cuantitativa en tiempo real (qPCR) se ha utilizado ampliamente para detectar ácidos nucleicos virales en muchos laboratorios. Esta técnica molecular avanzada ha obtenido un reconocimiento y aceptación significativos debido a sus ventajas de baja probabilidad de contaminación, excelente precisión, sensibilidad superior, especificidad excepcional, conveniencia y eficiencia (26, 27). Sin embargo, las desventajas de la qPCR incluyen principalmente la fluctuación de los valores de Ct en función de la configuración del umbral, la alta sensibilidad a los inhibidores de la reacción y el complejo procedimiento de generación de curvas de calibración, lo que limita la aplicación de la qPCR. Por lo tanto, la PCR digital (dPCR) es una nueva y mejor opción para la detección de copias bajas de ácidos nucleicos virales. La dPCR es una tecnología emergente en el campo de la microbiología, y tiene como principales ventajas una alta especificidad y sensibilidad, una excelente repetibilidad, la capacidad de lograr una cuantificación absoluta sin necesidad de un gen de referencia, el valor de Ct y la curva estándar, y una fuerte tolerancia a los inhibidores de la PCR (28, 29). La dPCR se puede dividir en PCR digital de cristal (cdPCR) y PCR digital de gotas (ddPCR) (30, 31). Varios informes han establecido la dPCR para detectar el virus de la PPA (32-36), pero nunca se ha informado de una dPCR múltiple para detectar simultáneamente el virus de la PPA de genotipo I y genotipo II. Aquí, se desarrolló una cdPCR triplex para detectar y diferenciar el virus de la PPA genotipo I y el genotipo II, y se utilizó para analizar 1.275 muestras clínicas para validar su aplicabilidad en el campo.
2 Materiales y métodos
2.1 Recogida de muestras clínicas
De marzo de 2023 a agosto de 2023, se obtuvieron un total de 1.275 muestras clínicas (incluidos pulmones, bazo, riñón, amígdalas y ganglios linfáticos de cada cerdo) de 1.275 cerdos muertos de diferentes 4 granjas porcinas, 6 plantas de tratamiento inocuas y 17 mataderos en la provincia de Guangxi, en el sur de China. Los cerdos muertos mostraron diferentes manifestaciones como fiebre, diarrea, tos y/o enrojecimiento, cianosis o sangrado en la piel. Se obtuvo el consentimiento por escrito de los propietarios de los animales, y el grupo de investigación se comprometió a no divulgar la información detallada sobre las granjas porcinas relevantes y la incidencia de enfermedades en las piaras de cerdos en este estudio. Estos tejidos se transportaron a ≤4 °C en 12 h al laboratorio y se almacenaron a -80 °C hasta su uso.
2.2 Obtención de cepas virales
El virus de la fiebre aftosa (virus de la fiebre aftosa, cepa O/Mya98/XJ/2010), el virus de la influenza porcina (VIS, cepa TJ), el virus del síndrome respiratorio y reproductivo porcino (PRRSV, cepa JXA1-R), el virus de la peste porcina clásica (PPC, cepa C), el virus diarreico epidémico porcino (PEDV, cepa CV777), el virus de la pseudorrabia (PRV, cepa Bartha-K61) y el circovirus porcino tipo 2 (PCV2, cepa ZJ/C) se obtuvieron de una empresa comercial (Huapai Co., Ltd., Chengdu, China). Las muestras clínicas de los genotipos I y II del virus de la PPA se obtuvieron del Centro para el Control y la Prevención de Enfermedades Animales (CADC) de Guangxi, China.
2.3 Diseño de cebadores y sondas
Se diseñaron tres conjuntos de cebadores y sondas específicos, tal como lo describieron Qian et al. en el informe anterior (23). Los cebadores y la sonda dirigidos al gen B646L se utilizaron para detectar los 24 genotipos diferentes del virus de la PPA, los dirigidos al gen F1055L se utilizaron para detectar específicamente el genotipo I del virus de la PPA y los dirigidos al gen E183L se utilizaron para detectar específicamente el genotipo II del virus de la PPA. Los cebadores y las sondas se muestran en la Tabla 1.
2.4 Extracción de ácidos nucleicos
Los tejidos clínicos fueron homogeneizados, liofilizados-descongelados, vortexados y centrifugados como lo describen Qian et al. (23). Los ácidos nucleicos totales se extrajeron de sobrenadantes de 200 μL utilizando el extractor automatizado de ácidos nucleicos GeneRotex 96 y el kit de aislamiento de ADN/ARN viral 4.0 (TIANLONG, Xian, China) de acuerdo con las construcciones del fabricante, y se almacenaron a -80 °C hasta su uso.
2.5 Generación de las construcciones plásmidas estándar
Las construcciones plásmidas estándar se generaron de acuerdo con Qian et al. (23) con modificaciones menores. Los ácidos nucleicos totales de las muestras positivas para el virus de la peste porcina africana se utilizaron como plantillas para amplificar los fragmentos diana mediante PCR utilizando los cebadores diseñados (Tabla 1). Los productos de amplificación se purificaron utilizando el kit de purificación de fragmentos de ADN MiniBEST Ver.4.0 (TaKaRa, Dalian, China), se clonaron en el vector pMD18-T (TaKaRa, Dalian, China) y luego se transformaron en células de E. coli DH5α (TaKaRa, Dalian, China). Los clones positivos se cultivaron a 37 °C durante 22-24 h, y las construcciones de plásmidos estándar recombinantes se extrajeron utilizando el kit de extracción de plásmidos MiniBEST Ver.5.0 (TaKaRa, Dalian, China). Los plásmidos se enviaron a IGE biotechnology LTD (Guangzhou, China) para su secuenciación utilizando el método de secuenciación Sanger, y las secuencias de los fragmentos insertados se confirmaron mediante el análisis BLAST en el Centro Nacional de Información Biotecnológica (NCBI).1 A continuación, las construcciones plásmidas correctas se denominaron p-dASFV-B646L, p-dASFV-F1055L y p-dASFV-E183L, respectivamente, y se utilizaron para establecer la cdPCR triplex.
Las construcciones plásmidas estándar se midieron utilizando el espectrofotómetro NanoDrop (Thermo Fisher, Waltham, MA, EE.UU.) a 260 nm y 280 nm, y determinaron sus concentraciones utilizando la siguiente fórmula: número de copias (copias/μL) = (concentración de constructo plásmido × 10−9 × 6.02 × 1023)/ (660 Dalton/bases × longitud del ADN).
2.6 Determinación de las condiciones de reacción
El sistema de cristal de zafiro Naica™ (Stilla Technologies™, Villejuif, Francia) se utilizó para optimizar varios parámetros de la cdPCR, es decir, la temperatura de recocido, los ciclos de reacción y las concentraciones de cebador y sonda. El sistema de reacción de cdPCR triplex se fijó en 25 μL. Estas condiciones se optimizaron para maximizar la precisión y fiabilidad de la reacción para una detección precisa y sensible de los analitos objetivo. Los procesos de la cdPCR triplex, incluida la preparación de los chips de zafiro, la amplificación de partición y PCR, la adquisición de imágenes de fluorescencia a tres colores y el análisis de los cristales de gotas, se realizaron de acuerdo con el manual de operación proporcionado por el fabricante (Stilla Technologies™, Villejuif, Francia).
2.7 Generación de las curvas estándar
Para generar las curvas estándar, se mezclaron tres construcciones de plásmidos estándar y se diluyeron en serie 10 veces, y las mezclas con concentraciones de reacción final que oscilaron entre 1,0 × 104 a 1.0 × 100 copias/μL se utilizaron como plantillas.
2.8 Evaluación de la especificidad
Para evaluar la especificidad se utilizaron los ácidos nucleicos totales de los siguientes virus: genotipos I y II de PPA, PCV2, PRV, PRRSV, PEDV, FMDV, CSFV y VIS. El agua destilada libre de nucleasa y las muestras de tejido negativas se utilizaron como controles negativos.
2.9 Evaluación de la sensibilidad
Las mezclas de tres construcciones plásmidas se diluyeron en serie 10 veces. Las mezclas con concentraciones de reacción final que oscilan entre 1,0 × 105 a 1.0 × 10−2 Se utilizaron copias/μL como plantillas, y los límites de detección (LOD) se determinaron mediante análisis de distribución de Poisson.
Además, se utilizaron como plantillas las mezclas con concentraciones de reacción final que oscilaron entre 250 y 0,25 copias/reacción, y los LOD se analizaron mediante regresión PROBIT en el software SPSS 26.0.2 y las cifras relacionadas se generaron utilizando el software Statacorp stata 17.3
2.10 Evaluación de la repetibilidad
Para evaluar la repetibilidad, se utilizaron las mezclas de tres construcciones plásmidas a concentraciones finales de 1,0 × 104, 1.0 × 103y 1,0 × 102 se utilizaron copias/μL para realizar las pruebas intraensayo e interensayo, y se calcularon los coeficientes de variación (CV).
2.11 Evaluación de las muestras clínicas
La cdPCR triplex desarrollada y la qPCR triplex reportada por Qian et al. (23) se utilizaron para analizar 1.275 muestras clínicas obtenidas en la provincia china de Guangxi. Se evaluó la sensibilidad clínica y la especificidad de la cdPCR triplex y se determinaron las tasas de concordancia de los resultados de detección entre ambos métodos mediante el programa SPSS 26.0 (ver nota 2).
3 Resultados
3.1 Generación de plásmidos patrón
Los fragmentos diana de los genes B646L, F1055L y E183L del virus de la PPA se obtuvieron mediante amplificación por PCR utilizando los cebadores de la Tabla 1, seguida de purificación y ligadura en el vector pMD18-T, y luego se transformaron en células competentes para E. coli DH5α. Se cultivaron los clones positivos y se extrajeron las construcciones plásmidas. Finalmente, se determinó que las concentraciones de tres construcciones plásmidas estándar denominadas p-dASFV-B646L, p-dASFV-F1055L y p-dASFV-E183L fueron de 3,69 × 1010, 1,81 × 1010y 1,0 × 1010 copias/μL, respectivamente. Todas las construcciones plásmidas se diluyeron a 1,0 × 1010 copias/μL, y almacenado a -80 °C hasta su uso.
3.2 Determinación de las condiciones de reacción
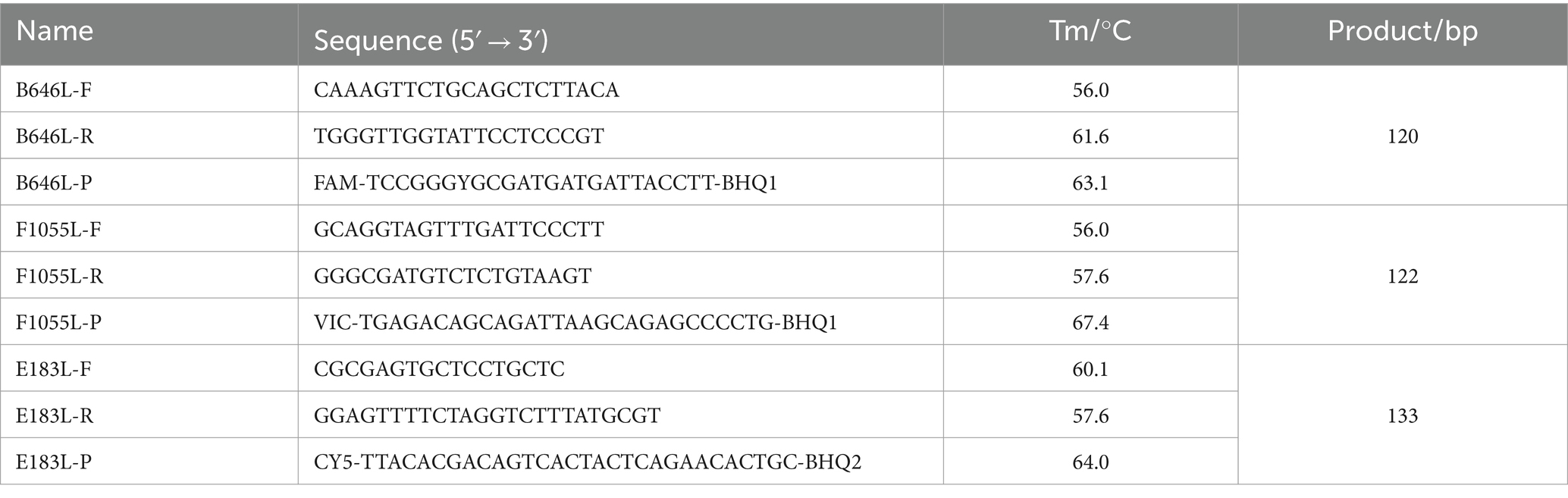
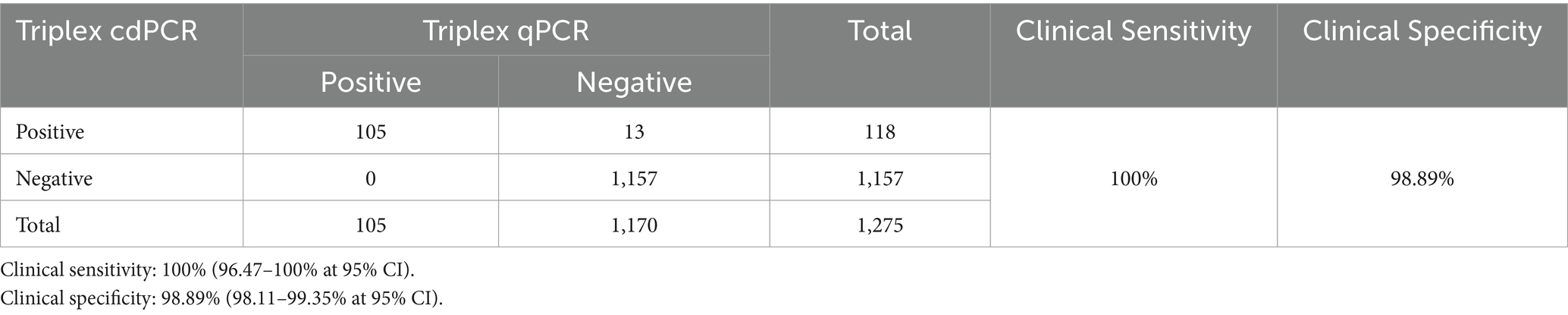
La cdPCR triplex se desarrolló utilizando el sistema de cristal de zafiro Naica™ (Stilla Technologies™, Villejuif, Francia). Después de experimentos para optimizar combinaciones de cebadores y sondas a diferentes concentraciones, temperaturas de recocido y ciclos de reacción, se obtuvieron las condiciones óptimas de reacción y se estableció una cdPCR triplex (Figura 1). El sistema de reacción contenía PerfeCTa Multiplex qPCR ToughMix (Quanta Biosciences, Gaithersburg, MD, EE. UU.), sal sódica fluoresceína (1 μM) (Apexbio Biotechnology, Beijing, China), tres cebadores y sondas, las mezclas de tres construcciones de plásmidos y agua libre de nucleasas (Tabla 2). El procedimiento de amplificación: 95 °C durante 30 s, 45 ciclos de 95 °C durante 5 s y 56 °C durante 30 s. Después de la amplificación, los chips Sapphire (Stilla Technologies, Francia) se trasladaron al Naica™ Prism3 (Stilla Technologies, Francia), y la concentración absoluta de cada muestra se informó automáticamente con 3 imágenes de alta resolución.
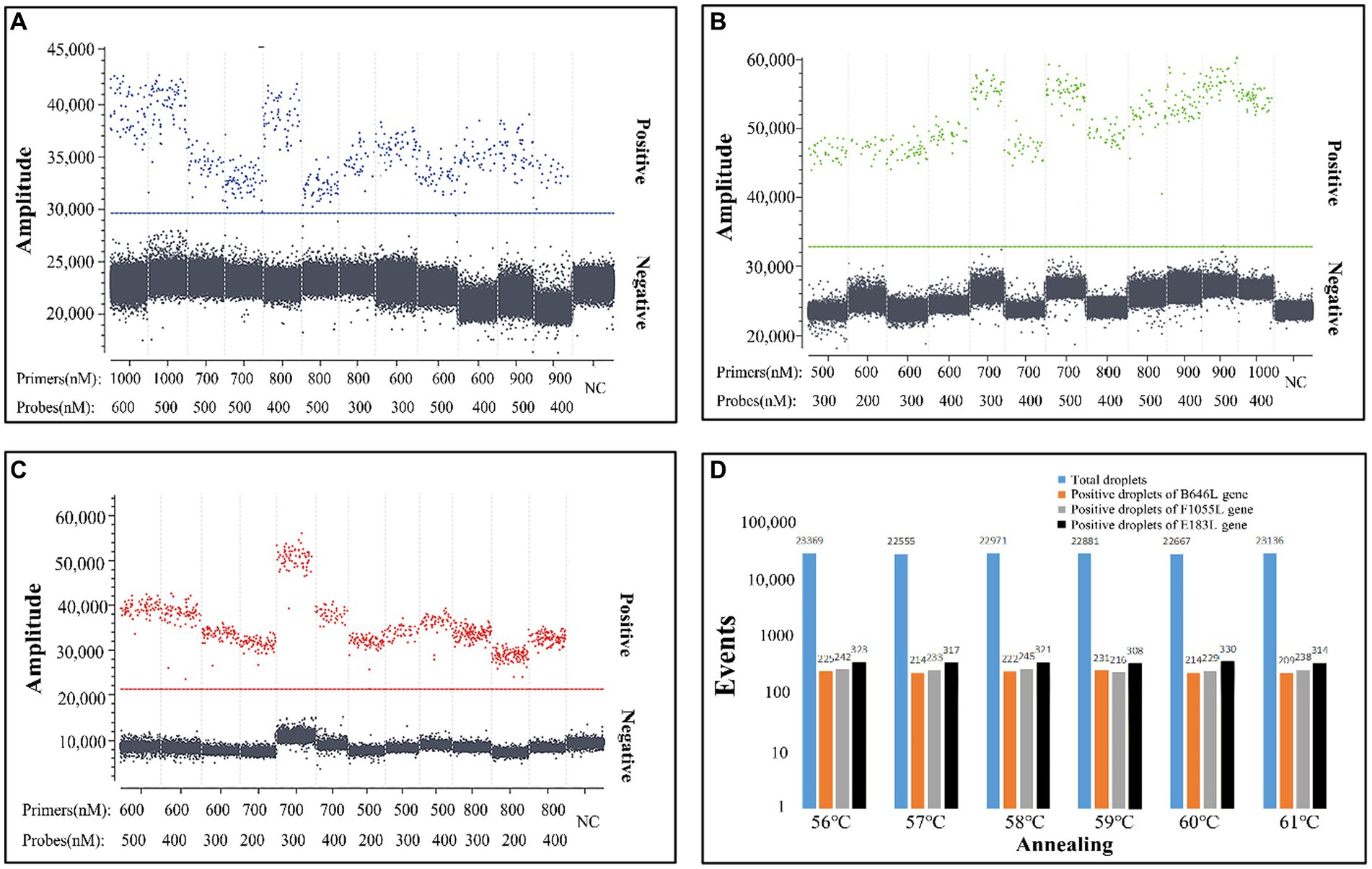 Figura 1. Optimización de las concentraciones de cebadores y sondas (A-C) y de la temperatura de recocido (D). Las tres construcciones plásmidas p-dASFV-B646L, p-dASFV-F1055L y p-dASFV-E183L se mezclaron en las concentraciones finales de 1.0 × 102 copias/μL. Los resultados de amplificación de 12 combinaciones diferentes de concentraciones de sonda y cebador se muestran en el panel (A-C), y los resultados de amplificación a diferentes temperaturas de recocido se muestran en el panel (D). NC, Control negativo.
Figura 1. Optimización de las concentraciones de cebadores y sondas (A-C) y de la temperatura de recocido (D). Las tres construcciones plásmidas p-dASFV-B646L, p-dASFV-F1055L y p-dASFV-E183L se mezclaron en las concentraciones finales de 1.0 × 102 copias/μL. Los resultados de amplificación de 12 combinaciones diferentes de concentraciones de sonda y cebador se muestran en el panel (A-C), y los resultados de amplificación a diferentes temperaturas de recocido se muestran en el panel (D). NC, Control negativo.
3.3 Generación de las curvas estándar
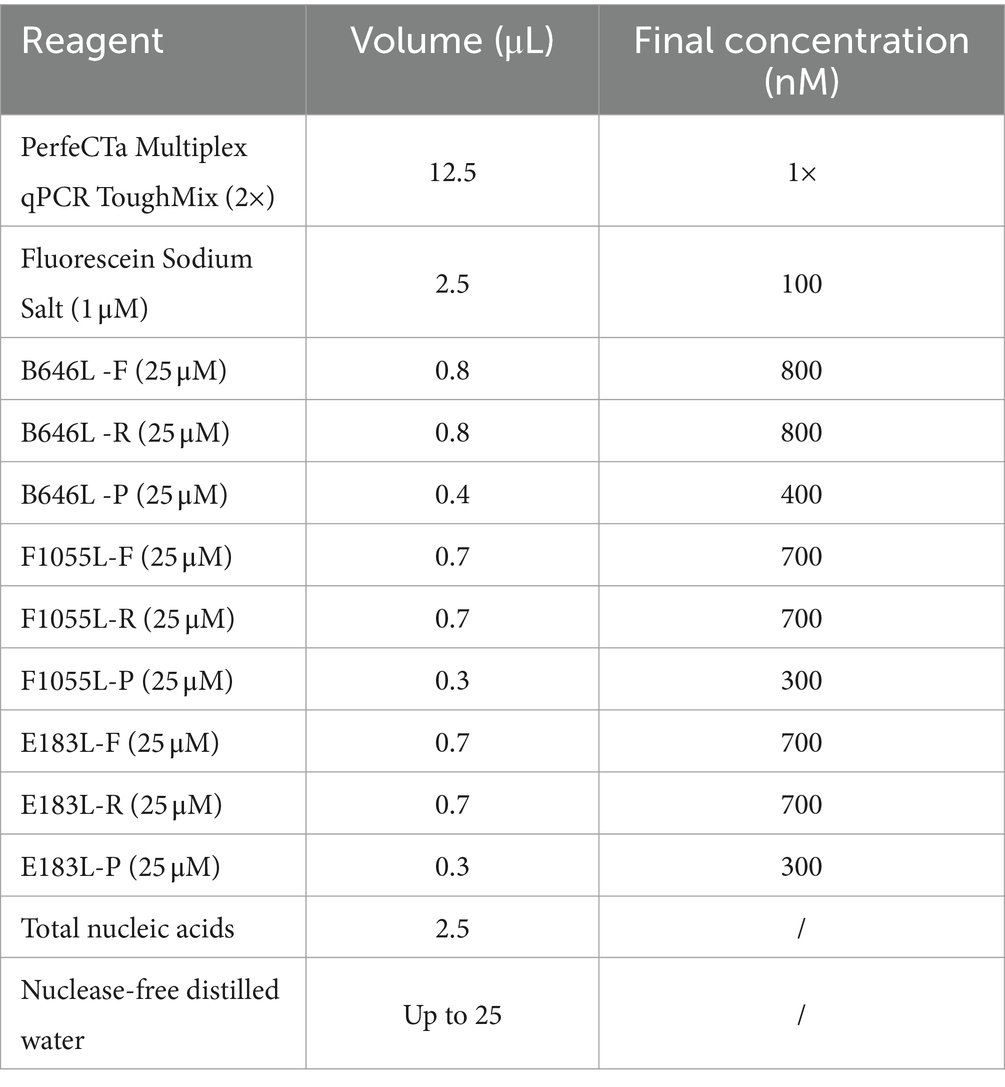
Para obtener las curvas estándar de la cdPCR triplex, el plásmido estándar construye p-dASFV-B646L, p-dASFV-F1055L y p-dASFV-E183L en concentraciones finales de 1.0 × 104 a 1.0 × 100 Se utilizaron copias/μL como plantillas. Los resultados mostraron que las pendientes y R2 fueron 0,982 y 0,9996, 0,9 y 0,9972, y 0,931 y 0,9973, para los genes B646L, F1055L y E183L, respectivamente (Figura 2).
 Figura 2. Las gotas positivas del plásmido construyen p-dASFV-B646L (A), p-dASFV- F1055L (B) y p-dASFV- E183L (C) a diferentes concentraciones finales de 1.0 × 104 a 1.0 × 100 copias/μL y las curvas estándar (D).
Figura 2. Las gotas positivas del plásmido construyen p-dASFV-B646L (A), p-dASFV- F1055L (B) y p-dASFV- E183L (C) a diferentes concentraciones finales de 1.0 × 104 a 1.0 × 100 copias/μL y las curvas estándar (D).
3.4 Análisis de especificidad
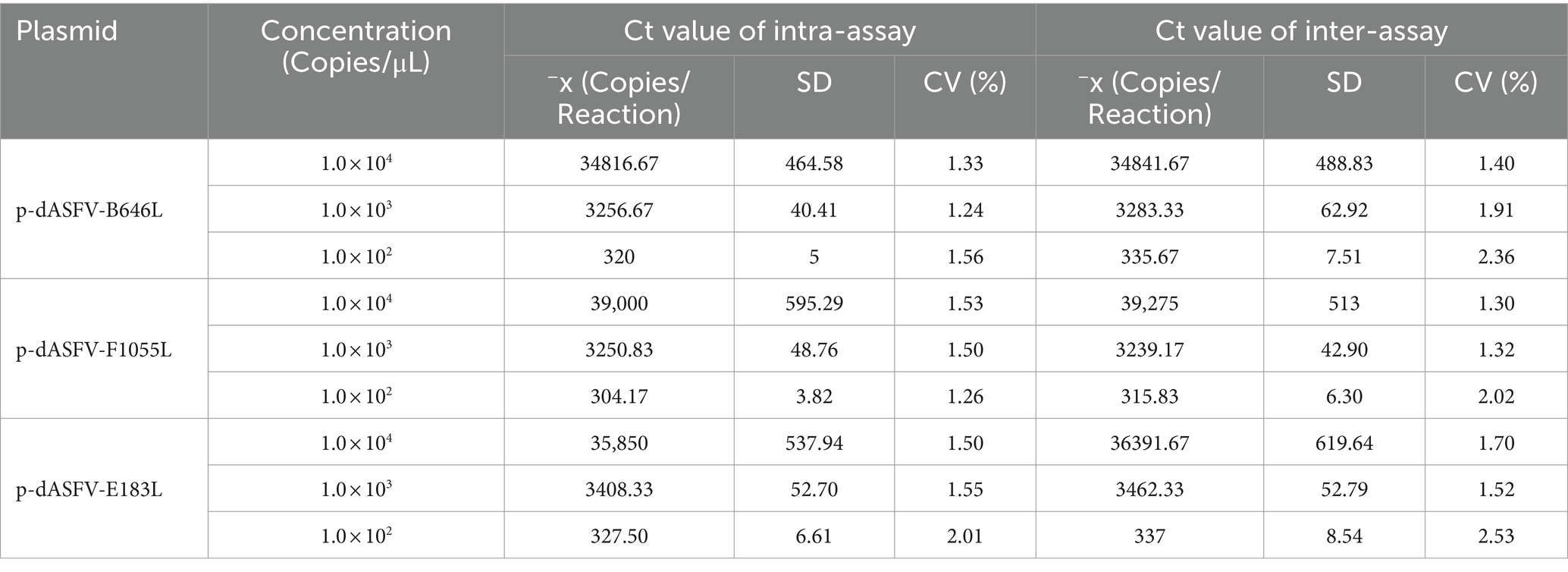
Para analizar la especificidad de la cdPCR se utilizaron los ácidos nucleicos totales del virus de la peste porcina africana genotipo I, el virus de la peste porcina africana genotipo II, el PCV2, el PRV, EL PRRSV, EL PEDV, EL FMDV, EL CSFV y el VIS. Los resultados mostraron que las gotitas positivas solo podían obtenerse de los genotipos I y II del virus de la PPA, pero no de los otros virus porcinos (Figura 3).
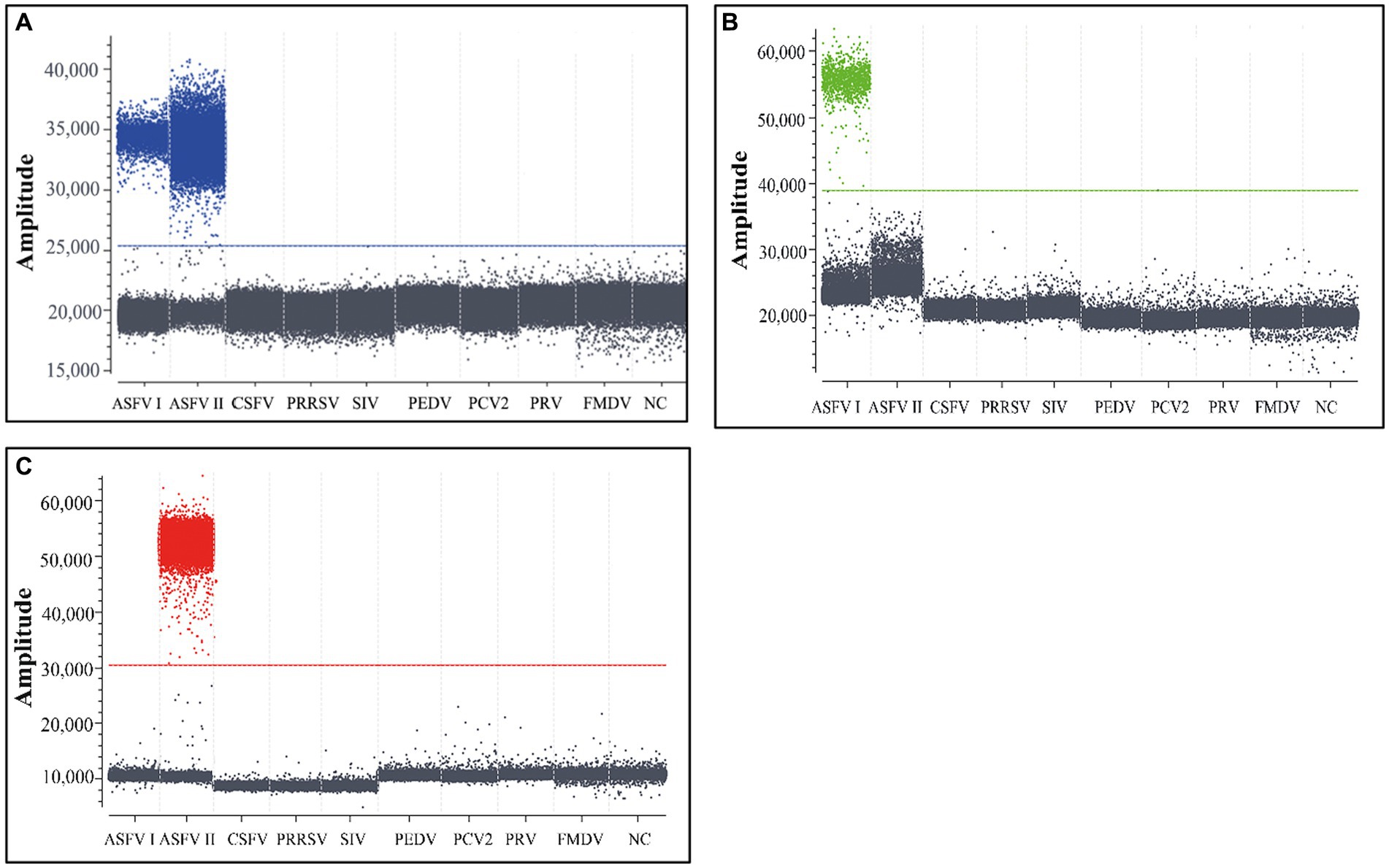 Figura 3. El análisis de especificidad de la cdPCR triplex (A-C). Se muestran los resultados para el gen B646L (A), el gen F1055L (B) y el gen E183L (C), respectivamente. Se utilizaron el virus de la peste porcina africana genotipo I, el virus de la peste porcina africana genotipo II, la PCV2, el PRV, EL PRRSV, EL PEDV, EL FMDV, EL CSFV y el VIS para analizar la especificidad de la cdPCR triplex. NC, Control negativo.
Figura 3. El análisis de especificidad de la cdPCR triplex (A-C). Se muestran los resultados para el gen B646L (A), el gen F1055L (B) y el gen E183L (C), respectivamente. Se utilizaron el virus de la peste porcina africana genotipo I, el virus de la peste porcina africana genotipo II, la PCV2, el PRV, EL PRRSV, EL PEDV, EL FMDV, EL CSFV y el VIS para analizar la especificidad de la cdPCR triplex. NC, Control negativo.
3.5 Análisis de sensibilidad
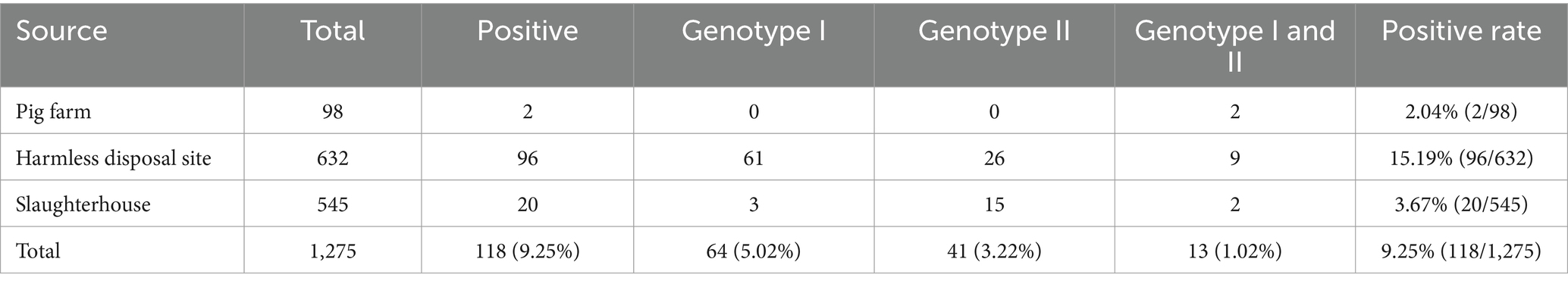
Con el fin de evaluar el LOD de la cdPCR triplex, se utilizaron las mezclas de tres construcciones plásmidas p-dASFV-B646L, p-dASFV-F1055L y p-dASFV-E183L de 1,0 × 105 a 1.0 × 10−2 Se utilizaron copias/μL (concentración final) como plantillas. Los resultados mostraron que el número de gotas positivas disminuyó en un gradiente a medida que disminuyeron las concentraciones de las mezclas. De acuerdo con la distribución de Poisson, los LOD de p-dASFV-B646L, p-dASFV-F1055L y p-dASFV-E183L fueron de 6,5, 4,5 y 5,75 copias/reacción, respectivamente (Figura 4).
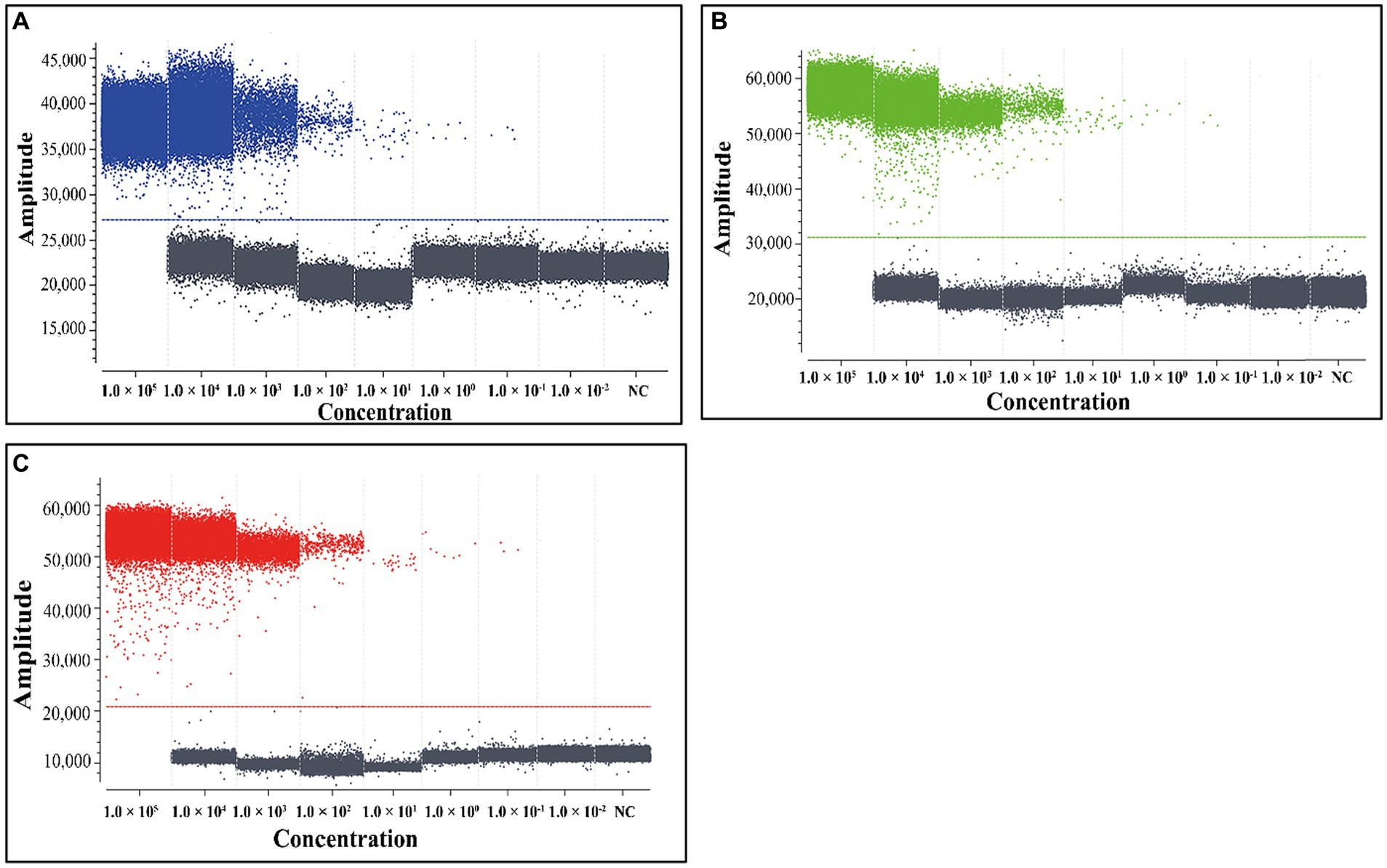 Figura 4. Análisis de sensibilidad de la cdPCR triplex (A-C). Las construcciones plásmidas p-dASFV-B646L (A), p-dASFV-F1055L (B) y p-dASFV-E183L (C) se utilizan para el análisis de sensibilidad, respectivamente. Las mezclas de tres construcciones plásmidas con concentraciones finales de 1,0 × 105 a 1.0 × 10−2 Se utilizaron copias/μL como plantillas. NC, Control negativo.
Figura 4. Análisis de sensibilidad de la cdPCR triplex (A-C). Las construcciones plásmidas p-dASFV-B646L (A), p-dASFV-F1055L (B) y p-dASFV-E183L (C) se utilizan para el análisis de sensibilidad, respectivamente. Las mezclas de tres construcciones plásmidas con concentraciones finales de 1,0 × 105 a 1.0 × 10−2 Se utilizaron copias/μL como plantillas. NC, Control negativo.
Se utilizaron las tres construcciones de plásmidos mezcladas, p-dASFV-B646L, p-dASFV-F1055L y p-dASFV-E183L con un rango de 250 a 0,25 copias/reacción (concentración final) para determinar el número de gotas positivas y las tasas de acierto. Los resultados se muestran en la Tabla 3. Los LOD de las tres construcciones de plásmidos analizadas mediante análisis de regresión PROBIT mostraron que los LOD de p-dASFV-B646L, p-dASFV-F1055L y p-dASFV-E183L fueron 5,120 (3,950-10,500 con un intervalo de confianza [IC] del 95 %), 4,218 (3,456-7,523 con IC del 95 %) y 4,588 (3,695-7,821 con un IC del 95 %), respectivamente (Figura 5).
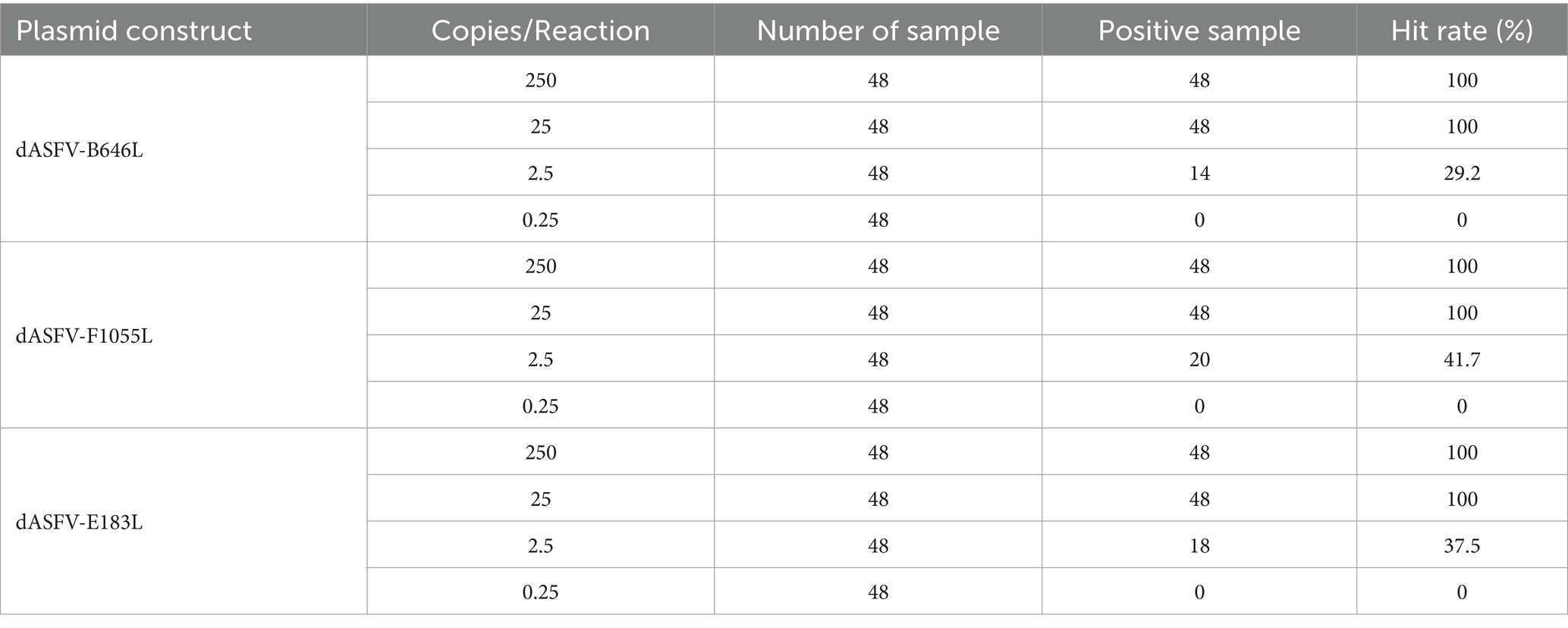 Tabla 3. Número de muestras positivas y tasas de aciertos para la dilución en serie de construcciones de plásmidos.
Tabla 3. Número de muestras positivas y tasas de aciertos para la dilución en serie de construcciones de plásmidos.
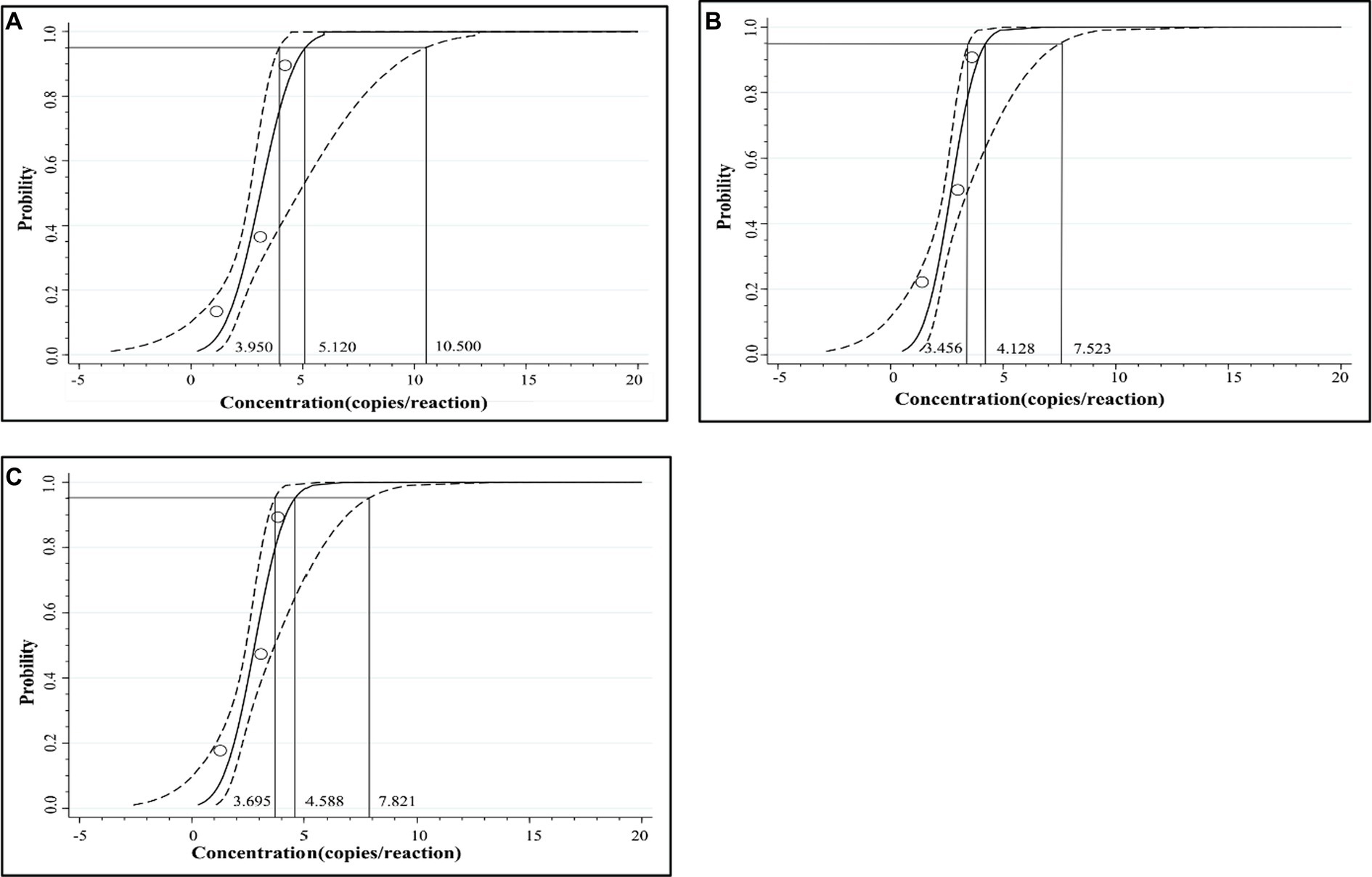 Figura 5. Los resultados del análisis de regresión PROBIT para la cdPCR triplex. Se determinó que los niveles de detalle de p-dASFV-B646L (A), p-dASFV-F1055L (B) y p-dASFV-E183L (C) eran 5,120 (3,950-10,500 con IC 95%), 4,218 (3,456-7,523 con IC 95%) y 4,588 (3,695-7,821 con IC 95%), respectivamente.
Figura 5. Los resultados del análisis de regresión PROBIT para la cdPCR triplex. Se determinó que los niveles de detalle de p-dASFV-B646L (A), p-dASFV-F1055L (B) y p-dASFV-E183L (C) eran 5,120 (3,950-10,500 con IC 95%), 4,218 (3,456-7,523 con IC 95%) y 4,588 (3,695-7,821 con IC 95%), respectivamente.
3.6 Análisis de repetibilidad
Las mezclas de tres construcciones plásmidas con la concentración final de 104, 103y 102 Se utilizaron copias/μL como plantillas. Los resultados mostraron que los CV intraensayo fueron del 1,24 al 2,01 % y los CV entre ensayos del 1,30 al 2,53 % (Tabla 4).
3.7 Ensayo de las muestras clínicas
Se utilizó la cdPCR triplex establecida para evaluar las 1.275 muestras clínicas de la provincia de Guangxi. Los resultados mostraron que se detectaron un total de 118 (9,25%, 118/1.275) muestras positivas para el virus de la PPA, incluyendo 64 (5,02%, 64/1.275) muestras positivas de genotipo I, 41 (3,22%, 41/1.275) muestras positivas de genotipo II y 13 (1,02%, 13/1.275) muestras positivas de coinfección con genotipos I y II (Tabla 5). Se muestra que los diagramas de puntos 3D muestran los datos de coinfecciones en muestras clínicas mediante el uso de diagramas de dispersión tridimensionales para permitir una visualización inmediata (Figura 6).
 Figura 6. Los diagramas de dispersión 3D de la intensidad de fluorescencia para las muestras clínicas. Los datos se obtuvieron en los canales de adquisición FAM (azul), Cy5 (rojo) y VIC (verde).
Figura 6. Los diagramas de dispersión 3D de la intensidad de fluorescencia para las muestras clínicas. Los datos se obtuvieron en los canales de adquisición FAM (azul), Cy5 (rojo) y VIC (verde).
Las 1.275 muestras clínicas también se analizaron mediante la qPCR triplex reportada por Qian et al. (23). Los resultados mostraron que se detectaron un total de 105 (8,24%, 105/1.275) muestras positivas para el virus de la PPA, incluyendo 58 (4,55%, 58/1.275) muestras positivas de genotipo I, 36 (2,82%, 36/1.275) muestras positivas de genotipo II y 11 (0,86%, 11/1.275) muestras positivas de coinfección con genotipos I y II (Tabla 6). Además, la sensibilidad clínica y la especificidad clínica de la cdPCR triplex fueron de 100 y 98,89%, respectivamente (Tabla 7). Las concordancias entre la cdPCR triplex y la qPCR triplex fueron superiores al 98,98% (Tabla 6).
4 Discusión
La PPA ha causado importantes pérdidas económicas a los cerdos domésticos y a los jabalíes debido al curso agudo de la enfermedad y a la elevada tasa de mortalidad (2). La Organización Mundial de Sanidad Animal (OMSA) identifica la PPA como una de las enfermedades de la lista. La peste porcina africana era endémica en el continente africano antes de 1957, pero se extendió fuera de este continente a partir de entonces. La PPA de genotipo II, altamente patogenética, se detectó por primera vez en China en 2018 (7), y la PPA de genotipo I, relativamente menos virulenta, se descubrió por primera vez en 2020 (21). Desde el 2018, se han notificado casos de PPA en muchos países asiáticos (5, 10, 11). Hasta la fecha, la PPA se ha fundado en una multitud de países de África, Asia, Europa y América (10-14). Aunque la PPA es epidémica en todo el mundo, los genotipos I y II del virus de la PPA fueron los dos únicos genotipos de los 24 genotipos del virus de la peste porcina africana que se han identificado fuera del continente africano hasta ahora (16-18). Sin embargo, debido al aumento uniforme del comercio mundial y al volumen de animales y productos animales importados y exportados, siempre existe el riesgo de que el virus de la PPA se propague desde el continente africano a otros continentes (37-39). El desarrollo de un método rápido, fiable y preciso para la detección, vigilancia y diagnóstico del virus de la PPA es muy urgente para los países en los que circula la PPA. La qPCR se ha utilizado ampliamente para detectar ácidos nucleicos virales en muchos laboratorios, ya que esta técnica es conocida por su alta sensibilidad, excelente especificidad y fiabilidad. Permite la identificación precisa y eficiente de las infecciones virales, lo que permite intervenciones oportunas y específicas para la prevención y el control de la enfermedad (29, 30). Varios informes han desarrollado qPCR para detectar el virus de la peste porcina africana y distinguir los genotipos I y II del virus de la peste porcina africana (23, 40-43). Sin embargo, la qPCR tiene las desventajas de la fluctuación de los valores de Ct en función de la configuración del umbral, la alta sensibilidad a los inhibidores de la reacción y el complejo procedimiento de generación de curvas de calibración. Por lo tanto, la dPCR es una nueva y mejor opción para la detección de ácidos nucleicos virales. La dPCR tiene las ventajas de la cuantificación absoluta de la plantilla independiente de los valores de Ct y las curvas estándar, la excelente sensibilidad y precisión para cargas bajas de plantillas y la baja sensibilidad a los inhibidores de la PCR. En varios informes se ha establecido la dPCR para la detección del virus de la PPA (32-36), pero no se ha establecido ninguna dPCR para detectar simultáneamente los genotipos I y II del virus de la PPA. En este estudio, se desarrolló una cdPCR triplex para detectar y diferenciar el virus de la PPA genotipo I y el genotipo II. Además de las ventajas mencionadas anteriormente de la cdPCR, la cdPCR múltiplex puede hacer un uso completo del aparato para detectar varios virus en una reacción al mismo tiempo, lo que disminuye drásticamente la tasa de detección. De acuerdo con nuestro cálculo anterior, cuesta alrededor de US $ 17.67 / muestra por la dPCR singleplex, US $ 7.86 / muestra por la dPCR múltiplex y US $ 3.83 / muestra por la qRT-PCR múltiplex [33]. Esto ayuda a que la cdPCR triplex establecida en este estudio se aplique para la detección de alto rendimiento de muestras clínicas, especialmente para las muestras clínicas de baja carga viral.
En este estudio, se diseñaron tres pares de cebadores específicos y las sondas correspondientes basados en el gen B646L, el gen F1055L y el gen E183L, respectivamente. Los cebadores y la sonda dirigidos al gen B646L se utilizaron como cebadores y sondas universales para detectar 24 genotipos de PPA. Las construcciones plasmídicas sintetizadas de 24 genotipos de PPA se han utilizado para validar y confirmar la viabilidad de los cebadores y la sonda (23). Los cebadores y la sonda dirigidos al gen F1055L se utilizaron para amplificar específicamente el virus de la PPA genotipo I, y los cebadores y la sonda dirigidos al gen E183L se utilizaron para amplificar específicamente el virus de la PPA del genotipo II. Después de optimizar los parámetros de la reacción, como las concentraciones de cebadores y sondas, las temperaturas de recocido y los ciclos de reacción, se desarrolló con éxito una cdPCR triplex. El ensayo logró una especificidad notable, asegurando que solo se amplificaran y detectaran los ácidos nucleicos virales diana del virus de la PPA. La sensibilidad del ensayo se ha mejorado considerablemente, obteniendo los LOD de 5.120, 4.218 y 4.588 copias/reacción para los genes B646L, F1055L y E183L, respectivamente, mientras que la qPCR multiplex utilizando los mismos cebadores y sonda tuvo los LOD de 399.647, 374.409, 355.083 copias/reacción para los genes B646L, F1055L y E183L, respectivamente (23), lo que indica que la cdPCR triplex tuvo 78,06, 88,76, 77,39 veces mayor que las de la qPCR triplex, respectivamente. La excelente sensibilidad del ensayo le permite detectar cargas virales muy bajas, lo cual es crucial para la etapa temprana de la infección. El análisis de repetibilidad del ensayo fue excelente, con los CV intraensayo e interensayo entre 1,24 y 2,53%. La R2 Los valores de las curvas estándar fueron ≥ 0,997, lo que indica una buena relación lineal entre las plantillas iniciales y los valores positivos de gota. El método cdPCR ofrece un LOD notablemente más bajo en comparación con la qPCR (23), lo que lo hace más adecuado para evaluar las muestras clínicas con cargas virales bajas. La cdPCR triplex tuvo una sensibilidad clínica y una especificidad de 100 y 98,89%, respectivamente, y concordancia superior al 98,98% con la qPCR triplex de referencia cuando se utilizaron para evaluar las 1.275 muestras clínicas. Estos avances han mejorado significativamente la precisión y fiabilidad de la cdPCR triplex desarrollada para la detección de ácidos nucleicos virales.
La cdPCR triplex desarrollada se utilizó para evaluar 1.275 muestras clínicas recogidas entre marzo de 2023 y agosto de 2023 en la provincia de Guangxi. Las tasas de positividad de los genotipos I, II y coinfección de los genotipos I + II fueron de 5,02, 3,22 y 1,02%, respectivamente, con una tasa de positividad total del 9,25% en las muestras clínicas, lo que indica que el virus de la PPA sigue siendo epidémico en la provincia de Guangxi. Sin embargo, en comparación con los datos anteriores notificados en la provincia de Guangxi (19, 22, 23, 33, 36, 44-46), la tasa de positividad del virus de la PPA en la provincia de Guangxi en este estudio disminuyó significativamente. En la provincia china de Guangxi, las tasas de positividad notificadas del virus de la PPA en muestras clínicas recogidas durante diferentes períodos fueron las siguientes: 57,14 % (192/336) de enero de 2019 a diciembre de 2020 (19), 45,58 % (232/509) de octubre de 2018 a diciembre de 2020 (22), 43,75 % (168/384) de octubre de 2018 a diciembre de 2019 (44), 30,10 % (87/289) de enero de 2018 a marzo de 2021 (33), 25,63% (293/1.143) de febrero de 2018 a marzo de 2021 (45), 14,17% (214/1.510) de enero de 2022 a diciembre de 2022 (36), 12,60% (534/4.239) de enero de 2021 a diciembre de 2021 (46), 8,16% (287/3.519) de marzo de 2019 a febrero de 2023 (23), 9,25% (118/1.275) de marzo de 2023 a agosto de 2023 (este estudio). En general, las tasas de positividad de la PPA en la provincia de Guangxi han disminuido gradualmente desde 2018, el año en que la PPA estalló por primera vez en China. Además, cabe destacar que la tasa de positividad del genotipo I fue mayor que la del genotipo II en este estudio, mientras que el genotipo II fue el genotipo predominante en los informes anteriores (19, 22, 23, 33, 36, 44-46), lo que indica que el genotipo I podría haberse convertido en el principal genotipo circulante en la provincia de Guangxi en 2023. lo que debe confirmarse a través de investigaciones epidemiológicas más amplias y prolongadas. Desafortunadamente, hay pocos informes sobre los resultados del monitoreo del virus de la PPA en varias provincias de China, por lo que no podemos conocer la situación epidémica actual en varias regiones. La disminución de la prevalencia del virus de la peste porcina africana en la provincia de Guangxi sugiere que las medidas de prevención y control llevadas a cabo en China fueron muy eficaces. Las principales medidas incluyeron una estricta bioseguridad, una detección precisa y un diagnóstico rápido, descartando a los cerdos positivos para el virus de la peste porcina africana en la fase muy temprana de la infección (47, 48). Por lo tanto, un método rápido, sensible y preciso para detectar el virus de la PPA es vital para identificar con precisión a los cerdos infectados en etapa temprana y eliminarlos de manera decisiva en los puntos designados. Este ensayo se puede utilizar para detectar de manera precisa y eficiente las cepas de los genotipos I y II del virus de la PPA, lo que permite intervenciones rápidas y específicas para prevenir una mayor propagación y mitigar el impacto en las poblaciones de cerdos.
5 Conclusión
Se desarrolló una cdPCR triplex rápida, sensible y precisa para detectar y diferenciar el virus de la PPA de genotipo I y el genotipo II. El ensayo altamente sensible, específico y reproducible es adecuado para la detección e investigación del virus de la PPA en muestras clínicas. Además, las cepas de genotipo I del virus de la peste porcina africana son las cepas circulantes más importantes que las cepas de genotipo II en la provincia de Guangxi en China en la actualidad.
Declaración de disponibilidad de datos
Los datos brutos que respaldan las conclusiones de este artículo serán puestos a disposición por los autores, sin reservas indebidas.
Declaración ética
Este estudio fue aprobado por el Centro para el Control y la Prevención de Enfermedades Animales (CADC) de Guangxi (No. 2020-A-01). El CADC de Guangxi fue aprobado por el Ministerio de Agricultura y Asuntos Rurales de China para la recolección y detección del virus de la PPA en muestras clínicas (número de aprobación: 2018-154-25). El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
KS: Redacción – revisión y edición, adquisición de fondos. XQ: Redacción – borrador original, Metodología. YS: Redacción – borrador original, Metodología. HW: Escritura – borrador original, Validación, Investigación. YP: Escritura – borrador original, software, curación de datos. FL: Redacción – borrador original, administración del proyecto. QZ: Escritura – borrador original, visualización. SM: Redacción, revisión y edición, administración de proyectos. LH: Redacción – borrador original, Validación, Investigación. ZL: Redacción, revisión y edición, adquisición de fondos.
Financiación
El/los autor/es declaran/n que se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este estudio contó con el apoyo del Programa Clave de Investigación y Desarrollo (No. AB21238003) de la Oficina de Ciencia y Tecnología de Guangxi (China) y el Programa de Ciencia y Tecnología Agrícola (Nº 202031) de la Oficina Agrícola y Rural de Guangxi (China).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Notas
1. ^https://blast.ncbi.nlm.nih.gov/Blast.cgi
2. ^https://www.ibm.com/cn-zh/spss
3. ^https://www.stata.com/products/windows/
Referencias
1. Wang, G, Xie, M, Wu, W y Chen, Z. Estructuras y diversidades funcionales de las proteínas del virus de la PPA. Virus. (2021) 13:2124. doi: 10.3390/v13112124
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Wang, F, Zhang, H, Hou, L, Yang, C, y Wen, Y. Avance del virus de la peste porcina africana en los últimos años. Res Vet Sci. (2021) 136:535–9. doi: 10.1016/j.rvsc.2021.04.004
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Galindo, I, y Alonso, C. Virus de la peste porcina africana: una revisión. Virus. (2017) 9:103. doi: 10.3390/v9050103
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Gaudreault, NN, Madden, DW, Wilson, WC, Trujillo, JD, y Richt, JA. Virus de la peste porcina africana: un arbovirus de ADN emergente. Front Vet Sci. (2020) 7:215. doi: 10.3389/fvets.2020.00215
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Xin, G, Kuang, Q, Le, S, Wu, W, Gao, Q, Gao, H, et al. Origen, diversidad genómica y evolución del virus de la peste porcina africana en Asia oriental. Virus Evol. (2023) 9:vead060. doi: 10.1093/ve/vead060
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Penrith, ML, Van Heerden, J, Heath, L, Abworo, EO y Bastos, ADS. Revisión de los virus de la peste porcina africana adaptados al cerdo dentro y fuera de África. Patógenos. (2022) 11:1190. doi: 10.3390/pathogens11101190
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Zhou, X, Li, N, Luo, Y, Liu, Y, Miao, F, Chen, T, et al. Aparición de la peste porcina africana en China, 2018. Transbound Emerg Dis. (2018) 65:1482–4. doi: 10.1111/tbed.12989
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Tao, D, Sun, D, Liu, Y, Wei, S, Yang, Z, An, T, et al. Un año del brote de peste porcina africana en China. Acta Trop. (2020) 211:105602. doi: 10.1016/j.actatropica.2020.105602
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Zhou, L, Yu, EYW, Wang, S y Sun, C. Epidemia de peste porcina africana en China. Rec. Veterinario (2019) 184:713. doi: 10.1136/vr.l4026
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Mighell, E, y Ward, diputado. La peste porcina africana se extendió por Asia, 2018-2019. Transbound Emerg Dis. (2021) 68:2722–32. doi: 10.1111/tbed.14039
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Dixon, LK, Stahl, K, Jori, F, Vial, L, y Pfeiffer, DU. Epidemiología y control de la peste porcina africana. Annu Rev Anim Biosci. (2020) 8:221–46. doi: 10.1146/annurev-animal-021419-083741
12. Gonzales, W, Moreno, C, Durán, U, Henao, N, Bencosme, M, Lora, P, et al. Peste porcina africana en República Dominicana. Transbound Emerg Dis. (2021) 68:3018–9. doi: 10.1111/tbed.14341
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Sánchez-Cordón, PJ, Montoya, M, Reis, AL, y Dixon, LK. Peste porcina africana: una enfermedad viral reemergente que amenaza a la industria porcina mundial. Veterinario J. (2018) 233:41–8. doi: 10.1016/j.tvjl.2017.12.025
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Ata, EB, Li, ZJ, Shi, CW, Yang, GL, Yang, WT y Wang, CF. Virus de la peste porcina africana: un aumento mundial y una amenaza continua para la cría de cerdos. Microb Pathog. (2022) 167:105561. doi: 10.1016/j.micpath.2022.105561
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Danzetta, ML, Marenzoni, ML, Iannetti, S, Tizzani, P, Calistri, P, y Feliziani, F. Peste porcina africana: lecciones que aprender de las experiencias pasadas de erradicación. Una revisión sistemática. Front Vet Sci. (2020) 7:296. doi: 10.3389/fvets.2020.00296
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Achenbach, JE, Gallardo, C, Nieto-Pelegrín, E, Rivera-Arroyo, B, Degefa-Negi, T, Arias, M, et al. Identificación de un nuevo genotipo del virus de la peste porcina africana en cerdos domésticos de Etiopía. Transbound Emerg Dis. (2017) 64:1393–404. doi: 10.1111/tbed.12511
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Qu, H, Ge, S, Zhang, Y, Wu, X y Wang, Z. Una revisión sistemática de los genotipos y serogrupos del virus de la peste porcina africana. Genes de virus. (2022) 58:77–87. doi: 10.1007/s11262-021-01879-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Gallardo, C, Casado, N, Soler, A, Djadjovski, I, Krivko, L, Madueño, E, et al. Un método de genotipado con enfoque multigénico identifica 24 grupos genéticos dentro del genotipo II-virus de la peste porcina africana europea que circulan entre 2007 y 2022. Front Vet Sci. (2023) 10:1112850. doi: 10.3389/fvets.2023.1112850
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Shi, K, Liu, H, Yin, Y, Si, H, Long, F y Feng, S. Caracterización molecular del virus de la peste porcina africana de los brotes de 2019-2020 en la provincia de Guangxi, sur de China. Front Vet Sci. (2022) 9:912224. doi: 10.3389/fvets.2022.912224
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Ito, S, Bosch, J, Martínez-Avilés, M, y Sánchez-Vizcaíno, JM. La evolución de la peste porcina africana en China: ¿una amenaza global? Front Vet Sci. (2022) 9:828498. doi: 10.3389/fvets.2022.828498
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. Sun, E, Huang, L, Zhang, X, Zhang, J, Shen, D, Zhang, Z, et al. Genotipo I Los virus de la peste porcina africana surgieron en cerdos domésticos en China y causaron infección crónica. Los microbios emergentes infectan. (2021) 10:2183–93. doi: 10.1080/22221751.2021.1999779
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Liu, H, Shi, K, Zhao, J, Yin, Y, Chen, Y, Si, H, et al. Desarrollo de un ensayo qRT-PCR multiplex de un solo paso para la detección del virus de la peste porcina africana, el virus de la peste porcina clásica y el pestivirus porcino atípico. BMC Vet Res. (2022) 18:43. doi: 10.1186/s12917-022-03144-4
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Qian, X, Hu, L, Shi, K, Wei, H, Shi, Y, Hu, X, et al. Desarrollo de una PCR cuantitativa triplex en tiempo real para la detección y diferenciación de genotipos I y II del virus de la peste porcina africana. Front Vet Sci. (2023) 10:1278714. doi: 10.3389/fvets.2023.1278714
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Zhao, D, Sun, E, Huang, L, Ding, L, Zhu, Y, Zhang, J, et al. Virus de la peste porcina africana recombinante de genotipo I y II altamente letales detectados en cerdos. Nat Commun. (2023) 14:3096. doi: 10.1038/s41467-023-38868-w
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Gao, X, Liu, T, Liu, Y, Xiao, J y Wang, H. Transmisión de la peste porcina africana en China a través del comercio legal de cerdos vivos. Transbound Emerg Dis. (2021) 68:355–60. doi: 10.1111/tbed.13681
26. Bustin, SA, y Mueller, R. PCR con transcripción inversa en tiempo real (qRT-PCR) y su uso potencial en el diagnóstico clínico. Clin Sci (Londres). (2005) 109:365–79. doi: 10.1042/CS20050086
27. Kralik, P, y Ricchi, M. Una guía básica para la PCR en tiempo real en el diagnóstico microbiano: definiciones, parámetros y todo. Microbiol frontal. (2017) 8:108. doi: 10.3389/fmicb.2017.00108
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Kuypers, J, Jerome, KR y Kraft, CS. Aplicaciones de la PCR digital para la microbiología clínica. J Clin Microbiol. (2017) 55:1621–8. doi: 10.1128/JCM.00211-17
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Kojabad, AA, Farzanehpour, M, Galeh, HEG, Dorostkar, R, Jafarpour, A, Bolandian, M, et al. PCR digital en gotas de ADN/ARN viral, avances actuales, retos y perspectivas de futuro. J Med Virol. (2021) 93:4182–97. doi: 10.1002/jmv.26846
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Tan, LL, Loganathan, N, Agarwalla, S, Yang, C, Yuan, W, Zeng, J, et al. Plataformas comerciales actuales de dPCR: tecnología y revisión del mercado. Crit Rev Biotechnol. (2023) 43:433–64. doi: 10.1080/07388551.2022.2037503
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Madic, J, Zocevic, A, Senlis, V, Fradet, E, Andre, B, Muller, S, et al. PCR digital de cristal de tres colores. Biomol Detect Quantif. (2016) 10:34–46. doi: 10.1016/j.bdq.2016.10.002
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Jia, R, Zhang, G, Liu, H, Chen, Y, Zhou, J, Liu, Y, et al. Novedosa aplicación de PCR digital con chip nanofluídico para la detección del virus de la peste porcina africana. Front Vet Sci. (2021) 7:621840. doi: 10.3389/fvets.2020.621840
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Shi, K, Chen, Y, Yin, Y, Long, F, Feng, S, Liu, H, et al. Una PCR digital de cristal múltiple para la detección del virus de la peste porcina africana, el virus de la peste porcina clásica y el virus del síndrome respiratorio y reproductivo porcino. Front Vet Sci. (2022) 9:926881. doi: 10.3389/fvets.2022.926881
Resumen de PubMed | Texto completo de Crossref | Google Académico
34. Zhu, J, Jian, W, Huang, Y, Gao, Q, Gao, F, Chen, H, et al. Desarrollo y aplicación de un ensayo de reacción en cadena de la polimerasa digital en gotas dúplex para la detección y diferenciación del virus de la peste porcina africana de tipo salvaje y con deleción EP402R. Front Vet Sci. (2022) 9:905706. doi: 10.3389/fvets.2022.905706
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Wu, X, Xiao, L, Lin, H, Chen, S, Yang, M, An, W, et al. Desarrollo y aplicación de una reacción en cadena de la polimerasa digital en gotas (ddPCR) para la detección e investigación del virus de la peste porcina africana. Can J Vet Res. (2018) 82:70–4.
36. Shi, K, Zhao, K, Wei, H, Zhou, Q, Shi, Y, Mo, S, et al. PCR digital de cristal triplex para la detección y diferenciación de la cepa de tipo salvaje y la cepa MGF505-2R e I177L del virus de la peste porcina africana. Patógenos. (2023) 12:1092. doi: 10.3390/pathogens12091092
Resumen de PubMed | Texto completo de Crossref | Google Académico
37. Cheng, J y Ward, diputados. Factores de riesgo para la propagación de la peste porcina africana en China: una revisión sistemática de la literatura en lengua china. Transbound Emerg Dis. (2022) 69:E1289–98. doi: 10.1111/tbed.14573
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Kedkovid, R, Sirisereewan, C, y Thanawongnuwech, R. Principales enfermedades virales porcinas: una perspectiva asiática después de la introducción de la peste porcina africana. Gestión de la Salud Porcina. (2020) 6:20. doi: 10.1186/s40813-020-00159-x
Resumen de PubMed | Texto completo de Crossref | Google Académico
39. Taylor, RA, Condoleo, R, Simons, RRL, Gale, P, Kelly, LA, y Snary, EL. El riesgo de infección por el virus de la peste porcina africana en cerdos europeos a través del movimiento de verracos y el comercio legal de cerdos y carne de cerdo. Front Vet Sci. (2020) 6:486. doi: 10.3389/fvets.2019.00486
Resumen de PubMed | Texto completo de Crossref | Google Académico
40. Cao, S, Lu, H, Wu, Z y Zhu, S. Un ensayo de PCR cuantitativa fluorescente dúplex para distinguir las cepas de genotipo I y II del virus de la peste porcina africana en cepas epidémicas chinas. Front Vet Sci. (2022) 9:998874. doi: 10.3389/fvets.2022.998874
Resumen de PubMed | Texto completo de Crossref | Google Académico
41. Li, X, Hu, Y, Liu, P, Zhu, Z, Liu, P, Chen, C, et al. Desarrollo y aplicación de un ensayo de PCR dúplex en tiempo real para la diferenciación de los genotipos I y II de los virus de la peste porcina africana. Transbound Emerg Dis. (2022) 69:2971–9. doi: 10.1111/tbed.14459
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Ilya, T, Monoldorova, S, Kang, SS, Yun, S, Byeon, HS, Mariia, N, et al. Desarrollo de un ensayo de amplificación de la polimerasa recombinasa en tiempo real para la detección rápida de los genotipos I y II del virus de la peste porcina africana. Patógenos. (2022) 11:439. doi: 10.3390/pathogens11040439
Resumen de PubMed | Texto completo de Crossref | Google Académico
43. Gao, Q, Feng, Y, Yang, Y, Luo, Y, Gong, T, Wang, H, et al. Establecimiento de un ensayo dual de PCR en tiempo real para la identificación de los genotipos I y II del virus de la peste porcina africana en China. Front Vet Sci. (2022) 9:882824. doi: 10.3389/fvets.2022.882824
Resumen de PubMed | Texto completo de Crossref | Google Académico
44. Liu, H, Shi, K, Sun, W, Zhao, J, Yin, Y, Si, H, et al. Desarrollo de un ensayo RT-PCR múltiple para la detección simultánea del virus de la peste porcina africana, el virus de la peste porcina clásica y el pestivirus porcino atípico. Métodos J Virol. (2021) 287:114006. doi: 10.1016/j.jviromet.2020.114006
Resumen de PubMed | Texto completo de Crossref | Google Académico
45. Chen, Y, Shi, K, Liu, H, Yin, Y, Zhao, J, Long, F, et al. Desarrollo de un ensayo qRT-PCR múltiple para la detección del virus de la peste porcina africana, el virus de la peste porcina clásica y el virus del síndrome respiratorio y reproductivo porcino. J Vet Sci. (2021) 22:E87. doi: 10.4142/jvs.2021.22.e87
Resumen de PubMed | Texto completo de Crossref | Google Académico
46. Zhao, K, Shi, K, Zhou, Q, Xiong, C, Mo, S, Zhou, H, et al. El desarrollo de un ensayo de PCR cuantitativo múltiple en tiempo real para la detección diferencial de la cepa de tipo salvaje y la cepa MGF505-2R, EP402R e I177L con deleción génica del virus de la peste porcina africana. Animales. (2022) 12:1754. doi: 10.3390/ani12141754
Resumen de PubMed | Texto completo de Crossref | Google Académico
47. Gao, L, Sun, X, Yang, H, Xu, Q, Li, J, Kang, J, et al. Situación epidémica y medidas de control de los brotes de peste porcina africana en China 2018-2020. Transbound Emerg Dis. (2021) 68:2676–86. doi: 10.1111/tbed.13968
Resumen de PubMed | Texto completo de Crossref | Google Académico
48. Liu, Y, Zhang, X, Qi, W, Yang, Y, Liu, Z, An, T, et al. Estrategias de prevención y control de la peste porcina africana y avances en la repoblación de granjas porcinas en China. Virus. (2021) 13:2552. doi: 10.3390/v13122552
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: Virus de la peste porcina africana (virus de la peste porcina africana), PCR digital de cristal múltiplex (cdPCR), coinfección, genotipo I, genotipo II
Cita: Shi K, Qian X, Shi Y, Wei H, Pan Y, Long F, Zhou Q, Mo S, Hu L y Li Z (2024) Una PCR digital de cristal triplex para la detección del virus de la peste porcina africana de genotipos I y II. Frente. Vet. Sci. 11:1351596. doi: 10.3389/fvets.2024.1351596
Recibido: 06 de diciembre de 2023; Aceptado: 19 de marzo de 2024;
Publicado: 02 Abril 2024.
Editado por:
Douglas Paul Gladue, Servicio de Investigación Agrícola (USDA), Estados Unidos
Revisado por:
Bin Zhou, Universidad Agrícola de Nanjing, China
Zaheer Ahmed, Departamento de Agricultura de los Estados Unidos (USDA), Estados Unidos
Derechos de autor © 2024 Shi, Qian, Shi, Wei, Pan, Long, Zhou, Mo, Hu y Li. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Kaichuang Shi, shikaichuang@126.com; Zongqiang Li, lizq20@163.com
†Estos autores han contribuido igualmente a este trabajo
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía