Comunidad microbiana hepática y transcriptoma asociado del huésped en terneros con acidosis inducida por alimento
Comunidad microbiana hepática y transcriptoma asociado del huésped en terneros con acidosis inducida por alimento
 Wenli Li1*
Wenli Li1*  Anna Larsen1,2
Anna Larsen1,2  Brianna Murphy3
Brianna Murphy3  Priscila Fregulia1,3
Priscila Fregulia1,3- 1Centro de Investigación de Forrajes Lácteos de EE. UU., Servicio de Investigación Agrícola del USDA, Madison, WI, Estados Unidos
- 2Departamento de Ciencias Animales y Lácteas, Universidad de Wisconsin-Madison, Madison, WI, Estados Unidos
- 3Instituto Oak Ridge para la Ciencia y la Educación, Oak Ridge, Tennessee, Estados Unidos
Introducción: En la industria láctea, los terneros suelen alimentarse con dietas ricas en carbohidratos altamente fermentables y bajas en alimentos fibrosos para maximizar el desarrollo de las papilas ruminales y los tejidos. Los terneros que siguen estas dietas son vulnerables a desarrollar acidosis ruminal. Prevalente en bovinos, el absceso hepático (AI) se considera una secuela de la acidosis ruminal. Los AL pueden causar una condena significativa de la función hepática y una disminución del crecimiento y la producción. Actualmente, sabemos poco sobre el microbioma hepático en terneros con acidosis inducida por piensos.
Métodos: Utilizando nuestro modelo establecido de acidosis ruminal, en el que los terneros jóvenes fueron alimentados con una dieta inductora de acidosis (AC) o embotada (control) desde el nacimiento hasta las 17 semanas de edad, investigamos los cambios en la comunidad microbiana en el hígado resultantes de la acidosis ruminal. Ocho terneros fueron asignados aleatoriamente a cada dieta, con cuatro animales por tratamiento. El epitelio ruminal y los tejidos hepáticos se recolectaron a las 17 semanas de edad, inmediatamente después de la eutanasia. Se extrajeron los ARN totales y se realizó la secuenciación del transcriptoma completo. Las lecturas de ARN microbiano se enriquecieron bioinformáticamente y se utilizaron para la clasificación de la taxonomía microbiana utilizando Kraken2.
Resultados: Los terneros AC mostraron una ganancia de peso significativamente menor en el transcurso del experimento, además de un pH ruminal significativamente más bajo y una degradación ruminal en comparación con el grupo control (p < 0,05). En el hígado, un total de 29 géneros mostraron un cambio significativo (p < 0.05) en abundancia (> 2 veces) entre los tratamientos a las 17 semanas de edad. Entre estos, se han reportado casos de Fibrobacter, Treponema, Lactobacillus y Olsenella en abscesos hepáticos en bovinos. Se observaron cambios simultáneos en la abundancia de 9 de los géneros tanto en el hígado como en los tejidos ruminales recolectados a las 17 semanas de edad, lo que indica una posible diafonía entre las comunidades microbianas epiteliales del hígado y el rumen. Se identificó una asociación significativa entre el gen hepático del huésped y sus taxones microbianos incluidos. Además de identificar taxones microbianos previamente reportados en hígado con abscesos bovinos, en este estudio se identificó un nuevo repertorio de taxones microbianos transcritos activamente.
Discusión: Mediante el empleo de la secuenciación del metatranscriptoma, nuestro estudio pintó una imagen del microbioma hepático en terneros jóvenes con o sin acidosis inducida por el alimento. Nuestro estudio sugirió que el microbioma hepático puede tener un impacto crítico en la fisiología hepática del huésped. Los nuevos hallazgos de este estudio enfatizan la necesidad de un análisis más profundo para descubrir el papel funcional del microbioma residente en el hígado en la acidosis metabólica hepática resultante de la acidosis ruminal relacionada con la alimentación.
1. Introducción
Una de las transformaciones más dramáticas del tracto gastrointestinal (TGI) en los terneros jóvenes ocurre durante el período de destete. Este período se asocia con frecuencia con un deterioro de la salud del tracto gastrointestinal y cambios extremos en el tracto gastrointestinal debido a cambios drásticos en la dieta (1, 2). Los terneros lecheros nacen con un entorno ruminal desprovisto de microbios intestinales y deben ser inoculados desde su entorno, que comienza ~ dentro de los 20 minutos posteriores al nacimiento (3). El desarrollo de la comunidad microbiana ruminal y el ambiente anaeróbico comienza en los primeros días después del nacimiento (4) y está influenciado por múltiples factores ambientales tempranos, incluido el modo de parto (5), la inoculación temprana (6-8) y la intervención dietética temprana en la vida (9-11).
En la industria láctea, los terneros suelen ser manejados para maximizar el desarrollo de las papilas ruminales y los tejidos mediante la alimentación con dietas ricas en carbohidratos altamente fermentables y bajas en alimentos fibrosos (12), debido en parte, a la mayor tasa y extensión del desarrollo ruminal que se puede lograr con estas dietas concentradas (13). Una consecuencia no deseada de estas dietas es el daño a la pared del rumen debido a la producción excesiva de ácidos grasos de cadena corta (14). Además, el aumento de las condiciones acidóticas del rumen puede tener efectos perjudiciales en la fisiología y la tasa de crecimiento de todo el animal (14, 15). El daño de la mucosa ruminal es una de las principales dolencias gastrointestinales que sufren los terneros jóvenes durante el período de destete de los terneros (16), que se combina con una permeabilidad intestinal repentina (17). El aumento de la permeabilidad de la TGI causado por la interrupción de las proteínas de unión estrecha (TJ), incluidas la ocludina, las claudinas, las moléculas de adhesión de unión (JAM) y las ocludinas de zonula (ZO) (18) puede conducir a la translocación bacteriana del TGI al torrente sanguíneo (19). La base de la alteración de la función de barrera del TGI involucra una serie de citocinas, incluyendo TNF-α e IFN-γ (20). Por otro lado, se ha demostrado que los microbios intestinales dirigen la expresión y localización de la proteína TJ tanto en modelos in vitro como in vivo (21) a través de la liberación de péptidos, toxinas (22, 23) o metabolitos (24-26).
Los abscesos hepáticos, prevalentes en el ganado bovino, se consideran auxiliares de la acidosis ruminal. Los AL pueden causar pérdidas económicas significativas como resultado de la condena de la función hepática y la disminución del crecimiento y la producción (27, 28). El impacto perjudicial de la LA en el crecimiento de los animales fue reportado por primera vez por 29, quienes discutieron la incidencia de la LA y su contribución a la reducción de los rendimientos de vísceras. De los bovinos sacrificados en 1940, la tasa de abscesos hepáticos fue del 5,5%. Desde entonces, la tasa de incidencia notificada se ha triplicado hasta el 17,8% (30). Esto podría atribuirse al manejo moderno del ganado y al tratamiento de la alimentación, ya que el desarrollo de AL generalmente se atribuye a la transición a dietas ricas en almidón rápidamente fermentable (28). El consumo de dietas ricas en almidón puede conducir a una producción excesiva de AGCC, disminuyendo el pH ruminal, lo que puede conducir a la acidosis ruminal. A su vez, la pared epitelial del rumen comprometida o cualquier ruptura de la capa epitelial en el tracto alimentario puede permitir que los microbios causales ingresen al hígado a través de la vena porta hepática (31). No está claro si la diversidad de la comunidad microbiana hepática está asociada con la gravedad del absceso hepático. Y no existen herramientas de diagnóstico precoz disponibles para la detección o prevención de las LA.
El estudio de las comunidades microbianas en el hígado absceso se basó tradicionalmente en métodos basados en cultivos, con los que se informó que Fusobacterium necrophorum era el principal agente causal del absceso hepático (32). Más recientemente, se ha utilizado el método de secuenciación de ARNr 16 s para perfilar la comunidad microbiana con el objetivo de evaluar la eficacia de la terapéutica en el pienso para reducir el absceso hepático (33). Aunque la LA y sus comunidades microbianas asociadas se perciben típicamente como una afección observada en el ganado enfermo, un estudio reciente realizado por Stotz y coautores (34) informó de la presencia de comunidades microbianas inherentes en el parénquima hepático bovino normal y sin abscesos, desafiando directamente la suposición común de que el hígado, como órgano inmunológico, no tolerará la colonización bacteriana dentro de sí mismo.
Los terneros alimentados con una dieta altamente fermentable durante el período de destete son los más vulnerables a desarrollar acidosis ruminal. El aumento de las condiciones acidóticas ruminales resultantes de la dieta altamente fermentable y la inducción de daño en la pared del rumen en una ventana crítica que coincide con un desarrollo ecológico microbiano sustancial podría resultar en una disfunción de la barrera intestinal en el ternero. Esto podría conducir a la translocación microbiana y la posterior colonización microbiana del hígado que conduce a la inflamación más adelante en la vida. Utilizando nuestro modelo establecido de acidosis ruminal inducida por alimento en terneros jóvenes, hemos observado cambios significativos en los transcriptomas epiteliales y hepáticos del rumen (35, 36). Además, en el epitelio ruminal y el hígado, identificamos una asociación significativa entre la expresión génica del huésped y la abundancia de las comunidades microbianas unidas al epitelio ruminal. En este estudio, nos sumergimos en las comunidades microbianas incrustadas en el tejido hepático, que es un nuevo ángel del estudio y no se ha informado antes en terneros jóvenes alimentados con una dieta que induce o embota la acidosis. Utilizando una tecnología de secuenciación de transcriptoma completo, nuestro experimento está diseñado para capturar los microbios activos en el hígado. A su vez, investigamos la correlación de abundancia entre los taxones microbianos en el hígado y los perfiles transcriptómicos previamente generados en el hígado del huésped.
2. Materiales y métodos
2.1. Acidosis inducida por piensos en terneros jóvenes
Los terneros incluidos en este estudio formaron parte de un estudio más amplio que se ha publicado (14, 35, 36). El protocolo animal (A005848) está aprobado por el Comité de Cuidado y Uso de Animales de la Universidad de Wisconsin-Madison. Todos los procedimientos relacionados con el cuidado y uso de los animales en este estudio se implementaron de acuerdo con las pautas y regulaciones del Centro de Investigación de Forrajes Lecheros de EE. UU.
El procedimiento para la acidosis inducida por el alimento fue descrito en nuestros estudios previos. En resumen, se inscribieron 8 terneros de toro Holstein para este experimento. Durante las primeras 8 semanas después del nacimiento, los terneros se alojaron en conejeras individuales (4,8 m2/ternero), luego fueron trasladados a superconejeras divididas (5.0 m2/ternero) hasta las 16 semanas de edad. Se administraron dos dietas a los terneros. Una de ellas era una dieta rica en almidón y baja en fibra. Esta dieta fue diseñada para causar acidosis ruminal (Aci; peletizada, 42,7% de almidón, 15,1% de fibra detergente neutra (FDN) y 5,56% de azúcar). Se administró como control un iniciador texturizado, que fue diseñado para atenuar la acidosis ruminal (Con, 35,3% de almidón, 25,3% de FDN y 6,17% de azúcar). La composición completa de nutrientes de cada dieta se enumeró en el manuscrito complementario (37). Cuatro terneros fueron asignados aleatoriamente a cada tratamiento dietético. El ensayo comenzó a la semana de edad y duró hasta las 1 semanas. A las 17 semanas de edad, se colocaron cánulas de goma blanda (3 mm de diámetro interior) a cada ternero siguiendo el método de Kristensen y coautores (28). Entre las 38 y las 7 semanas de edad, las cánulas de caucho blando más grandes (9 mm de diámetro interior; Bar-Diamond Inc., Parma, ID, Estados Unidos) para reemplazar las cánulas originales debido al aumento en el tamaño de la fístula. Se insertó un electrodo de pH calibrado en el rumen a través de la cánula antes de cada recolección para medir el pH ruminal. A las 51 h se ofrecía diariamente una cantidad medida de entrante, y diariamente se determinaban los rechazos. El pH ruminal se evaluó en siete puntos temporales (−0800, −8, 4, 0, 2, 4 y 8 h en relación con la alimentación inicial) en un solo día cada dos semanas desde 12 semanas hasta 6 semanas. A las 16 semanas, se realizó una biopsia epitelial ruminal en el saco craneal utilizando una herramienta de biopsia uterina. A las 8 semanas de edad, todos los terneros fueron sacrificados. Se recolectaron tejidos hepáticos y ruminales. Para asegurar la recolección aséptica de los tejidos hepáticos, se extrajo una hoja de hígado y se enjuagó con PBS estéril 17X. La superficie del hígado antes de la incisión se limpió con una toallita estéril con alcohol. A continuación, se utilizó un bisturí de grado quirúrgico para hacer una incisión de 1 cm y se extrajo el tejido hepático del interior, debajo de la incisión, con un nuevo juego de pinzas estériles y bisturí. En una superficie estéril, el tejido hepático se cortó de 5 a 4 mm2 piezas y colocadas en tubos de cierre de caja fuerte Eppendorf (Eppendorf North America, Hauppauge, NY, Estados Unidos). Los tejidos recolectados se congelaron inmediatamente en nitrógeno líquido y se almacenaron a -80 °C para su posterior extracción de ARN.
2.2. Preparación de la biblioteca de extracción y secuenciación de ARN para tejidos hepáticos
Para la extracción del ARN hepático del huésped, se homogeneizaron 50 mg de tejido hepático con el instrumento Precellus Evolution (Instrumento Bertin, Francia) a 6800 rmp durante 30 s por ciclo y se repitió 4 veces. El homogeneizado de tejido se colocó en hielo entre ciclos durante 1 min. Después de la homogeneización de los tejidos, la extracción total de ARN se realizó utilizando RNeasy mini Kit (Qiagen, Alemania) siguiendo las instrucciones del fabricante. Los problemas de las papilas ruminales se homogeneizaron en el homogeneizador Precyllus (Instrumento de Bertin, Francia) a 7.500 RPM durante 30 s por ciclo, con 4 repeticiones. El homogeneizado de tejido se colocó en hielo durante 1 min entre ciclos. Después de la homogeneización, se extrajeron los ARN totales de ambos tipos de tejido siguiendo el protocolo miRNeasy con un instrumento QIAcube (Qiagen, Alemania). La calidad de los ARN extraídos se evaluó utilizando el kit Bioanalyzer RNA 6000 nano (Agilent Technologies, Estados Unidos). Se buscaron muestras de ARN con un valor de número de integridad de ARN (RIN) ≥8 para la cuantificación de ARN utilizando Qubit (ThermoFisher Scientific, Estados Unidos). Un total de ocho muestras de ARN (4 muestras por tratamiento y todas superaron el umbral de RIN) se prepararon en bibliotecas de secuenciación de ARN utilizando el kit Illumina TruSeq ribo-zero Gold siguiendo las instrucciones del fabricante. Para cada muestra, se utilizó 1 μg de ARN total para la preparación de la biblioteca de secuenciación. La cuantificación de las bibliotecas preparadas se realizó utilizando un kit de cuantificación Kapa (KK4873, Kapa systems, Roche, Suiza) con un instrumento QuantStudio 5 RT-qPCR (Thermo Fisher, Estados Unidos). La concentración de la biblioteca se calculó utilizando un kit de cuantificación Kapa (KK4873, Roche, Suiza), siguiendo las instrucciones del fabricante. Utilizando la concentración generada por el kit Kapa, se preparó la agrupación secuencial de acuerdo con el cálculo ofrecido por la calculadora de agrupación.1 Las bibliotecas agrupadas se secuenciaron inicialmente utilizando un kit Illumina nano de 300 ciclos. La agrupación se normalizó aún más para garantizar la misma profundidad de secuenciación de todas las bibliotecas, de acuerdo con las proporciones de índice generadas por la ejecución del nanokit. Las bibliotecas agrupadas finalmente normalizadas se secuenciaron en el instrumento Illumina NextSeq 500, utilizando un cartucho de 300 ciclos de alto rendimiento para generar lecturas de 2 × 150 pb de extremo emparejado.
2.3. Análisis bioinformático y clasificación taxonómica de lecturas microbianas para los tejidos hepáticos
El análisis de los datos de secuenciación siguió el procedimiento descrito en nuestro trabajo publicado anteriormente (35, 36). Las lecturas brutas de secuenciación de ARN se asignaron al genoma de referencia del ganado bovino (ARS-UCD 1.2) utilizando STAR (2.5.2b) (39). El análisis de la expresión génica diferencial entre el tratamiento AC y Control se realizó mediante el paquete de manguitos en gemelos2 (40). Las lecturas no mapeadas del genoma bovino se consideraron de origen microbiano. Para enriquecer aún más las lecturas microbianas, se utilizó SortMeRNA (versión, 2.1b) (41) para mapear las lecturas no mapeadas del genoma del huésped a las bases de datos de ARNr de referencia proporcionadas por Silva (versión 119) (42) y Rfam 11.0 (43). Las lecturas de ARNr enriquecidas se utilizaron para la clasificación taxonómica bacteriana, utilizando Kraken2 (v.2.0.8-beta) (44). Los recuentos de lectura en bruto a nivel de filo y género identificados por Kraken2 se normalizaron mediante la profundidad de secuenciación siguiendo el método publicado anteriormente. Brevemente, el número total de lecturas asignadas al nivel taxonómico dado se dividió por 1.000.000 para obtener el «factor por millón»; (2) El número total de lecturas asignadas a cada nivel taxonómico específico se dividió por el «factor por millón» para obtener el recuento de lecturas normalizado.
2.4. Análisis de la comunidad microbiana hepática
Se utilizó el método multivariado mixMC implementado en el paquete mixOmics R (45) para identificar asociaciones entre el microbioma hepático y los grupos AC x Control. Para este análisis, solo se consideraron taxones con abundancia relativa >0.01% (Tabla Suplementaria S1). A continuación, se utilizó el análisis discriminante de mínimos cuadrados parciales dispersos (sPLS-DA) (46) para identificar las firmas microbianas relacionadas con los grupos AC y control. Seleccionamos el número óptimo de componentes en función de la tasa de error de clasificación balanceada promedio utilizando la distancia máxima en 10 repeticiones de un modelo sPLS-DA con 2 componentes. Se utilizó la tasa de error de clasificación balanceada promedio más baja después de ajustar el modelo sPLS-DA para elegir el número óptimo de variables. Se graficaron los 10 principales géneros discriminantes que contribuyen al componente 1 en sPLS-DA.
2.5. Análisis de asociación entre la expresión génica del huésped y la abundancia de taxones microbianos en el hígado
Para los datos de expresión génica del huésped, nuestro grupo publicó previamente recuentos de lectura a nivel de gen para el hígado del huésped (36). Para el análisis de asociación realizado aquí, se utilizó el recuento de lecturas normalizado (Tabla Suplementaria S1). Se utilizaron recuentos de lectura normalizados a nivel de género como medida para el análisis de abundancia microbiana (Tabla Suplementaria S2). El análisis de correlación r de Pearson entre la expresión génica del hígado huésped y su abundancia a nivel de género microbiano se realizó mediante el uso de la función cor en R. Los valores límite de |r| > 0,7 y p < 0,001 para determinar correlaciones significativas. Se utilizó el recuento de lectura normalizado de 2 para determinar la presencia/expresión de los taxones. Para los genes con asociación significativa con taxones microbianos, la ontología génica (GO) y el análisis de vías se realizaron mediante el uso de DAVID (v2022q4) (47).
2.6. Comparación de la comunidad microbiana entre el hígado y el epitelio ruminal a las 17 semanas de edad
Para este análisis, incluimos las lecturas crudas de ARNr de los datos de la comunidad microbiana de los tejidos del epitelio ruminal recolectados a las 17 semanas de edad. Las lecturas brutas para el epitelio ruminal fueron publicadas previamente por nuestro grupo con un número de acceso NCBI SRA de PRJNA948013 (35). Para ambos tipos de tejidos, se utilizaron recuentos de lectura sin procesar a nivel de género calculados por Kraken2 (v.2.0.8-beta) (44) para realizar un análisis de abundancia diferencial entre los grupos de tratamiento, AC vs. Control usando DEseq2 (v 1.41.5) (48). Los géneros microbianos mostraron una abundancia diferencial significativa (p < 0,05 y cambio de pliegue >2) entre los grupos de tratamiento se compararon entre los tejidos del epitelio hepático y ruminal.
2.7. Análisis de prueba de concepto de taxones microbianos previamente identificados en hígado con absceso bovino
Para el estudio de prueba de concepto, evaluamos la presencia de taxones microbianos previamente reportados significativamente asociados con abscesos hepáticos. Entre estos, varios taxones microbianos han sido considerados como el agente causal del absceso hepático en el ganado bovino. Entre ellos se encuentran Fusobacterium necrophorum (31, 49), Trueperella pyogenes y Salmonella enterica (50, 51). F. necrophorum ganó una atención significativa debido a su presencia ubicua en estudios basados en cultivos (50, 52, 53). En un estudio exhaustivo reciente, Whitlow y sus coautores (54) informaron de una abundancia significativa de Fusobacterium, Bacteroides y Porphyromonas. Y los géneros discriminantes para las comunidades de abscesos dominadas por Fusobacteria como Acinetobacter, Lactobacillus, Psdudomonas y Psychrobacter. Para los abscesos dominados por Bacteroidetes, los géneros discriminantes incluyeron Atopobium, Campylobacter, Filifactor, Helocococcus, Parvimonas y Trueperella. Examinamos la presencia y abundancia de estos taxones en nuestras muestras. Stotz y sus coautores (34) investigaron las comunidades microbianas hepáticas en niños de 6 meses de edad. Los géneros de las 10 OTU más abundantes reportadas por Stotz y sus coautores también se incluyeron en el análisis de prueba de concepto. Para tener en cuenta la variación en la profundidad de secuenciación, se utilizó para este análisis el recuento de lecturas normalizado para cada taxón. Para cada taxón, se accede a su presencia para las 4 muestras de cada grupo de tratamiento y se calculó el conteo promedio de lecturas (Media ± S.E.) para cada grupo de tratamiento.
3. Resultados
3.1. Enriquecimiento de las lecturas microbianas a partir de las lecturas totales de secuenciación de ARN
Para cada muestra de hígado se obtuvo un promedio de 44,7 M ± 1,7 M de lecturas brutas. Un promedio del 74% ± 2% de estas lecturas se mapearon en el genoma del ganado. Utilizando nuestra estrategia de filtrado bioinformático, un promedio de 1,7 M ± 0,17 M de lecturas brutas por muestra fueron de origen microbiano de ARNr. Representa un promedio del 15% ± el 1% de las lecturas no bovinas. Se utilizaron lecturas de ARNr microbiano enriquecido para la clasificación de taxones microbianos utilizando Kraken. La mayoría de las lecturas se clasificaron con éxito, con una tasa de clasificación promedio de 97 ± 0,34% con un nivel de confianza alto. Se realizó un seguimiento de los taxones microbianos tanto a nivel de género como de filo para el análisis comparativo entre los tejidos hepáticos y ruminales.
3.2. Comunidad microbiana hepática y sus cambios entre los grupos de tratamiento a las 17 semanas de edad
Utilizando un punto de corte del recuento de lectura (RC) promedio a nivel de género de 5, se determinaron 92 géneros como presentes en el hígado en este estudio. Se observó un perfil de comunidad microbiana consistente en el hígado en el grupo tratado. En este grupo, más del 98% de las lecturas pertenecen a 8 filos, siendo Proteobacteria, Firmicutes y Actinobacteria los 3 filos más abundantes (Figura 1A). Y más del 95% de las lecturas pertenecen a 18 géneros (Figura 1B). A nivel de filo, 5 filos mostraron un aumento significativo en la abundancia de transcritos en el grupo tratado (Figura 2A). A nivel de género, un total de 29 géneros mostraron cambios significativos (p < 0.05) en abundancia (>2 veces) entre los tratamientos (Figura 2B). De estos, 19 mostraron aumentos en el grupo tratado, y 10 de ellos mostraron disminución en el grupo tratado. Fibrobacter, Treponema, Lactobacillus y Olsenella se han reportado previamente en abscesos hepáticos en bovinos.
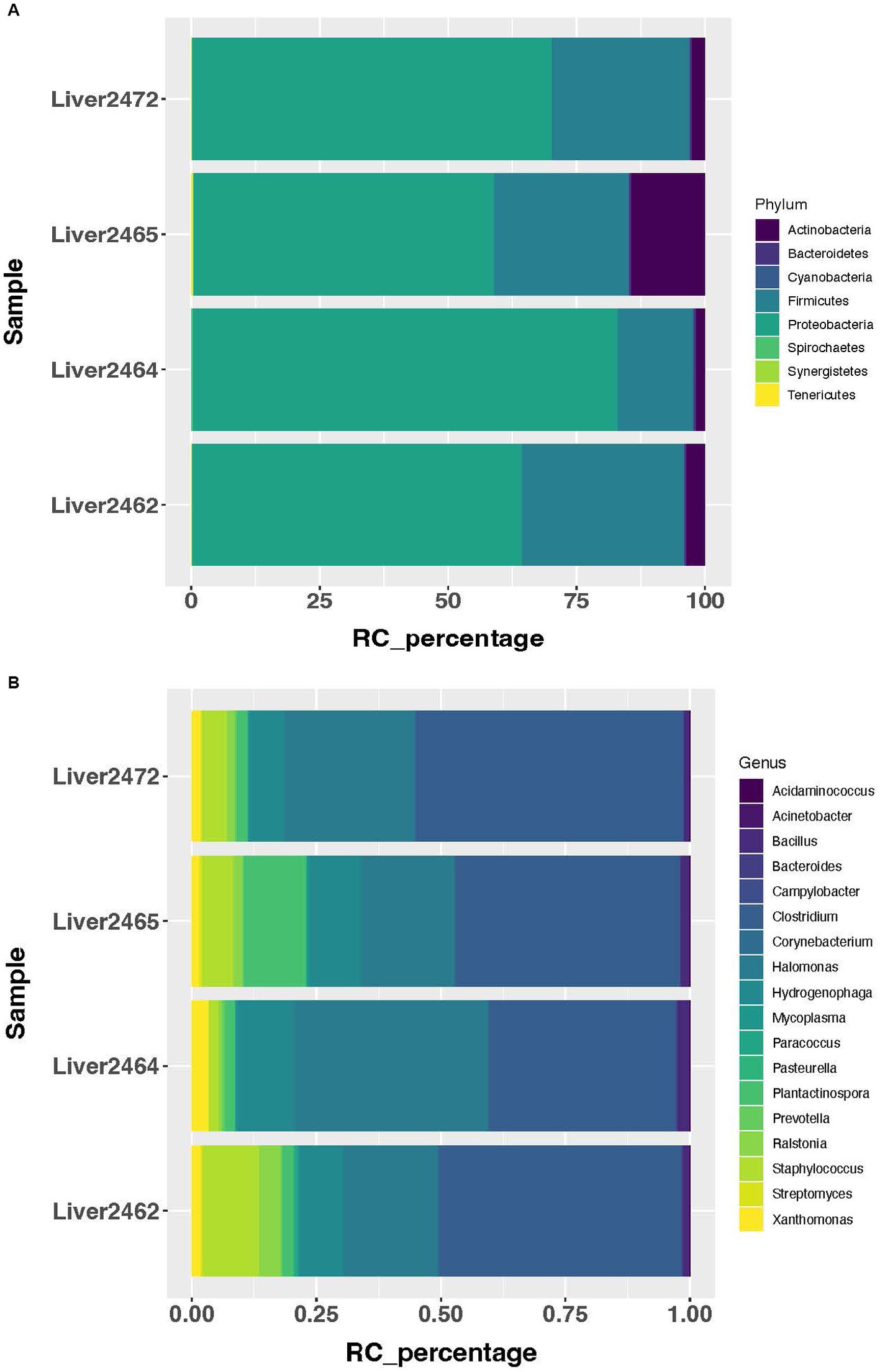 Figura 1. Taxones microbianos más abundantes identificados en el grupo tratado. (A) Filos microbianos más abundantes en el grupo tratado. (B) Géneros microbianos más abundantes en el grupo tratado.
Figura 1. Taxones microbianos más abundantes identificados en el grupo tratado. (A) Filos microbianos más abundantes en el grupo tratado. (B) Géneros microbianos más abundantes en el grupo tratado.
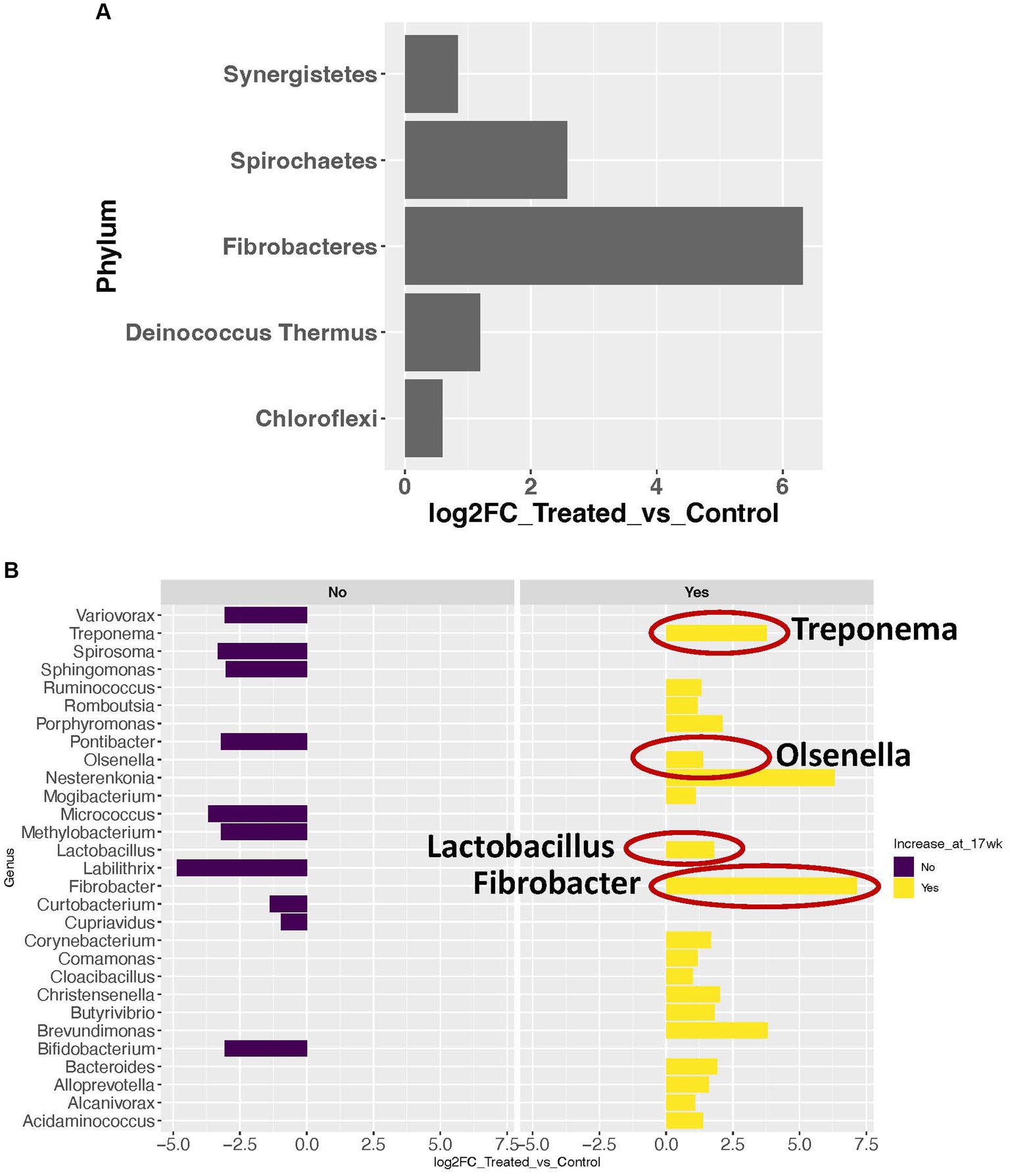 Figura 2. Taxones microbianos con cambios significativos en la abundancia entre los grupos tratados y control. (A) Aumento significativo de la abundancia en 5 filos. (B) Cambios significativos en la abundancia a nivel de género. Las barras de color púrpura oscuro representan los géneros con disminución significativa en abundancia en el grupo tratado. Las barras amarillas representan los géneros con un aumento significativo en abundancia en el grupo tratado. Los géneros con un círculo rojo son los reportados previamente en el hígado absceso en bovinos.
Figura 2. Taxones microbianos con cambios significativos en la abundancia entre los grupos tratados y control. (A) Aumento significativo de la abundancia en 5 filos. (B) Cambios significativos en la abundancia a nivel de género. Las barras de color púrpura oscuro representan los géneros con disminución significativa en abundancia en el grupo tratado. Las barras amarillas representan los géneros con un aumento significativo en abundancia en el grupo tratado. Los géneros con un círculo rojo son los reportados previamente en el hígado absceso en bovinos.
El análisis sPLS-DA reveló una clara separación en la estructura de la comunidad microbiana para el grupo control frente al grupo AC (Figura 3). Para identificar los taxones discriminantes para el grupo AC y control, el análisis sPLS-DA estimó que el 60% de la firma microbiana seleccionada en el componente 1 del sPLS-DA caracterizó el microbioma hepático de los animales del grupo control, que incluyó Buchnera, Enterococcus, Lactobacillus, Candidatus Carsonella, Plantactinospora y Sphingomonas. Por otro lado, la firma microbiana caracterizó el microbioma hepático del grupo tratado compuesto por Xanthomonas, Ruminococcus, Bifidobacterium y Paracoccus (Figura 4).
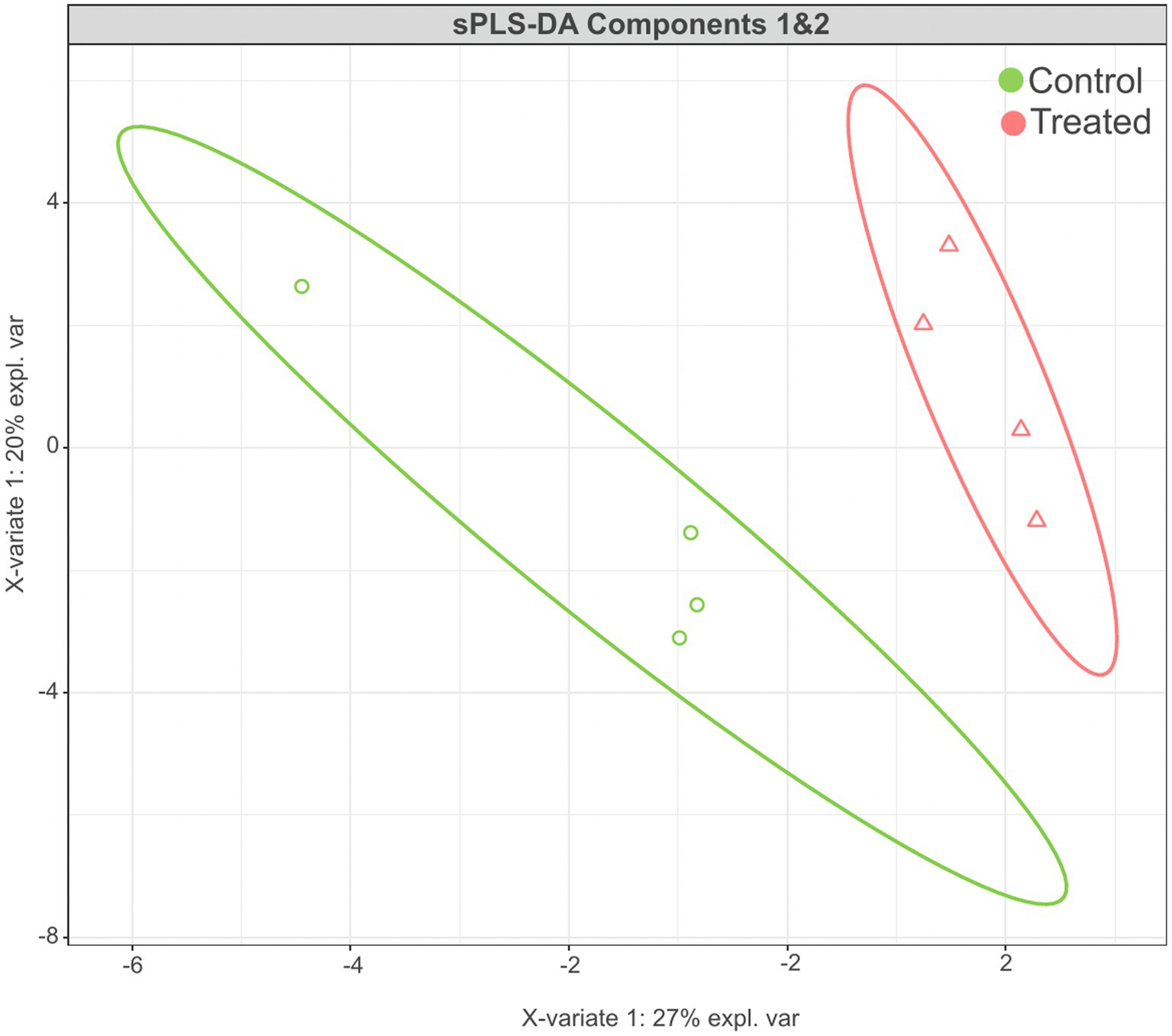 Figura 3. Resultados de sPLS-DA para el perfil de la comunidad microbiana a nivel de género en el hígado de los grupos control y tratados. Gráfico de puntuación de los dos primeros componentes, con un nivel de confianza del 95%.
Figura 3. Resultados de sPLS-DA para el perfil de la comunidad microbiana a nivel de género en el hígado de los grupos control y tratados. Gráfico de puntuación de los dos primeros componentes, con un nivel de confianza del 95%.
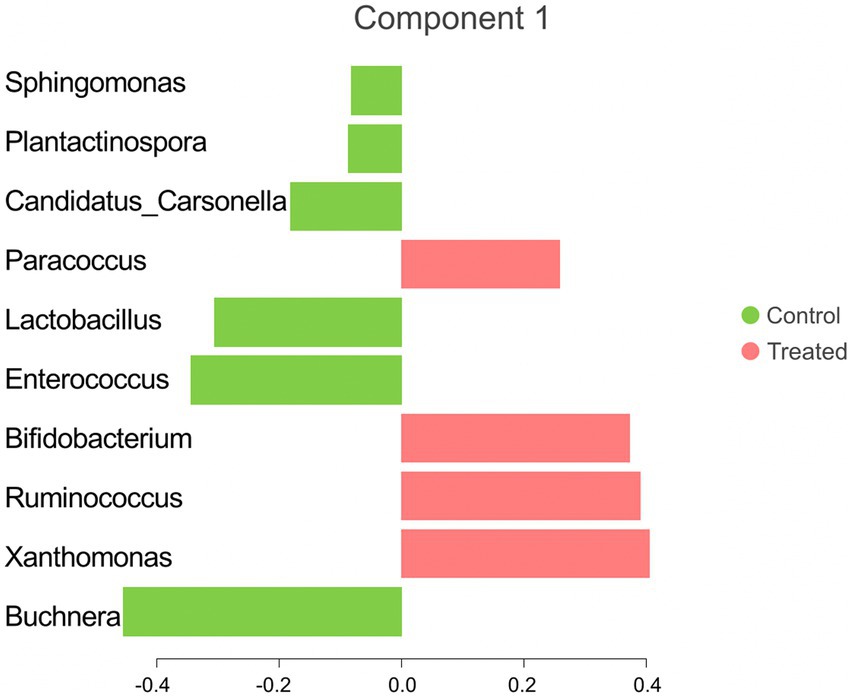 Figura 4. Contribución de los taxones microbianos clasificados de abajo (los más importantes) a los más altos. Los colores indican el grupo de tratamiento en el que la característica era más relevante.
Figura 4. Contribución de los taxones microbianos clasificados de abajo (los más importantes) a los más altos. Los colores indican el grupo de tratamiento en el que la característica era más relevante.
3.3. Análisis de asociación entre los genes del huésped y los taxones microbianos
Este análisis de asociación se realizó utilizando el recuento de lectura a nivel de gen, normalizado, publicado previamente de los genes del hígado del huésped, y el recuento de lectura normalizado a nivel de género de la comunidad microbiana del hígado, que se obtuvo para este informe. Usando un valor límite de valores de |r| > 0,7 y p < 0,001, se identificaron 139 genes hepáticos con asociación significativa con 38 géneros microbianos identificados en el hígado. El promedio de lecturas normalizadas de estos genes fue de 159,97 ± 34,02 (media ± S.E.). Los 4 genes más expresados son ITIH4, C4A, SPP2 y TMBIM6. Sus géneros microbianos asociados se enumeran en la Tabla 1. Los genes con 4 o más géneros microbianos asociados incluyeron ARRDC3, TTC7A, MOSPD1, FAM105B, CTNNAL1, CRELD2, GMPPA, NUCB2 y CSAD (Tabla 2). Los géneros microbianos asociados con 15 o más genes incluyeron Megasphaera, Ruminococcus, Dialister, Treponema, Fibrobacter y Prevotella. El análisis de GO utilizando los genes identificados determinó el enriquecimiento en las siguientes vías: GO:006090 (proceso metabólico de piruvato); GO:004736 (actividad piruvato carboxilasa). BTA04918 (síntesis de hormonas tiroideas). bta00510 (biosíntesis de N-glicanos). GO:0003756 (actividad de la proteína disulfuro isomerasa); GO:0005788 (luz del retículo endoplásmico); GO:0042470 (melanosoma); GO:0005788 (luz del retículo endoplásmico) y bta04141 (Procesamiento de proteínas en el retículo endoplásmico); bta00510 (biosíntesis de N-glicanos); GO:0016021 (componente integral de la membrana) (Tabla 3).
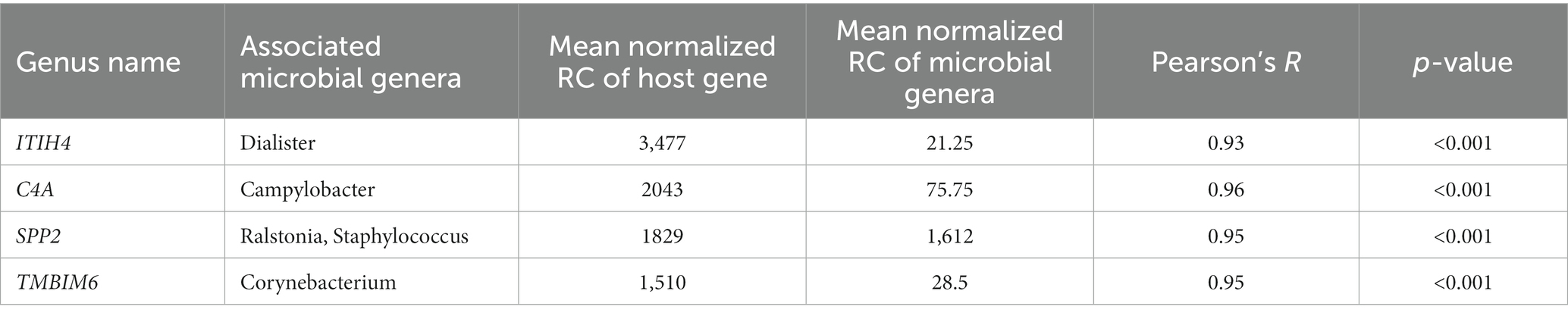 Tabla 1. La mayoría de los genes altamente expresados y sus géneros microbianos asociados en muestras tratadas y de control.
Tabla 1. La mayoría de los genes altamente expresados y sus géneros microbianos asociados en muestras tratadas y de control.
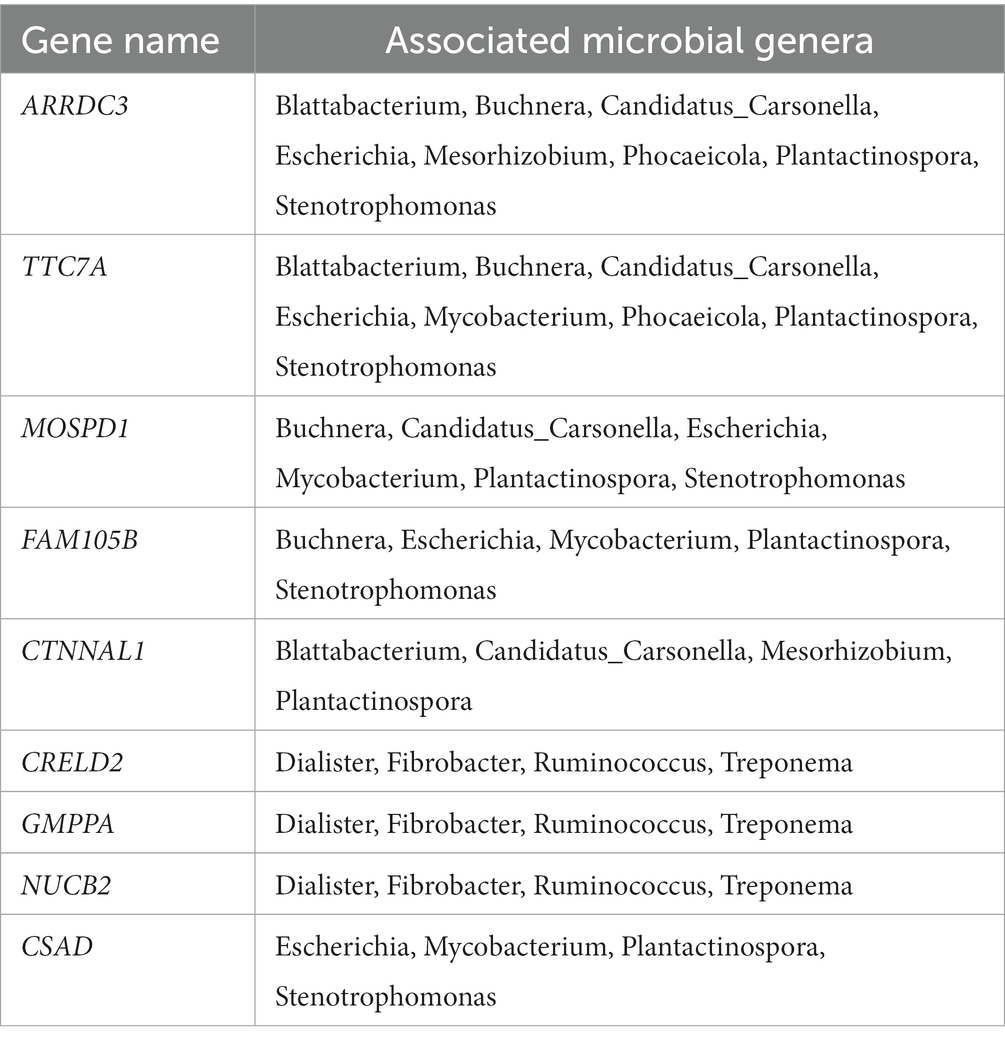 Tabla 2. Los genes del hígado del huésped identificaron una asociación significativa con sus microbios incrustados.
Tabla 2. Los genes del hígado del huésped identificaron una asociación significativa con sus microbios incrustados.
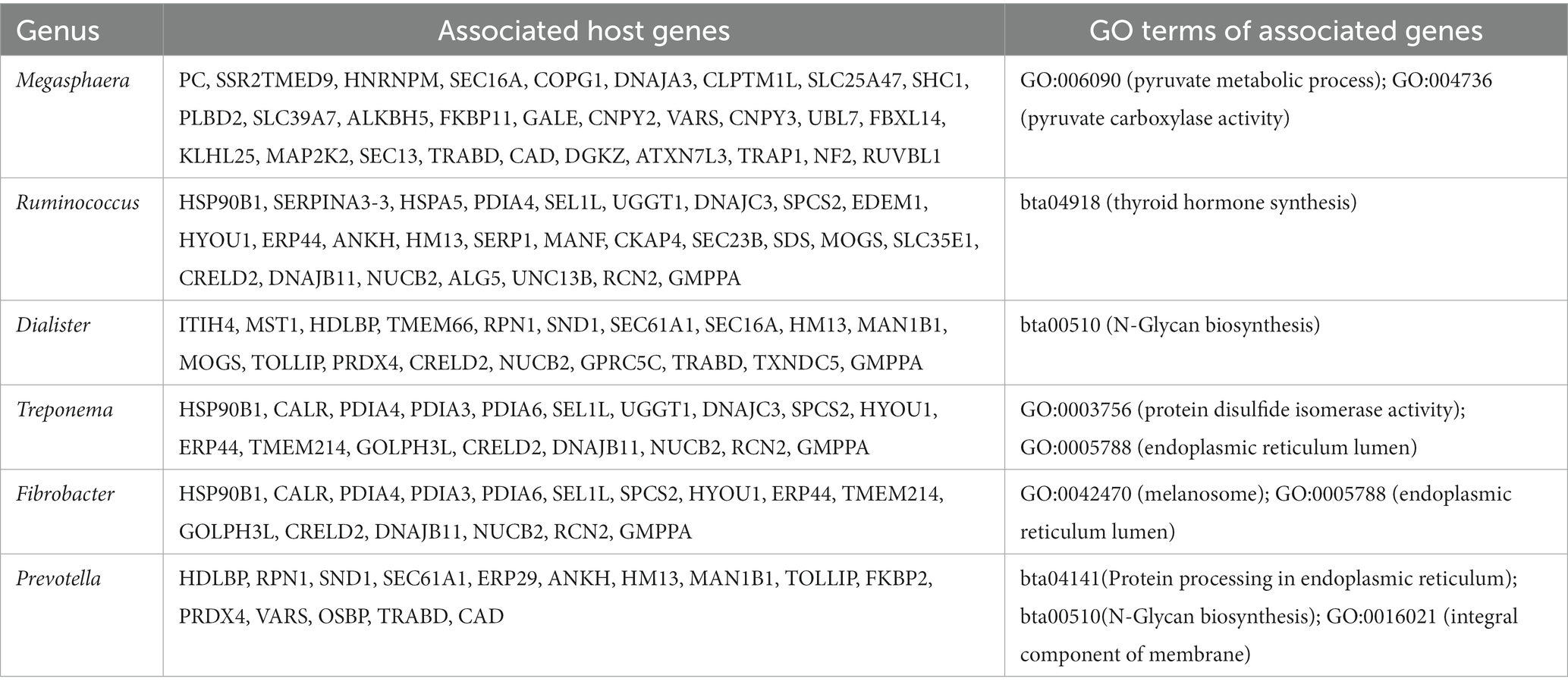 Tabla 3. Taxones microbianos hepáticos identificados con asociación significativa con genes hepáticos del huésped.
Tabla 3. Taxones microbianos hepáticos identificados con asociación significativa con genes hepáticos del huésped.
3.4. Comparación de la comunidad microbiana entre el hígado y el epitelio ruminal a las 17 semanas de edad
Los tejidos del epitelio ruminal recolectados a las 17 semanas de edad se incluyeron en esta comparación. En el epitelio ruminal colectado a las 17 semanas de edad, 57 géneros mostraron cambios significativos en la abundancia de transcritos de ARNr (p < 0,05 y cambio de pliegue >2). También se identificaron nueve de los géneros con un aumento significativo (p < 0.05 y cambio de pliegue >2) en abundancia en el grupo AC en tejidos hepáticos (Figura 5).
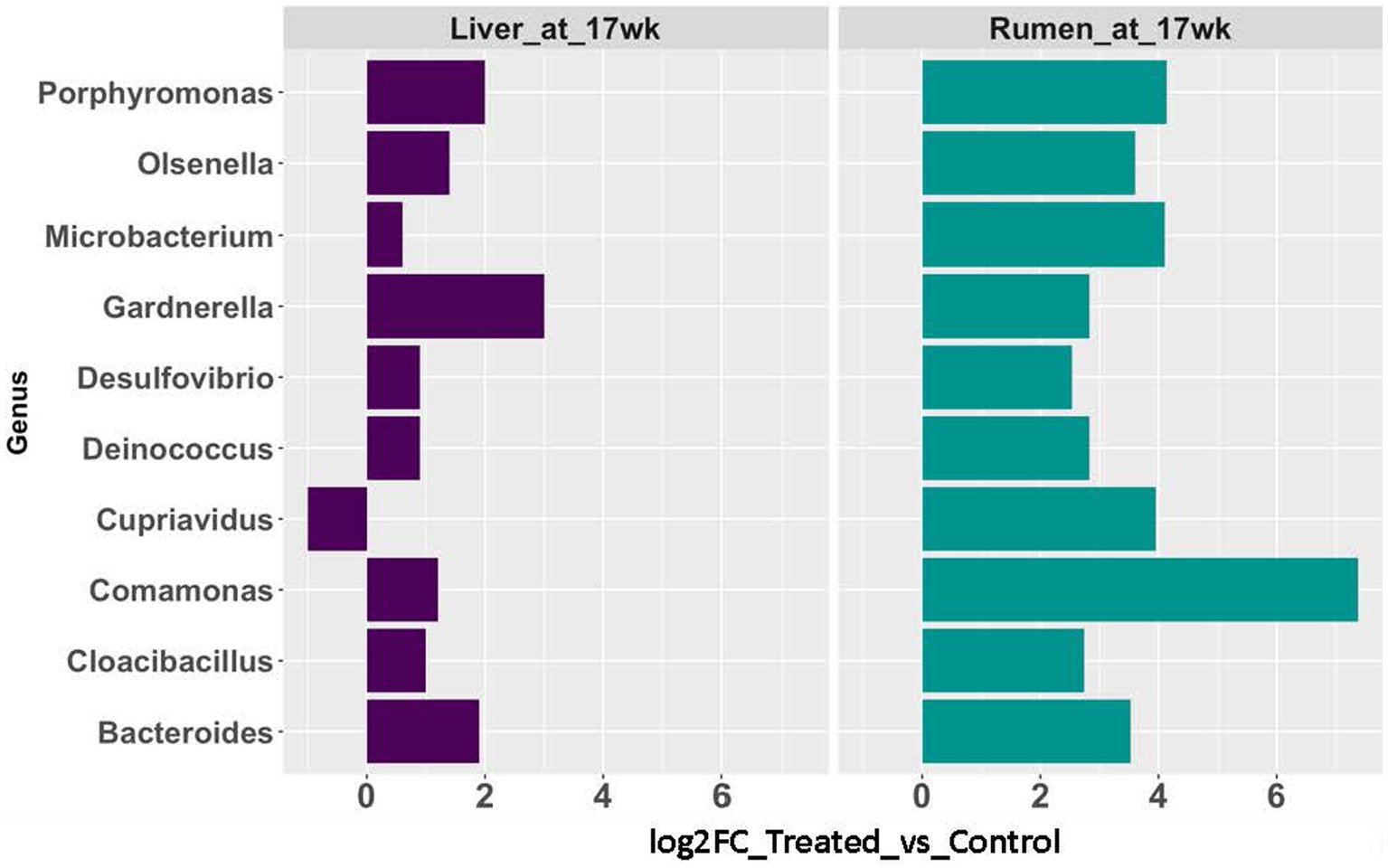 Figura 5. Cambios en la abundancia de taxones microbianos identificados tanto en el epitelio ruminal (barras azul turquesa) como en el hígado (barras púrpuras) a las 17 semanas de edad.
Figura 5. Cambios en la abundancia de taxones microbianos identificados tanto en el epitelio ruminal (barras azul turquesa) como en el hígado (barras púrpuras) a las 17 semanas de edad.
3.5. Identificación de géneros microbianos previamente identificados como discriminantes o muy abundantes en el hígado absceso
Para el análisis de prueba de concepto, se incluyeron en el análisis 17 géneros microbianos previamente reportados como taxones discriminantes para el hígado absceso en el ganado bovino o como altamente abundantes en el hígado absceso. En nuestro estudio se detecta una mayoría predominante (15 de 17) de ellas (Tabla 4). Varios de estos taxones mostraron una abundancia diferencial entre los grupos control y los tratados. En todas las muestras incluidas en este estudio, el promedio de RC de estos géneros osciló entre 4,75 y 2.128. En ambos grupos, los géneros más abundantes (RC > 200) incluyen Prevotella, Acinetobacter, Bacteroides, Campylobacter, Acinetobacter, Pseudomonas, Lactobacillus y Campylobacter. Los géneros con menor abundancia (RC < 100) incluyen Fusobacterium, Salmonella y Porphyromonas. El resto de los géneros no tienen recuentos de lectura detectables, o solo se detectaron en una de las muestras de cualquiera de los grupos de tratamiento.
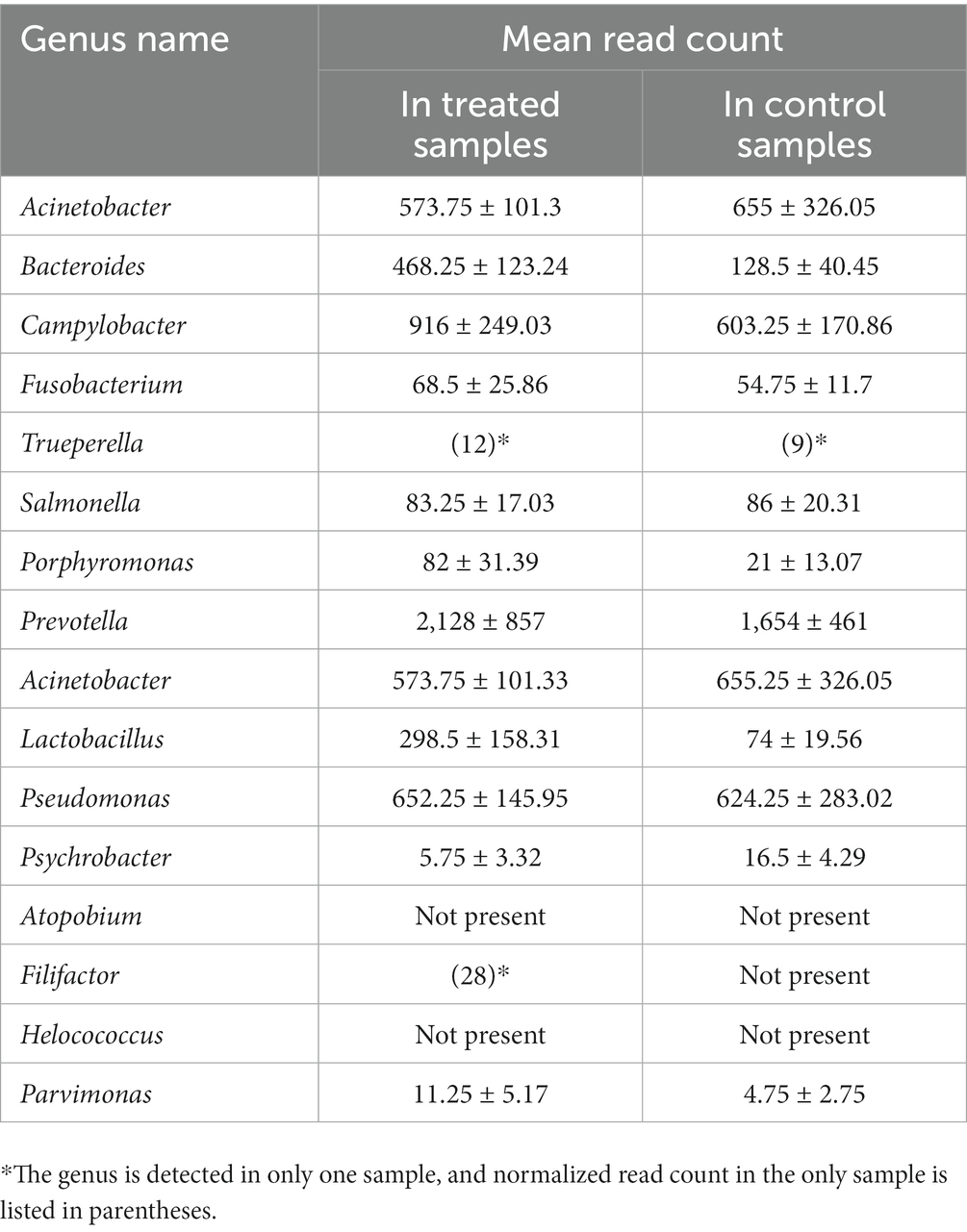 Tabla 4. Recuentos de lectura normalizados promedio para los géneros previamente reportados como altamente abundantes en abscesos hepáticos, comparando muestras tratadas y de control.
Tabla 4. Recuentos de lectura normalizados promedio para los géneros previamente reportados como altamente abundantes en abscesos hepáticos, comparando muestras tratadas y de control.
4. Discusiones
4.1. Genes hepáticos del huésped con asociación significativa con sus taxones microbianos
A través del análisis de asociación del nivel de expresión génica del huésped y la abundancia de taxones microbianos en el hígado, surgieron varias ideas valiosas. Se identificaron cuatro genes altamente expresados con asociación significativa (r > de Pearson 0,9 y p < 0,0001). Dos de ellos (ITIH4 y C4A) están implicados en la respuesta inflamatoria como se ha informado previamente. Además, se informó que la ITIH4 es un biomarcador inflamatorio en la infección bacteriana del torrente sanguíneo (55). A su asociación con la enfermedad inflamatoria intestinal (56). Este gen codifica una proteína de fase aguda y se expresa principalmente en el hígado. Se ha descrito su aumento significativo en el suero durante las fases agudas en pacientes humanos (57). En bovinos, varias proteínas de fase aguda se utilizan rutinariamente como marcadores inflamatorios. Incluyen la amiloide A sérica, la haptoglobina, la glicoproteína alfa-1-ácida y el inhibidor de la alfa-1-proteinasa (58-60). Y como proteína de fase aguda recientemente establecida, la concentración sérica de la proteína ITIH4 se correlacionó positivamente con la gravedad de la enfermedad de la mastitis y también se confirmó su mayor concentración en bovinos con infección por el virus respiratorio sincitial bovino (61). Curiosamente, el taxón microbiano asociado con este gen es Dialister, que fue reportado como el patógeno causal en los abscesos hepáticos humanos (62). Aunque no se ha establecido la relación causal entre Dialister y el absceso hepático en bovinos, ITIH4 se erige como un biomarcador de alto potencial a desarrollar para la detección temprana de la inflamación hepática o sanguínea inducida por bacterias en bovinos.
Para el análisis de la asociación entre la expresión de los genes hepáticos y la abundancia de taxones microbianos en el hígado, se revelaron ideas interesantes por la función de los genes y las vías GO en las que estaban involucrados. En el caso de los genes asociados con el mayor número de taxones microbianos, previamente habían informado de su papel en el metabolismo energético y lipídico, y en la regulación de la inflamación en el intestino y el hígado. Estos hallazgos sugieren que, a nivel de la fisiología del huésped, las respuestas significativas impulsadas por los cambios en la comunidad microbiana ya están en marcha antes de la expresión manifiesta de los signos clínicos de acidosis ruminal. Para los términos GO identificados, nuestro análisis indicó que la comunidad microbiana hepática puede contribuir significativamente a los cambios en la fisiología hepática resultantes de la acidosis inducida por el alimento. Esto se refleja en el enriquecimiento del componente celular (GO:0005788, luz del retículo endoplásmico; GO:0016021, componente integral de la membrana), metabolismo del piruvato (GO:006090, proceso metabólico del piruvato; GO:004736, actividad piruvato carboxilasa). Ambos enriquecimientos se identificaron previamente para los genes expresados diferencialmente en el hígado entre los grupos de tratamiento (36). Específicamente, el piruvato fue reportado como un tampón corrector de la acidosis intracelular (63). Además, se identificó un enriquecimiento de la vía de síntesis de hormonas tiroideas para los genes asociados con los microbios incrustados en el tejido. La hormona tiroidea baja, T3, fue reportada como un predictor de mal pronóstico en pacientes con absceso hepático piógeno (64). El hígado es importante para la excreción, el transporte, el metabolismo y el almacenamiento de hormonas tiroideas (65), aunque actualmente los estudios sobre la abundancia de hormonas tiroideas en el ganado bovino se centran principalmente en la gestación y la lactancia (66, 67). Nuestros datos sugirieron que la comunidad microbiana hepática puede afectar el perfil de hormonas tiroideas en el hígado en caso de acidosis metabólica hepática causada por acidosis ruminal inducida por el alimento.
4.2. Cambios simultáneos en la comunidad epitelial del rumen y microbiana hepática
Observamos cambios concurrentes en las comunidades microbianas tanto en el epitelio hepático como en el ruminal a las 17 semanas de edad, evidenciados por los 10 géneros con cambios significativos en la abundancia entre los grupos AC y control. Nueve de estos géneros mostraron la misma tendencia de cambios de expresión en ambos tipos de tejidos. En reportes previos de nuestro grupo, se identificaron lesiones significativas en los tejidos epiteliales del rumen a las 17 semanas de edad (14). En combinación con nuestra observación, es posible que la función de las uniones estrechas y de la barrera intestinal se vea comprometida antes de las 17 semanas de edad. Una vez comprometida, la barrera intestinal dañada abre la puerta a la translocación de microbios del rumen al hígado a través del sistema circulatorio. Nuestros hallazgos proporcionaron información sobre el transcriptoma ruminal del huésped y los cambios en la comunidad microbiana asociados tanto en el rumen como en el hígado como resultado de la acidosis prolongada en terneros posteriores al destete. Los cambios concurrentes en la abundancia de géneros microbianos tanto en el hígado como en el rumen a las 17 semanas de edad indicaron la posible translocación de microbios del epitelio ruminal al hígado. Dado el importante papel del hígado como órgano metabólico, nuestros hallazgos justifican futuras investigaciones sobre el papel del microbioma hepático en el desarrollo y la respuesta del huésped a la acidosis ruminal.
4.3. Presencia significativa de taxones microbianos previamente identificados y nuevos taxones en el hígado de terneros con acidosis inducida por piensos
Como investigación de prueba de concepto, verificamos la abundancia de transcritos de ARNr de 17 géneros microbianos previamente reportados como taxones discriminantes o altamente abundantes para el hígado absceso. La mayoría de ellos fueron detectados en nuestro estudio. Las fusobacterias se han considerado durante mucho tiempo el principal patógeno causal del absceso hepático en el ganado bovino, ya que este taxón microbiano se ha identificado sistemáticamente utilizando métodos basados en cultivos (50, 52, 53, 68) o un método basado en secuenciación de ADN basado en amplicones de ARNr de 16 s (33). Sin embargo, la limitación de cada uno de estos métodos puede dar lugar a una evaluación sesgada. En nuestro estudio, la presencia de transcripciones es sustancial para este taxón identificado por nuestro método basado en la secuenciación de ARN, aunque no parece ser el más abundante medido por el recuento de lecturas de transcripciones de ARNr. Para ser identificados con éxito por métodos basados en cultivos, los medios de cultivo deben ser compatibles con los microbios objetivo. Además, los microbios anaeróbicos requieren un entorno vacío de oxígeno, lo que dificulta el cultivo de microbios anaeróbicos en un entorno de laboratorio. La secuenciación basada en el ADN no puede diferenciar las células muertas o lisadas de las células vivas. A pesar de su abundancia predominante identificada en los estudios basados en amplicones de ARNr 16 s, no indica directamente que este género se transcriba activamente. Y debido a esta limitación, muchos géneros microbianos determinados como altamente abundantes por secuenciación basada en ADN pueden sufrir el ruido proveniente del ADN extraído de las células muertas.
Además de la identificación exitosa de muchos géneros microbianos previamente identificados en el hígado absceso, en nuestro estudio se detectaron muchos géneros microbianos nuevos con una abundancia significativa de transcritos de ARNr mediante nuestro enfoque de secuenciación del transcriptoma. Curiosamente, varios de los nuevos géneros identificados en nuestro estudio ya han sido reportados como patógenos causales potenciales en abscesos hepáticos humanos y otras enfermedades de alto impacto en el ganado. Por ejemplo, los géneros Pasteurella y Clostridium presentaron una alta abundancia en las muestras hepáticas de nuestro estudio (Tabla Suplementaria S2). Pasteurella sp. fue reportada previamente en absceso epidural de hígado, pulmón y columna vertebral en humanos (69, 70). Clostridium sp. (71, 72) y Pasteurella sp. (69) fueron reportados en abscesos hepáticos humanos y animales domésticos. Clostridium es una bacteria anaeróbica formadora de esporas. Su presencia se encuentra a menudo en el suelo, en los piensos o en el manurer (73). Una vez ingerido, el Clostridium puede proliferar en la mucosa intestinal, llegar al hígado a través del sistema circulatorio y persistir en las células fagocíticas (74). Cuando la dieta del animal cambia o experimenta lesiones en los tejidos, se crea una condición favorable para que las bacterias proliferen y se vuelvan patógenas causando infecciones en el hígado y otros órganos. Pasteurella sp. es altamente prevalente en poblaciones de animales domésticos y silvestres (74). La infección sintomática por Pasteurella fue considerada una enfermedad de alto impacto en el ganado, según la Organización Mundial de Sanidad Animal.2 Aunque no todos los nuevos taxones microbianos identificados en nuestro estudio mostraron cambios significativos entre los grupos de tratamiento, este nuevo repertorio de taxones microbianos transcritos activamente identificados en nuestro estudio allana el camino para futuros análisis en profundidad del microbioma hepático y su conexión con la acidosis inducida por piensos. Cabe destacar que el momento de la recolección de la muestra vale la pena realizar esfuerzos adicionales, ya que es posible que las transcripciones microbianas no se produzcan a un ritmo constante y un estudio más amplio que cubra múltiples puntos de tiempo podría ayudar a descubrir nuevos microbios causales del absceso hepático en el ganado.
Nuestro estudio y otros indican que la patogenia del absceso hepático es polimicrobiana. La patobiología de muchos de los taxones recién identificados en nuestro estudio aún no se ha dilucidado. Además, la mayoría de los trabajos publicados anteriormente se centran en el ganado adulto. Nuestro estudio es uno de los pocos que se centra en terneros jóvenes. Y lo que es más importante, nuestro estudio sugirió que, para capturar cambios significativos en la comunidad microbiana hepática, la secuenciación basada en transcriptomas puede ofrecer una nueva capa de conocimientos por su poder discriminatorio de capturar transcripciones transcritas activamente de células microbianas hepáticas incrustadas.
5. Conclusión
Este estudio investigó la comunidad microbiana incrustada en el tejido en el hígado en terneros con o sin acidosis inducida por alimento. En este estudio se identificaron muchos de los taxones microbianos previamente reportados en abscesos bovinos. Y lo que es más importante, al emplear la secuenciación del metatranscriptoma, nuestro estudio permitió vislumbrar muchos microbios nuevos que mostraron cambios significativos en la abundancia entre los grupos de tratamiento. El análisis de asociación entre la expresión génica del hígado del huésped y la abundancia de su comunidad microbiana sugirió que el microbioma hepático puede desempeñar un papel fundamental en la fisiología del hígado del huésped. Específicamente, se identificaron genes involucrados en la vía relacionada con la hormona tiroidea con una asociación significativa con los taxones microbianos en el hígado, aunque las respuestas de la hormona tiroidea anteriormente solo se investigaron en el ganado durante la gestación o la lactancia. Estos nuevos hallazgos enfatizan la necesidad de un análisis más profundo, que ayudará a pintar una imagen completa de la diversidad y el ensamblaje de los microbios incrustados en el tejido en el hígado, y sus roles fisiológicos en la acidosis metabólica en el hígado como resultado de la acidosis ruminal relacionada con la alimentación.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres de los repositorios y los números de acceso se pueden encontrar en: https://www.ncbi.nlm.nih.gov/bioproject; PRJNA948013, PRJNA962842.
Declaración ética
El estudio en animales fue aprobado por el Comité de Cuidado y Uso de Animales de la Universidad de Wisconsin-Madison. El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
WL concibió el experimento de secuenciación de ARN y analizó los datos de secuenciación del transcriptoma microbiano y del huésped. AL y BM procesaron los tejidos crudos, extrajeron el ARN total, realizaron el control de calidad del ARN y prepararon la biblioteca de secuenciación de ARN. PF realizó un análisis de la firma microbiana. WL y PF contribuyeron a la redacción del manuscrito. Todos los autores contribuyeron al artículo y aprobaron la versión enviada.
Financiación
El financiamiento para este experimento es proporcionado por los proyectos 5090-31000-026-000D y 5090-31000-028-000D del Servicio de Investigación Agrícola del USDA. La mención de cualquier marca comercial o producto patentado en este manuscrito no constituye una garantía del producto por parte del USDA o del Servicio de Investigación Agrícola y no implica su aprobación con exclusión de otros productos que también puedan ser adecuados.
Reconocimientos
Agradecemos a Sonia Gelsinger por diseñar la dieta de alimentación y realizar los ensayos de alimentación. Andrea Edwards ayudó con el procesamiento de la muestra de tejido. El equipo de la granja en el Centro de Investigación de Forrajes Lácteos de EE. UU. ayudó con el manejo diario de los terneros.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1193473/full#supplementary-material
Notas
Referencias
1. Khan, MA , Bach, A , Cansado, DM y von Keyserlingk, MAG . Revisión invitada: transición de leche a alimento sólido en novillas lecheras. J Lácteos Sci. (2016) 99:885–902. doi: 10.3168/jds.2015-9975
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Khan, MA , Cansado, DM y von Keyserlingk, MA . Revisión invitada: efectos de la ración de leche sobre la ingesta de alimento sólido, el destete y el rendimiento en novillas lecheras. J Lácteos Sci. (2011) 94:1071–81. doi: 10.3168/jds.2010-3733
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Guzmán, CE , Bereza-Malcolm, LT , De Groef, B y Franks, AE . Presencia de metanógenos, bacterias fibrolíticas y proteobacterias seleccionadas en el tracto gastrointestinal de terneros lecheros neonatos desde el nacimiento hasta las 72 horas. PLoS Uno. (2015) 10:133048. doi: 10.1371/journal.pone.0133048
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Jami, E , Israel, A , Kotser, A y Mizrahi, I . Explorando la comunidad bacteriana del rumen bovino desde el nacimiento hasta la edad adulta. ISME J. (2013) 7:1069–79. doi: 10.1038/ismej.2013.2
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Furman, O , Shenhav, L , Sasson, G , Kokou, F , Honig, H , Jacoby, S, et al. La estocasticidad, limitada por los efectos deterministas de la dieta y la edad, impulsa la dinámica de ensamblaje del microbioma ruminal. Nat Commun. (2020) 11:1904.
6. Cersosimo, LM , Radloff, W y Zanton, GI . La composición del inóculo microbiano y la edad del ternero lechero antes del destete alteran el entorno microbiano del rumen en desarrollo. Microbiol frontal. (2019) 10:1651. doi: 10.3389/fmicb.2019.01651
7. Parque, T , Cersosimo, LM , Li, W , Radloff, W y Zanton, GI . Administración ruminal antes del destete de comunidades bacterianas ruminales en forma de inóculos derivados del rumen enriquecidas diferencialmente y redes de co-ocurrencia de terneros lecheros posdestetados. Microbiol frontal. (2021) 12:625488. doi: 10.3389/fmicb.2021.625488
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Li, W , Edwards, A , Riehle, C , Cox, MS , Raabis, S , Skarlupka, JH, et al. Análisis transcriptómico del hígado del huésped y análisis del metatranscriptoma de la comunidad microbiana epimural ruminal en terneros jóvenes tratados con dosificación artificial del contenido ruminal de vaca donante adulta. Sci Rep. (2019) 9:790. doi: 10.1038/s41598-018-37033-4
9. Meale, SJ , Popova, M , Saro, C , Martín, C , Bernard, A , Lagree, M, et al. La intervención dietética temprana en la vida de los terneros lecheros da como resultado una reducción a largo plazo de las emisiones de metano. Sci Rep. (2021) 11:3003. doi: 10.1038/s41598-021-82084-9
10. Dias, J , Marcondes, MI , Motta de Souza, S , Cardoso da Mata, ESB , Fontes Noronha, M , Tassinari Resende, R, et al. Dinámica de la comunidad bacteriana a través del tracto gastrointestinal de los terneros lecheros durante el desarrollo previo al destete. Appl Environ Microbiol. (2018) 84:17. doi: 10.1128/AEM.02675-17
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Dill-McFarland, KA , Weimer, PJ , Interruptor, JD y Suen, G . La dieta influye en el desarrollo temprano de la microbiota en los terneros lecheros sin repercusiones a largo plazo en la producción de leche. Appl Environ Microbiol. (2019) 85:18. doi: 10.1128/AEM.02141-18
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Kehoe, SI , Dill-McFarland, KA , Interruptor, JD y Suen, G . Efectos de la inclusión de ensilado de maíz en dietas de terneros predestete. J Lácteos Sci. (2019) 102:4131–7. doi: 10.3168/jds.2018-15799
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Flatt, WP , Warner, RG y Loosli, JK . Influencia de los materiales purificados en el desarrollo del estómago de los rumiantes. J Lácteos Sci. (1958) 302:91138. doi: 10.3168/jds. S0022-0302(58)91138-X
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Gelsinger, S.L. , Coblentz, WK , Zanton, GI , Ogden, RK y Akins, MS . Efectos fisiológicos de la acidosis ruminal inducida por iniciadores en terneros antes, durante y después del destete. J Lácteos Sci. (2020) 103:2762–72. doi: 10.3168/jds.2019-17494
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Chishti, GA , Salfer, IJ , Suárez-Mena, FX , Harvatine, KJ y Heinrichs, AJ . Comunicación corta: relaciones entre la forma física de la avena en el iniciador, el pH ruminal y los ácidos grasos volátiles sobre la expresión hepática de genes implicados en el metabolismo y la inflamación en terneros lecheros. J Lácteos Sci. (2020) 103:439–46. doi: 10.3168/jds.2019-16296
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Toro, LS , Bush, LJ , Amigo, JD , Harris, B Jr y Jones, EW . Incidencia de paraqueratosis ruminal en terneros alimentados con diferentes raciones y su relación con la absorción de ácidos grasos volátiles. J Lácteos Sci. (1965) 48:1459–66. doi: 10.3168/jds. S0022-0302(65)88499-5
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Madera, KM , Palmer, SI , Steele, MA , Metcalf, JA y Penner, GB . Influencia de la edad y el destete en la permeabilidad del tracto gastrointestinal en terneros de toro Holstein. J Lácteos Sci. (2015) 98:7226–37. doi: 10.3168/jds.2015-9393
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. Kirschner, N , Houdek, P , Fromm, M , Moll, I y Brandner, JM . Las uniones estrechas forman una barrera en la epidermis humana. Eur J Cell Biol. (2010) 89:839–42. doi: 10.1016/j.ejcb.2010.07.010
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Gatt, M , Reddy, BS y Mac Fie, J . Artículo de revisión: translocación bacteriana en enfermos críticos: evidencia y métodos de prevención. Aliment Pharm ther. (2007) 25:741–57. doi: 10.1111/j.1365-2036.2006.03174.x
20. Sanders, DS . Integridad de la mucosa y función barrera en la patogénesis de lesiones tempranas en la enfermedad de Crohn. J Clin Pathol. (2005) 58:568–72. doi: 10.1136/jcp.2004.021840
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Anderson, RC , Cookson, AL , McNabb, WC , Parque, Z , McCann, MJ , Kelly, WJ, et al. Lactobacillus plantarum MB452 mejora la función de la barrera intestinal al aumentar los niveles de expresión de los genes implicados en la formación de uniones estrechas. BMC Microbiol. (2010) 10:316. doi: 10.1186/1471-2180-10-316
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Fasana, CF , Donelli, G , Uzzau, S , Kaper, JB , Margaretten, K , Ding, X, et al. La toxina Zonula occludens modula las uniones estrechas a través de la reorganización de la actina dependiente de la proteína quinasa C, in vitro. J Clin Invest. (1995). doi: 10.1172/JCI118114
23. Ewaschuk, JB , Díaz, H , Meddings, L , Diederichs, B , Dmytrash, A , Backer, J, et al. Los factores bioactivos secretados por Bifidobacterium infantis mejoran la función de barrera de las células epiteliales. Am J Physiol Gastrointest Liver Physiol. (2008) 295:G1025–34. doi: 10.1152/ajpgi.90227.2008
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Bansal, T , Alaniz, RC , Madera, TK y Jayaraman, A . La señal bacteriana indol aumenta la resistencia de las uniones estrechas de las células epiteliales y atenúa los indicadores de inflamación. Proc Natl Acad Sci U S A. (2010) 107:228–33. doi: 10.1073/pnas.0906112107
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Hamer, HM , Jonkers, D , Venema, K , Vanhoutvin, S , Troost, FJ y Brummer, RJ . Artículo de revisión: el papel del butirato en la función colónica. Aliment Pharmacol Ther. (2008) 27:104–19. doi: 10.1111/j.1365-2036.2007.03562.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Shinji Fukuda, HT , Hase, K , Oshima, K , Nakanishi, Y , Yoshimura, K , Tobe, T, et al. Las bifidobacterias pueden proteger de la infección enteropatógena a través de la producción de acetato. Naturaleza. (2011) 469:543–7. doi: 10.1038/nature09646
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Marrón, TR y Lawrence, TE . Asociación de anomalías hepáticas con el rendimiento y el valor de la clasificación de la canal. J Anim Sci. (2010) 88:4037–43. doi: 10.2527/jas.2010-3219
28. Reinhardt, C y Hubbert, M . Control de abscesos hepáticos en ganado de engorde: una revisión. Prof. Anim Sci. (2015) 31:101–8. doi: 10.15232/pas.2014-01364
30. Eastwood, LC , Boykin, CA , Harris, MK , Arnold, AN , Hale, DS , Kerth, CR, et al. National Beef Quality Audit-2016: Transportation, mobility, and harvest-floor assessments of targeted characteristics that affect quality and value of cattle, carcasss, and sub-products. Trad. Anim. Sci. (2017) 1:229–38. doi: 10.2527/tas2017.0029
31. Nagaraja, TG , Narayanan, SK , Stewart, GC y Chengappa, MM . Infecciones por Fusobacterium necrophorum en animales: patogénesis y mecanismos patogénicos. Anaerobio. (2005) 11:239–46. doi: 10.1016/j.anaerobe.2005.01.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Nagaraja, TG y Chengappa, MM . Abscesos hepáticos en ganado de engorde: una revisión. J Anim Sci. (1998) 76:287–98. doi: 10.2527/1998.761287x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Amachawadi, RG , Tom, WA , Hays, diputado , Fernando, SC , Hardwidge, Relaciones Públicas y Nagaraja, TG . Análisis de la comunidad bacteriana del material purulento de abscesos hepáticos de bovinos cruzados y novillos Holstein alimentados con dietas de engorde con o sin tilosina. J Anim Sci. (2021) 99:76. doi: 10.1093/jas/skab076
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Stotz, MK , Enrique, DD y Crossland, WL . Caracterización del ADN bacteriano identificado en tejido hepático bovino absceso y no absceso en el momento de la cosecha. J Anim Sci. (2021) 99:280. doi: 10.1093/jas/skab280
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Li, W , Gelsinger, S , Edwards, A , Riehle, C y Koch, D . Análisis del transcriptoma del epitelio ruminal y análisis del metatranscriptoma de la comunidad microbiana epimural ruminal en terneros jóvenes con acidosis inducida por alimento. Sci Rep. (2019) 9:4744. doi: 10.1038/s41598-019-40375-2
36. Li, W , Gelsinger, S , Edwards, A , Riehle, C y Koch, D . Cambios en el metatranscriptoma de la comunidad microbiana epimural ruminal y el transcriptoma hepático en terneros jóvenes con acidosis inducida por alimento. Sci Rep. (2019) 9:18967. doi: 10.1038/s41598-019-54055-8
37. Gelsinger, S.L. , Coblentz, WK , Zanton, GI , Ogden, RK y Akins, MS . Desaparición ruminal in situ y digestión de todo el tracto de los piensos iniciadores en terneros antes, durante y después del destete. J Lácteos Sci. (2019) 102:2196–206. doi: 10.3168/jds.2018-15551
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Kristensen, NB , Engbaek, M , Vestergaard, M y Harmon, DL . Nota técnica: técnica de canulación ruminal en terneros Holstein jóvenes: efectos de la canulación sobre la ingesta de alimento, la ganancia de peso corporal y el desarrollo ruminal a las seis semanas de edad. J Lácteos Sci. (2010) 93:737–42. doi: 10.3168/jds.2009-2488
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Dobin, A , Davis, CA , Schlesinger, F , Drenkow, J , Zaleski, C , Jha, S, et al. STAR: alineador universal ultrarrápido de RNA-seq. Bioinformática. (2013) 29:15–21. doi: 10.1093/bioinformatics/bts635
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Trapnell, C , Roberts, A , Goff, L , Pertea, G , Kim, D , Kelley, DR, et al. Análisis diferencial de expresión génica y de transcripción de experimentos de RNA-seq con TopHat y gemelos. Nat Protoc. (2012) 7:562–78. doi: 10.1038/nprot.2012.016
Resumen de PubMed | Texto completo de CrossRef | Google Académico
41. Kopylova, E , Noe, L y Touzet, H . SortMeRNA: filtrado rápido y preciso de ARN ribosómicos en datos metatranscriptómicos. Bioinformática. (2012) 28:3211–7. doi: 10.1093/bioinformatics/bts611
Resumen de PubMed | Texto completo de CrossRef | Google Académico
42. Quast, C , Pruesse, E , Yilmaz, P , Gerken, J , Schweer, T , Yarza, P, et al. El proyecto de base de datos de genes de ARN ribosómico SILVA: mejora del procesamiento de datos y herramientas basadas en la web. Ácidos nucleicos Res. (2013) 41:D590-6. doi: 10.1093/nar/gks1219
43. Burge, SW , Daub, J , Eberhardt, R , Tate, J , Barquista, L , Nawrocki, EP, et al. Rfam 11.0: 10 años de familias de ARN. Ácidos nucleicos Res. (2013) 41:D226-32. doi: 10.1093/nar/gks1005
44. Madera, DE y Salzberg, S.L. . Kraken: clasificación ultrarrápida de secuencias metagenómicas utilizando alineaciones exactas. Genoma Biol. (2014) 15:R46. doi: 10.1186/gb-2014-15-3-r46
45. Rohart, F , Gautier, B , Singh, A y Le Cao, KA . mixOmics: Un paquete de R para la selección de características ómicas y la integración de múltiples datos. PLoS Comput Biol. (2017) 13:e1005752. doi: 10.1371/journal.pcbi.1005752
46. Lê Cao, KA , Boitard, S y Besse, P . Análisis discriminante PLS disperso: selección de características biológicamente relevantes y representaciones gráficas para problemas multiclase. BMC Bioinformática. (2011) 12:253. doi: 10.1186/1471-2105-12-253
47. Jiao, X , Sherman, BT , Huang da, W , Stephens, R , Baseler, MW , Lane, HC, et al. DAVID-WS: un servicio web con estado para facilitar el análisis de listas de genes/proteínas. Bioinformática. (2012) 28:1805–6. doi: 10.1093/bioinformatics/bts251
Resumen de PubMed | Texto completo de CrossRef | Google Académico
48. Amor, MI , Huber, W y Anders, S . Estimación moderada del cambio de pliegue y la dispersión para los datos de RNA-seq con DESeq2. Genoma Biol. (2014) 15:550. doi: 10.1186/s13059-014-0550-8
49. Amachawadi, RG , Purvis, TJ , Lubbers, BV , Homm, JW , Maxwell, CL y Nagaraja, TG . Flora bacteriana de abscesos hepáticos en bovinos de carne cruzados y novillos Holstein alimentados con dietas de engorde con o sin tilosina. J Anim Sci. (2017) 95:3425–34. doi: 10.2527/jas.2016.1198
Resumen de PubMed | Texto completo de CrossRef | Google Académico
50. Nagaraja, TG , Beharka, AB , Chengappa, MM , Carroll, LH , Raun, AP , Laudert, SB, et al. Flora bacteriana de abscesos hepáticos en bovinos de corral de engorde alimentados con tilosina o sin tilosina. J Anim Sci. (1999) 77:973–8. doi: 10.2527/1999.774973x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
51. Amachawadi, RG y Nagaraja, TG . Primer reporte de aislamiento anaeróbico de Salmonella enterica a partir de abscesos hepáticos de bovinos de corral de engorde. J Clin Microbiol. (2015) 53:3100–1. doi: 10.1128/JCM.01111-15
Resumen de PubMed | Texto completo de CrossRef | Google Académico
52. Lechtenberg, KF , Nagaraja, TG , Leipold, HW y Chengappa, MM . Estudios bacteriológicos e histológicos de abscesos hepáticos en bovinos. Am J Vet Res. (1988) 49:58–62.
53. Bronceado, ZL , Nagaraja, TG y Chengappa, MM . Infecciones por Fusobacterium necrophorum: factores de virulencia, mecanismo patogénico y medidas de control. Vet Res Commun. (1996) 20:113–40. doi: 10.1007/BF00385634
Resumen de PubMed | Texto completo de CrossRef | Google Académico
54. Pinnell, L , Whitlow, CW , Huebner, K , Bryant, T , Martín, J , Belk, KE, et al. No todos los abscesos hepáticos son iguales: el impacto de la tilosina y las alternativas a los antibióticos en las comunidades microbianas de abscesos hepáticos bovinos y un primer vistazo a las comunidades dominadas por Bacteroidetes. Microbiol frontal. (2022) 13:882419. doi: 10.3389/fmicb.2022.882419
Resumen de PubMed | Texto completo de CrossRef | Google Académico
55. Ma, YT , Li, RB , Wang, JA , Jiang, WC , Yuan, XZ , Cui, JY, et al. ITIH4, como biomarcador de inflamación, aumenta principalmente en la infección bacteriana del torrente sanguíneo. Citoquina. (2021) 138:155377. doi: 10.1016/j.cyto.2020.155377
Resumen de PubMed | Texto completo de CrossRef | Google Académico
56. Wen, N , Zhao, N , Xu, H , Zhao, Y y Ma, J . Inhibidor sérico de la cadena pesada 4 de la inter-alfa-tripsina en pacientes con enfermedad inflamatoria intestinal: correlación con el riesgo de enfermedad, la inflamación, la actividad y su variación después del tratamiento. Ir J Med Sci. (2022) 191:2105–11. doi: 10.1007/s11845-021-02837-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
57. Piñeiro, M , Álava, MA , González-Ramón, N , Osada, J , Lasierra, P , Larrad, L, et al. La concentración sérica de ITIH4 aumenta durante los procesos de fase aguda en pacientes humanos y está regulada al alza por la interleucina-6 en las células HepG2 del hepatocarcinoma. Biochem Bioph Res Co. (1999) 263:224–9. doi: 10.1006/bbrc.1999.1349
Resumen de PubMed | Texto completo de CrossRef | Google Académico
58. Conner, JG , Eckersall, PD , Wiseman, A , Aitchison, TC y Douglas, TA . Respuesta de fase aguda bovina después de la inyección de trementina. Res Vet Sci. (1988) 44:82–8. doi: 10.1016/0034-5288(88)90018-5
Resumen de PubMed | Texto completo de CrossRef | Google Académico
59. Hirvonen, J , Pyorala, S y Jousimies-Somer, H . Respuesta de fase aguda en novillas con mastitis inducida experimentalmente. J Dairy Res. (1996) 63:351–60. doi: 10.1017/S0022029900031873
Resumen de PubMed | Texto completo de CrossRef | Google Académico
60. Horadagoda, A , Eckersall, PD , Hodgson, JC , Gibbs, HA y Moon, GM . Respuestas inmediatas en concentraciones séricas de Tnf-alfa y proteínas de fase aguda a la infección por Pasteurella-Haemolytica A1 en terneros. Res Vet Sci. (1994) 57:129–32. doi: 10.1016/0034-5288(94)90094-9
Resumen de PubMed | Texto completo de CrossRef | Google Académico
61. Piñeiro, M , Andrés, M , Iturralde, M , Carmona, S , Hirvonen, J ITIH4 (cadena pesada 4 inhibidora de la inter-alfa-tripsina) es una nueva proteína de fase aguda aislada del ganado bovino durante la infección experimental. Infectar a Immun. (2004) 72:3777–82. doi: 10.1128/IAI.72.7.3777-3782.2004
Resumen de PubMed | Texto completo de CrossRef | Google Académico
62. Soeiro, C , Quilici, IR , Legoff, A , Oussalah, MB , Morin, M , Alauzet, C, et al. Absceso hepático debido a neumosintes de Dialister: reporte de un caso. Anaerobio. (2019) 59:35–7. doi: 10.1016/j.anaerobe.2019.05.006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
63. Zhou, FQ . El piruvato en la corrección de la acidosis intracelular: una base metabólica como novedoso tampón superior. Am J Nephrol. (2005) 25:55–63. doi: 10.1159/000084141
Resumen de PubMed | Texto completo de CrossRef | Google Académico
64. Xu, J y Wang, L . Síndrome de T3 baja como predictor de mal pronóstico en pacientes con absceso hepático piógeno. Endocrinol frontal. (2019) 10:541. doi: 10.3389/fendo.2019.00541
65. Sheridan, P . Las hormonas tiroideas y el hígado. Clin Gastroenterol. (1983) 12:797–818. doi: 10.1016/S0300-5089(21)00606-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
66. Fazio, E , Bionda, A , Chiofalo, V , Crepaldi, P , Lopreiato, V , Medica, P, et al. Respuestas adaptativas de las hormonas tiroideas, la insulina y la glucosa durante la gestación y la lactancia en vacas lecheras. Animales. (2022) 12:395. doi: 10.3390/ani12111395
Resumen de PubMed | Texto completo de CrossRef | Google Académico
67. Steinhoff, L , Jung, K , Meyerholz, MM , Heidekorn-Dettmer, J , Hoedemaker, M y Schmicke, M . Perfiles de hormonas tiroideas y evaluación de la TSH durante la gestación temprana y el período de transición en vacas lecheras. Teriogenología. (2019) 129:23–8. doi: 10.1016/j.theriogenology.2019.01.023
Resumen de PubMed | Texto completo de CrossRef | Google Académico
68. Lechtenberg, KF , Nagaraja, TG y Chengappa, MM . Susceptibilidad antimicrobiana de Fusobacterium necrophorum aislado de abscesos hepáticos bovinos. Am J Vet Res. (1998) 59:44–7.
69. Goussard, P , Gie, RP , Steyn, F , Rossouw, GJ y Kling, S . Absceso pulmonar y hepático de Pasteurella multocida en un niño inmunocompetente. Pediatr Pulmonol. (2006) 41:275–8. doi: 10.1002/ppul.20327
Resumen de PubMed | Texto completo de CrossRef | Google Académico
70. Oh, K , Inoue, T , Saito, T , Nishio, C y Konishi, H . Absceso epidural espinal causado por Pasteurella multocida que imita disección aórtica: reporte de un caso. BMC infecta dis. (2019) 19:448. doi: 10.1186/s12879-019-4097-x
71. Paasch, C , Wilczek, S y Strik, MW . Absceso hepático y sepsis causada por Clostridium perfringens y Klebsiella oxytoca. Int J Surg Case Rep. (2017) 41:180–3. doi: 10.1016/j.ijscr.2017.10.033
Resumen de PubMed | Texto completo de CrossRef | Google Académico
72. Derecho, ST y Lee, MK . Mujer de mediana edad con un absceso hepático piógeno causado por Clostridium perfringens. Mundo J Hepatol. (2012) 4:252–5. doi: 10.4254/wjh.v4.i8.252
Resumen de PubMed | Texto completo de CrossRef | Google Académico
74. Navarro, MA y Uzal, FA . Patobiología y diagnóstico de la hepatitis clostridial en animales. J Vet Diagn Investig. (2020) 32:192–202. doi: 10.1177/1040638719886567
Resumen de PubMed | Texto completo de CrossRef | Google Académico
75. Wilson, BA y Ho, M . Pasteurella multocida: de la zoonosis a la microbiología celular. Clin Microbiol Rev. (2013) 26:631–55. doi: 10.1128/CMR.00024-13
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: acidosis inducida por alimentación, microbioma hepático, transcriptoma hepático, secuenciación de ARN, terneros jóvenes
Cita: Li W, Larsen A, Murphy B y Fregulia P (2023) Comunidad microbiana hepática y transcriptoma del huésped asociado en terneros con acidosis inducida por alimento. Frente. Vet. Sci. 10:1193473. doi: 10.3389/fvets.2023.1193473
Recibido: 24 de marzo de 2023; Aceptado: 26 de septiembre de 2023;
Publicado: 23 octubre 2023.
Editado por:
Haoyu Liu, Universidad de Yangzhou, China
Revisado por:
Muhammad Zahoor Khan, Universidad de Agricultura, Pakistán
Stafford Vigors, University College Dublin, Irlanda
Derechos de autor © 2023 Li, Larsen, Murphy y Fregulia. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY)
*Correspondencia: Wenli Li, wenli.li@usda.gov
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía


