El análisis del transcriptoma completo revela interacciones patógeno-huésped específicas

El análisis del transcriptoma completo revela interacciones patógeno-huésped específicas de virulencia en la placenta en la neosporosis bovina
 Pilar Horcajo1†,
Pilar Horcajo1†,  Montserrat Coronado2†,
Montserrat Coronado2†,  Iván Pastor-Ferández1,
Iván Pastor-Ferández1,  Esther CollantesFernández1,
Esther CollantesFernández1,  Laura Rico-San Román1,
Laura Rico-San Román1,  Armando Reyes-Palomares2* y
Armando Reyes-Palomares2* y  Luis-Miguel Ortega-Mora1*
Luis-Miguel Ortega-Mora1*- 1SALUVET, Departamento de Sanidad Animal, Facultad de Ciencias Veterinarias, Universidad Complutense de Madrid, Madrid, España
- 2Departamento de Bioquímica y Biología Molecular, Facultad de Ciencias Veterinarias, Universidad Complutense de Madrid, Madrid, España
La investigación sobre la neosporosis bovina ha logrado hitos relevantes, pero los mecanismos subyacentes a la aparición de la muerte fetal o la protección contra la muerte fetal siguen sin estar claros. En un estudio reciente, las placentas de novillas desafiadas con el aislado de alta virulencia Nc-Spain7 exhibieron necrosis focal e infiltrados inflamatorios tan pronto como 10 días después de la infección (dpi), aunque la detección del parásito fue mínima. Estas lesiones fueron más frecuentes a 20 dpi, coincidiendo con mayores tasas de detección de parásitos y la ocurrencia de muerte fetal en algunos animales. En contraste, tales lesiones no se observaron en placentas de animales infectados con el aislado de baja virulencia Nc-Spain1H, donde el parásito se detectó solo en la placenta de un animal a 20 dpi. Este trabajo tuvo como objetivo estudiar qué mecanismos se activan en las placentas (carúnculas y cotiledones) de estas novillas preñadas en etapas tempranas de la infección (10 y 20 dpi) a través del análisis del transcriptoma completo. En las carúnculas, la infección con el aislado de alta virulencia provocó una fuerte respuesta proinflamatoria a 10 dpi. Este efecto no se observó en novillas infectadas con el aislado de baja virulencia, donde la señalización de IL-6/JAK/STAT3 y la señalización de TNF-alfa a través de las vías NF-κB se regularon a la baja. Curiosamente, la expresión de los genes diana E2F, relacionados con la restricción de la respuesta inflamatoria, fue mayor en estos animales. A 20 dpi, se detectaron firmas genéticas proinflamatorias más pronunciadas en novillas infectadas con el aislado de alta virulencia, siendo más intensas en novillas portadoras de fetos muertos. Sin embargo, el aislado de baja virulencia continuó sin activar la respuesta proinflamatoria. En los cotiledones, la respuesta a la infección con el aislado de alta virulencia fue similar a la observada en las carúnculas; Sin embargo, el aislado de baja virulencia indujo señales proinflamatorias leves a 20 dpi. Finalmente, un análisis deconvolucional de las firmas genéticas de ambos tejidos placentoma reveló una fracción marcadamente mayor de asesinos naturales activados, macrófagos M1 y células T CD8 + para el aislado de alta virulencia. Por lo tanto, nuestro análisis transcriptómico apoya la hipótesis de que una respuesta inmune intensa probablemente desencadenada por la multiplicación del parásito podría ser un contribuyente clave al aborto. Se requieren más estudios para determinar los efectores del parásito que gobiernan las distintas interacciones de los aislados de alta y baja virulencia con el huésped, lo que podría ayudar a dilucidar los procesos moleculares subyacentes a la patogénesis de la neosporosis en el ganado.
1 Introducción
Neospora caninum es un parásito apicomplejo responsable de una enfermedad infecciosa mundial que causa fallas reproductivas en el ganado, llamada neosporosis bovina. A pesar de las importantes pérdidas económicas debidas a la neosporosis bovina, todavía se sabe muy poco sobre los mecanismos moleculares que desencadenan el aborto y la muerte fetal en vacas infectadas con N. caninum (1, 2). Se han propuesto dos mecanismos principales para determinar el resultado de la gestación durante la infección: (i) lesiones fetales y/o placentarias resultantes de la multiplicación del parásito y (ii) el cambio de la respuesta antiinflamatoria Th2 predominante durante la gestación hacia una respuesta proinflamatoria Th1 (3). Sin embargo, otros factores relacionados con el parásito (variabilidad biológica intraespecífica, estadio del parásito) o con el huésped (madre y estado inmunológico fetal) tienen una marcada influencia en la dinámica de la infección parasitaria. Este escenario multifactorial dificulta la descripción de los mecanismos que podrían desencadenar el aborto (por ejemplo, deterioro temprano de las respuestas inmunes locales) o comprometer la protección fetal (por ejemplo, expresión de factores que modulan los procesos antiinflamatorios y la remodelación tisular) (4, 5).
En estudios anteriores, hemos intentado ilustrar las complejas interacciones huésped-parásito que ocurren durante la infección a través de PCR cuantitativa, RNA-seq y enfoques proteómicos utilizando líneas celulares placentarias bovinas específicas y macrófagos cultivados in vitro (6-9). Aunque la información proporcionada por estos trabajos sugiere diferentes formas de interacción dependiendo del aislado, descuida las intrincadas relaciones entre las diferentes poblaciones de células placentarias y la presión inmune impuesta por las células inmunes residentes. En los rumiantes, las principales funciones placentarias ocurren en los placentomos. Estos están formados por los compartimentos fetal (cotiledón) y materno (carúncula), cuyos sistemas circulatorios están físicamente separados, lo que agrega cierta complejidad a la respuesta desarrollada en la placenta. Tal complejidad también se ve incrementada por la actividad de las células inmunes innatas como los macrófagos, que podrían determinar la resistencia contra la infección por N. caninum debido a su capacidad para detectar, fagocitar y eliminar parásitos (10).
Estas limitaciones se han eludido parcialmente en los últimos años a través de ensayos in vivo realizados en novillas preñadas (1). Estas infecciones experimentales han demostrado que el resultado de la infección depende en gran medida del momento de la gestación cuando ocurre la infección. En resumen, el ganado preñado desafiado en el primer término de gestación (65-70 días) exhibe una muerte fetal rápida con una diseminación generalizada del parásito en la placenta (11-15), mientras que los infectados a mediados de la gestación (110-140 días de gestación) pueden mostrar fetopatía o el nacimiento de terneros infectados congénitamente (15-17). Por último, cuando la infección ocurre en el último tercio de la gestación, todos los terneros nacen sanos, pero infectados congénitamente (12, 15).
Teniendo en cuenta que los animales naturalmente infectados pueden dar a luz animales sanos pero infectados congénitamente o abortar, y que el aborto generalmente ocurre en el segundo trimestre de gestación (18, 19), los modelos de mitad de gestación se consideran una herramienta adecuada para el estudio de la patogénesis de la neosporosis bovina. Recientemente, y utilizando novillas gestantes desafiadas experimentalmente en el día 110 de gestación, encontramos claras diferencias en la dinámica temprana de la infección por N. caninum –10 y 20 días post-infección (dpi)– entre dos aislados diferentes, el altamente virulento Nc-Spain7 y el Nc-Spain1H de baja virulencia (20). Apenas 10 días después de la infección, las placentas de novillas desafiadas con el aislado de alta virulencia exhibieron necrosis focal e infiltrados inflamatorios, aunque se detectaron parásitos solo el 25% de los animales. Estas lesiones fueron más frecuentes a 20 dpi, coincidiendo con mayores tasas de detección de parásitos y la ocurrencia de muerte fetal en el 40% de los animales. Por el contrario, las placentas de animales infectados con el aislado de baja virulencia Nc-Spain1H no mostraron lesiones ni muerte fetal a 10 o 20 dpi, y el parásito se detectó solo en el 20% de los animales a 20 dpi (20). Los resultados previos que evaluaron la respuesta inmune local en estos animales mostraron que el aislado de baja virulencia desencadenó la expresión de citoquinas proinflamatorias, citoquinas antiinflamatorias y otros factores involucrados en el mantenimiento de la integridad de la ECM a 10 dpi. Sin embargo, a medida que la infección progresó (20 dpi), se observó una respuesta proinflamatoria basada en Th1 más fuerte en animales infectados por el aislado de alta virulencia, que fue más fuerte para algunas citoquinas como IL-1 y TNF-α en placentomas de animales portadores de fetos no viables (8). La placenta juega un papel clave en la patogénesis de la neosporosis: por un lado, actúa como una barrera física e inmunológica contra la infección, pero por otro lado, es capaz de desencadenar el aborto a través de mecanismos inmunomediados (5). De hecho, estudios previos que analizaron la expresión de algunas citoquinas en la interfaz materno-fetal después de la infección por N. caninum, mostraron la regulación positiva de citoquinas proinflamatorias (por ejemplo, IFN, IL-1, TNF), a menudo asociadas a la muerte fetal, y también de citoquinas Th12 y Treg (IL-2, IL-4), que con frecuencia se asocian a la supervivencia fetal (10-13, 15, 21). Esta estimulación inmune es más acentuada en la gestación temprana en el lado materno de la placenta, pero en infecciones posteriores a la gestación, la respuesta aumenta en el lado fetal de la placenta (22, 13, 21). La mayor parte de los trabajos anteriores se limitan al estudio de un pequeño número de citoquinas, por lo tanto, para descifrar los mecanismos desencadenados durante las interacciones tempranas huésped-parásito que podrían estar involucradas en el resultado final de la infección, analizamos todo el transcriptoma de carúnculas y cotiledones de animales infectados experimentalmente con aislados de N. caninum de alta (Nc-Spain22) y baja virulencia (Nc-Spain7H) utilizando una técnica de alto rendimiento para la secuenciación de ARN (RNA-seq).
2 Materiales y métodos
2.1 Declaración ética
Todos los protocolos con animales fueron aprobados por el Comité Ético de Investigación Animal del Principado de Asturias, España (número de referencia PROAE 25/2016), siguiendo los procedimientos descritos en las legislaciones española y comunitaria (Ley 32/2007, R.D. 53/2013 y Directiva 2010/63/UE del Consejo). Todos los animales fueron manejados en estricta conformidad con las buenas prácticas clínicas y se hicieron todos los esfuerzos para minimizar el sufrimiento.
2.2 Animales, diseño experimental y recolección de muestras
El diseño experimental ha sido descrito en detalle previamente (20). Brevemente, 24 novillas asturianas gestantes se dividieron en 3 grupos, dos grupos de 9 vacas que fueron inoculadas por vía intravenosa con 107 taquizoítos de los aislados Nc-Spain1H o Nc-Spain7 a los 110 días de gestación y un grupo control de 6 vacas que fueron inoculadas con solución salina tamponada con fosfato al mismo tiempo. La mitad de los animales (3 del grupo control y 4 de cada grupo infectado) fueron sacrificados a 10 dpi, y los animales restantes (3 del grupo control y 5 de cada grupo infectado) fueron sacrificados a 20 dpi (Figura 1A). En este último momento post-infección, se observó muerte fetal en 2 novillas infectadas con el aislado Nc-Spain7.
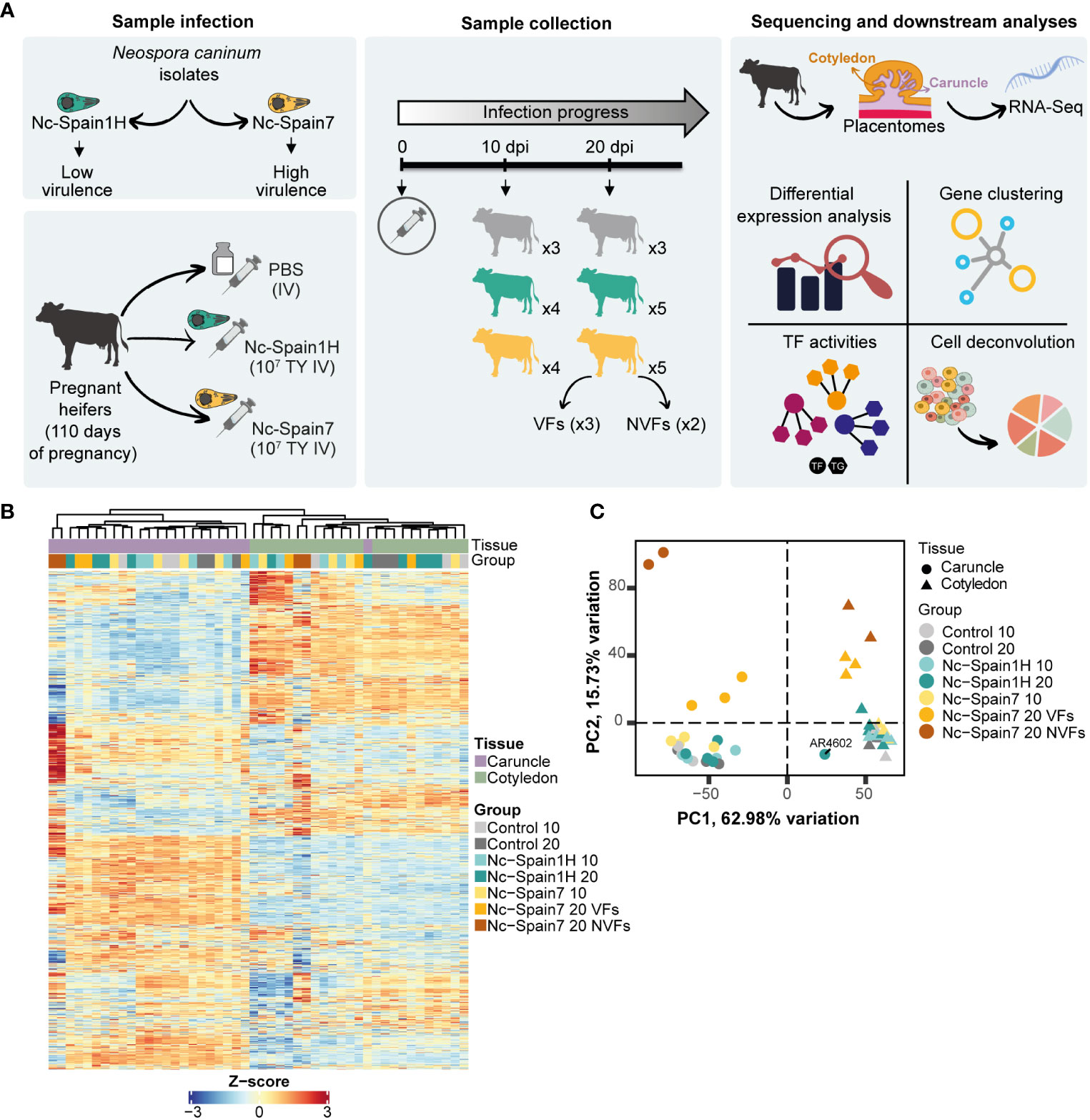 Figura 1 Diseño experimental para análisis de transcriptomas. (A) Visión general del diseño experimental. Los colores representan los diferentes grupos de novillas: gris para las novillas control inoculadas con PBS, verde para las novillas infectadas con el aislado Nc-Spain1H y amarillo para las novillas infectadas con el aislado Nc-Spain7. FV: fetos viables; NVF: fetos no viables; TF: factor de transcripción; TG: gen diana; TY: taquizoítos; IV, por vía intravenosa. (B) El mapa de calor representa el perfil transcriptómico de cada muestra utilizando puntuaciones Z de genes expresados en la carúncula (púrpura) y el cotiledón (verde). (C) Análisis de componentes principales (PCA) de los 5000 genes más variables de muestras de carúncula (círculo) y cotiledón (triángulo) en 10 y 20 dpi. Cada punto representa una muestra de RNA-Seq. Las muestras infectadas con Neospora caninum y las muestras de control se indican con diferentes colores. Los valores atípicos están etiquetados.
Figura 1 Diseño experimental para análisis de transcriptomas. (A) Visión general del diseño experimental. Los colores representan los diferentes grupos de novillas: gris para las novillas control inoculadas con PBS, verde para las novillas infectadas con el aislado Nc-Spain1H y amarillo para las novillas infectadas con el aislado Nc-Spain7. FV: fetos viables; NVF: fetos no viables; TF: factor de transcripción; TG: gen diana; TY: taquizoítos; IV, por vía intravenosa. (B) El mapa de calor representa el perfil transcriptómico de cada muestra utilizando puntuaciones Z de genes expresados en la carúncula (púrpura) y el cotiledón (verde). (C) Análisis de componentes principales (PCA) de los 5000 genes más variables de muestras de carúncula (círculo) y cotiledón (triángulo) en 10 y 20 dpi. Cada punto representa una muestra de RNA-Seq. Las muestras infectadas con Neospora caninum y las muestras de control se indican con diferentes colores. Los valores atípicos están etiquetados.
Para los análisis de RNA-seq, tres placentomas mediales aleatorios se separaron cuidadosamente a mano, y las carúnculas maternas (AC) y los cotiledones fetales (CO) se separaron y se colocaron en RNAlater (Thermo Fisher Scientific, Paisley, Reino Unido) y se almacenaron a -80 ° C hasta que se usaron para los análisis.
2.3 Extracción de ARN
Se mezclaron tres CA y OC mediales de cada animal por separado para cada tejido, y el ARN total se extrajo utilizando el kit de purificación Maxwell 16 LEV simplyRNA, desarrollado para el sistema automatizado Maxwell 16 (Promega, Madison, WI, EUA), siguiendo las recomendaciones del fabricante.®®
2.4 Preparación y secuenciación de la biblioteca Illumina
La cantidad y calidad de la muestra total de ARN se determinó mediante un kit de ensayo Qubit RNA BR (Thermo Fisher Scientific) y un ensayo RNA 6000 Nano Bioanalyzer 2100 (Agilent, Santa Clara, CA, EUA). La biblioteca RNA-Seq se preparó con el kit de plataformas KAPA Stranded mRNA-Seq Illumina (Roche Diagnostics Corporation, Indianapolis, IN, EUA) siguiendo las recomendaciones del fabricante. Brevemente, se utilizaron 500 ng de ARN total como material de entrada, la fracción poli-A se enriqueció con perlas magnéticas oligo-dT y el ARNm se fragmentó. La especificidad de la hebra se logró durante la síntesis de la segunda hebra realizada en presencia de dUTP. El ADNc bicatenario de extremo romo fue adenilado 3′, y se ligaron adaptadores compatibles con la plataforma Illumina con índices duales únicos e identificadores moleculares únicos (Integrated DNA Technologies, Inc., Coralville, Iowa, EE. El producto de ligadura se enriqueció con 15 ciclos de PCR, y la biblioteca final se validó en un bioanalizador Agilent 2100 con el ensayo DNA 7500.®®
Las bibliotecas se secuenciaron en un NovaSeq 6000 (Illumina, San Diego, CA, EUA) con una longitud de lectura de 2×101 pb siguiendo el protocolo del fabricante para la indexación dual. El análisis de imágenes, la llamada base y la puntuación de calidad de la ejecución se procesaron utilizando el software del fabricante Análisis en tiempo real (RTA v3.4.4), seguido de la generación de archivos de secuencia FASTQ.
Las lecturas sin procesar de RNA-Seq se preprocesaron utilizando la herramienta Fastp (23) para filtrar lecturas de baja calidad y secuencias de adaptadores. El control de calidad de los archivos fastq se realizó utilizando el software FastQC (v0.11.4) (24) antes y después del recorte. Luego, las lecturas de extremo pareado del filtro se mapearon contra el genoma de referencia de Bos taurus (ARS-UCD1.2) proporcionado por la versión 103 de la base de datos ENSEMBL usando STAR (v2.7.0) (25), y las abundancias a nivel de gen se estimaron utilizando la opción genecounts del software STAR.
2.5 Análisis diferencial de la expresión génica
El análisis de expresión diferencial de RNA-Seq se realizó por separado para muestras de carúncula y cotiledón utilizando el paquete DESeq2 (v1.30.1) (26). Los genes con baja expresión (aquellos genes con una expresión promedio en todas las muestras del mismo tejido menos de 1 lectura) se eliminaron antes del análisis de expresión génica, dejando 19392 y 18468 genes únicos para las muestras de carúncula y cotiledón, respectivamente. Los datos de expresión se normalizaron mediante la transformación de estabilización de varianza (VST) para el análisis exploratorio de datos mediante análisis de componentes principales (PCA) y agrupamiento jerárquico. La PCA de los 5000 genes más variables se realizó utilizando el paquete PCAtools (v2.2.0) (27) de Bioconductor. El análisis de expresión diferencial se realizó comparando muestras infectadas por aislados de Nc y todas las muestras control (Nc-Spain1H comparado con el control y Nc-Spain7 comparado con el control) a 10 y 20 dpi. Para cada análisis, los genes con una tasa de falso descubrimiento (FDR) del 5% y al menos 30 recuentos de lectura en bruto en el 70% de las muestras de un grupo incluido en el contraste se consideraron genes expresados diferencialmente (DEG). Para calcular la similitud entre los DEGs obtenidos de los diferentes contrastes, utilizamos la función overlap_coefficient del paquete Genetonic (v1.2.0) (28). Brevemente, el algoritmo de coeficiente de superposición calcula la similitud entre dos conjuntos de entidades utilizando la intersección de estos dos conjuntos y dividiéndolo por el conjunto más pequeño.
2.6 Análisis funcional y de enriquecimiento de conjuntos de genes
Utilizamos el paquete clusterProfiler (v3.18.1) (29) para realizar análisis de enriquecimiento del proceso biológico (BP) de Gene Ontology (GO) y de la Enciclopedia de Genes y Genomas de Kioto (KEGG) utilizando DEG de cada comparación y genes expresados como lista de fondo. Utilizamos el paquete biomaRt (v2.46.3) (30) para construir una anotación GO para procesos biológicos asociados con identificaciones de genes ENSEMBL. Org. Bt.eg.db (v3.12.0) (31) de Bioconductor se utilizaron para recuperar anotaciones KEGG del genoma de Bos taurus. Los términos de enriquecimiento con al menos 3 genes y los valores de P ajustados por FDR < 0,05 se consideraron términos de enriquecimiento significativos.
Para reducir las listas de términos del análisis de enriquecimiento GO, calculamos la similitud semántica entre los términos GO por pares y los agrupamos utilizando el paquete rrvgo (v1.2.0) (32) de Bioconductor. Los árboles de racimo se cortaron con un umbral entre 0,6 y 0,9 dependiendo del número de términos, y el término enriquecido con GO más significativo (el que tiene un bajo valor FDR) se seleccionó como término representativo.
El análisis de enriquecimiento de conjuntos de genes (GSEA) (33) también se realizó utilizando el paquete clusterProfiler. Realizamos GSEA utilizando una lista de genes clasificada por el parámetro stat de DESeq2 y el conjunto de genes distintivos de la Base de Datos de Firmas Moleculares (MSigDB) (34) (paquete msigdbr R v7.4.1) (35). Se seleccionaron las vías con un valor de FDR < 0,1 como significativas.
2.7 Análisis de agrupamiento
Los genes expresados se utilizaron para analizar la dinámica de cada infección aislada. La expresión normalizada de VST se escaló a la puntuación z, y la media de los genes de cada grupo se utilizó para la agrupación. Los genes se agruparon utilizando el método de partición alrededor de los medoides (PAM). Para validar los resultados del clúster, calculamos el coeficiente de silueta y lo visualizamos utilizando la función fviz_silhouette del paquete factoextra (v1.0.7) (36). También realizamos un análisis de enriquecimiento funcional para cada subconjunto de genes utilizando la anotación GO BP como se describió anteriormente.
2.8 Análisis de deconvolución
Para estimar el porcentaje de poblaciones de células inmunes en muestras de ARN de carúncula y cotiledón, utilizamos el algoritmo CIBERSORTx (37). La expresión génica se normalizó a la transcripción por millón (TPM), y ENSEMBL ID del genoma de Bos taurus se convirtió en ortólogos humanos utilizando biomaRt (v2.46.3). Para seleccionar los mejores pares de ortólogos en caso de que hubiera más de un ortólogo humano para diferentes genes de vaca, clasificamos los genes por su puntaje de conservación de orden de genes humanos, cobertura de alineación del genoma completo y promedio entre identidad porcentual objetivo y consulta y ortólogos seleccionados con una confianza de ortología de homología igual a 1. Después de eso, ENSEMBL ID se convirtió en símbolos de genes para crear el archivo de mezcla. Como matriz de firma, utilizamos la matriz de firma de leucocitos (LM22) disponible en CIBERSORTx, que tiene genes de firma de 22 tipos diferentes de células inmunes. Para ejecutar CIBERSORTx, utilizamos la corrección por lotes en modo B con el archivo de perfil de expresión génica (GEP) de LM22, incluido en su sitio web, 100 permutaciones y modo absoluto.
2.9 Análisis de actividad del factor de transcripción
Utilizamos el paquete decoupleR (v2.3.2) (38) de Bioconductor para inferir las actividades del factor de transcripción (TF) de dos maneras diferentes, utilizando la matriz de expresión génica y utilizando el cambio log2 fold (FC) obtenido del análisis de expresión diferencial. La expresión génica se normalizó utilizando la función VST del paquete Deseq2. Para inferir las actividades de TF, utilizamos regulones DoRothEA (39) del genoma humano con el nivel de confianza ABC. Los nombres de genes del genoma de Bos taurus en la matriz de expresión génica normalizada se convirtieron en ortólogos humanos como se explicó anteriormente. Se utilizó el método de la media ponderada para inferir actividades mediante la función run_wmean con 1000 permutaciones y un número mínimo de dianas por TF = 5, y se seleccionaron norm_wmean actividades.
2.10 Análisis estadístico y gráficos
Todos los análisis estadísticos se realizaron en R (v4.0.3) (40). Los paquetes ComplexHeatmap (v2.6.2) (41) y ComplexUpset (v1.3.3) (42, 43) se utilizaron para representar los mapas de calor y los gráficos de trastorno, respectivamente. La gráfica del coeficiente de superposición se generó utilizando el paquete corrplot (v0.92) (44). Los otros gráficos se generaron utilizando el paquete ggplot2 (v3.3.6) (45).
3 Resultados
3.1 El tejido placentario y el estado de infección dominan la remodelación del transcriptoma
Para estudiar las respuestas placentarias a la infección por diferentes aislados de N. caninum a nivel transcripcional, realizamos un análisis exploratorio de los datos de RNA-Seq de carúnculas y cotiledones obtenidos de novillas infectadas con el aislado de alta virulencia Nc-Spain7 (n = 9) y el aislado de baja virulencia Nc-Spain1H (n = 9) y novillas no infectadas (n = 6) (Figura 1A). Se perfilaron un total de 48 muestras con un promedio de 34,1 millones de lecturas por muestra (Figura suplementaria 1A). De todas las muestras, entre el 75 y el 96% de las lecturas se mapearon contra el genoma de referencia (Tabla suplementaria 1). Las carúnculas y los cotiledones exhiben transcriptomas claramente distinguibles (Figura 1B) y las muestras de novillas donde la viabilidad fetal estaba comprometida muestran firmas transcriptómicas reconocibles (Figura 1B). Los coeficientes de correlación de Pearson revelaron dos grupos principales de muestras correspondientes cada uno al tejido de origen (es decir, cotiledóneo o carúncula) (Figura suplementaria 1B). Esta separación prominente entre las muestras de cotiledóneo y carúncula indica la existencia de un programa transcripcional específico para cada componente de la placenta.
El análisis de los componentes principales de la mayoría de los genes variables reveló grupos de muestras según el tejido placentario, el aislado utilizado para la infección experimental y la fecha de sacrificio. Como era de esperar, el componente principal 1 (PC1) mostró que las firmas genéticas específicas de la carúncula y el cotiledón representaron la principal fuente de variación (62,98%). Además, el componente principal 2 (PC2) reveló conglomerados de muestras correspondientes a animales infectados por el aislado de alta virulencia a 20 dpi (15,73% de variación) (Figura 1C). También encontramos una separación en PC2 entre muestras de animales infectados por Nc-Spain7 de novillas que habían sufrido muerte fetal (NVFs, fetos no viables) y aquellos cuyos fetos aún estaban vivos (VFs, fetos viables) a 20 dpi (Figura 1C). Sin embargo, no encontramos una separación obvia entre las muestras de carúnculas o cotiledón de animales no infectados versus los infectados con el aislado de baja virulencia (Nc-Spain1H) en cualquier dpi o con el aislado de alta virulencia (Nc-Spain7) a 10 dpi. Durante estos análisis, detectamos un valor atípico (AR4602) entre las muestras de carúncula infectadas con el aislado Nc-Spain1H que se agrupó con otras muestras de cotiledón tanto en el PCA (Figura 1C) como en el mapa de calor de los genes expresados (Figura 1B). Atribuimos esto a una separación incorrecta de la carúncula y el cotiledón durante el desprendimiento mecánico de placentomos, y en consecuencia, esta muestra se eliminó para su posterior análisis.
Los transcriptomas resultantes de animales no infectados se distinguieron notablemente por su origen cotiledonario o caruncular, pero no por el día de la eutanasia (10 o 20 dpi) (Figura 1B). Por lo tanto, independientemente del momento de la eutanasia, todas las muestras de control recogidas del mismo tipo de tejido se consideraron como una condición única para análisis adicionales. Además, el mapa de calor reveló una menor correlación del grupo NVF en comparación con otros grupos, lo que fue más notable en las muestras de carúncula. Por lo tanto, las muestras de NVF de la carúncula y el cotiledón se descartaron para el análisis de expresión diferencial para evitar la interferencia potencial del proceso de muerte fetal en las modificaciones transcriptómicas observadas.
3.2 La remodelación del transcriptoma aumenta robustamente en las carúnculas durante el curso de la infección y de manera dependiente del aislamiento
Neospora caninum indujo cambios transcriptómicos robustos en los placentomas durante la infección, que fueron más prominentes para el aislado de alta virulencia. En las carúnculas, se encontraron diferencias sutiles en los niveles de expresión génica en las primeras etapas de la infección (10 dpi) y, más particularmente, en el caso del aislado de baja virulencia (Nc-Spain1H) donde solo se expresaron diferencialmente 4 genes (por ejemplo, SLC12A7, KIFC3, LRFN4 y TOLLIP) con niveles de expresión media más bajos en animales infectados que en animales no infectados (Figura 2A). En el caso del aislado de alta virulencia (Nc-Spain7), un total de 245 (DEGs resultaron de la comparación entre animales infectados y no infectados, donde TOLLIP también tuvo una expresión media más baja en las muestras infectadas (Figura 2A). En la etapa posterior de la infección (20 dpi), las diferencias transcriptómicas fueron más pronunciadas para ambos aislados, representando 801 y 2.607 DEG para los aislados Nc-Spain1H y Nc-Spain7, respectivamente. La Figura 2A muestra el número de DEG y las intersecciones resultantes entre los contrastes probados, y estos se resumen en la Tabla Suplementaria 2.
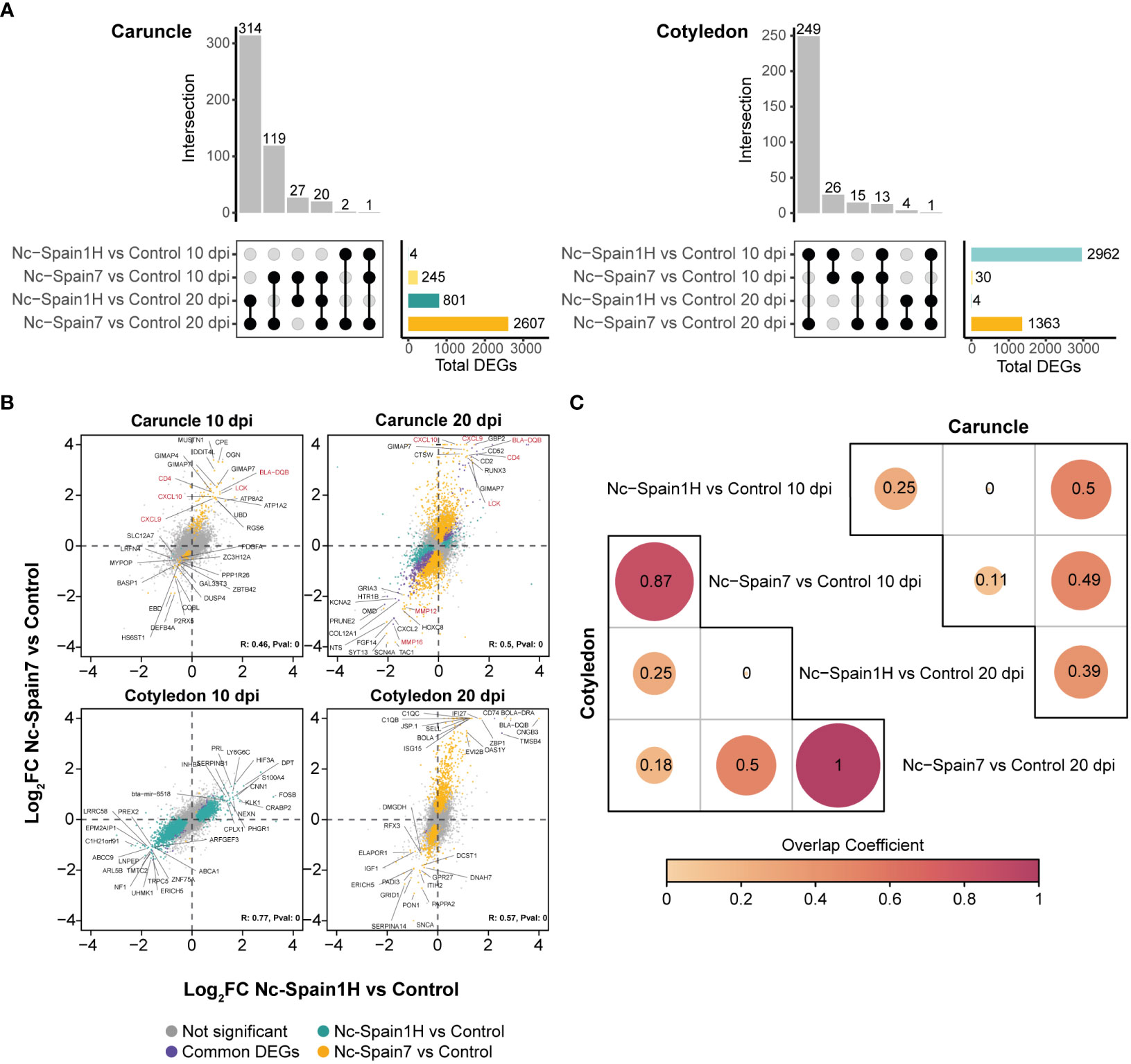 Figura 2 Análisis transcriptómico de muestras infectadas por aislados de N. caninum y muestras no infectadas. (A) El diagrama UpSet muestra la intersección de genes expresados diferenciales (DEGs) obtenidos del análisis de expresión diferencial de cada contraste de la carúncula (izquierda) y el cotiledón (derecha). Las barras verticales muestran el número de DEG comunes entre diferentes contrastes, de acuerdo con los círculos negros conectados. Las barras horizontales muestran el número total de DEG en cada comparación. Los genes con un 5% de FDR se consideraron DEG. (B) Correlación de Pearson del cambio log2 veces entre infecciones de alta y baja virulencia. Cada punto representa un gen, y se etiquetan los 15 genes principales con el cambio log2 veces absoluto superior significativo en al menos un contraste. El verde representa los DEG entre las muestras infectadas por Nc-Spain1H y las muestras control, el amarillo representa los DEG entre las muestras infectadas por Nc-Spain7 y las muestras control, el púrpura representa los DEG comunes entre los dos contrastes y el gris no representa genes significativos en ninguna comparación. (C) Corrplot muestra el coeficiente de solapamiento entre los DEG de las muestras infectadas por Nc-Spain1H en comparación con las muestras de control y las muestras infectadas por Nc-Spain7 en comparación con las muestras de control a 10 y 20 dpi por separado en la carúncula (superior) y el cotiledón (inferior).
Figura 2 Análisis transcriptómico de muestras infectadas por aislados de N. caninum y muestras no infectadas. (A) El diagrama UpSet muestra la intersección de genes expresados diferenciales (DEGs) obtenidos del análisis de expresión diferencial de cada contraste de la carúncula (izquierda) y el cotiledón (derecha). Las barras verticales muestran el número de DEG comunes entre diferentes contrastes, de acuerdo con los círculos negros conectados. Las barras horizontales muestran el número total de DEG en cada comparación. Los genes con un 5% de FDR se consideraron DEG. (B) Correlación de Pearson del cambio log2 veces entre infecciones de alta y baja virulencia. Cada punto representa un gen, y se etiquetan los 15 genes principales con el cambio log2 veces absoluto superior significativo en al menos un contraste. El verde representa los DEG entre las muestras infectadas por Nc-Spain1H y las muestras control, el amarillo representa los DEG entre las muestras infectadas por Nc-Spain7 y las muestras control, el púrpura representa los DEG comunes entre los dos contrastes y el gris no representa genes significativos en ninguna comparación. (C) Corrplot muestra el coeficiente de solapamiento entre los DEG de las muestras infectadas por Nc-Spain1H en comparación con las muestras de control y las muestras infectadas por Nc-Spain7 en comparación con las muestras de control a 10 y 20 dpi por separado en la carúncula (superior) y el cotiledón (inferior).
A pesar de las diferencias cuantitativas en el análisis de expresión diferencial entre aislados, encontramos una mayor expresión de algunos genes relacionados con la inmunidad (por ejemplo, LCK, CXCL9, CXCL10, CD4, BLA-DQB) a 10 y 20 dpi y una disminución de la expresión de genes que codifican metaloproteinasas de matriz (por ejemplo, MMP12 o MMP16) a 20 dpi en todas las muestras infectadas con los aislados Nc-Spain7 o Nc-Spain1H. Además, para evaluar la relación entre el estado de infección (no infectado versus infectado) y la virulencia del aislado, calculamos las correlaciones por pares de los cambios en la expresión génica de cada contraste. Registro2 La CF de los contrastes entre los animales infectados por Nc-Spain7- y Nc-Spain1H en comparación con los controles resultó en correlaciones positivas débiles a 10 dpi (R = 0.46, valor de P = 0) y 20 dpi (R = 0.5, valor de P = 0) (Figura 2B). Las correlaciones resultantes sugieren un predominio de las firmas génicas que se asocian principalmente con el estado de infección, pero hay algunas diferencias que podrían explicarse por otros factores experimentales, como los mecanismos de virulencia de cada aislado (Figura 2B).
3.3 Los cambios transcriptómicos inducidos por aislados de baja y alta virulencia fueron similares en los cotiledones
En los cotiledones, el número de DEG entre animales infectados y no infectados a 10 dpi fue notablemente mayor en los infectados con el aislado de baja virulencia (2.962 DEG) que en los infectados con el aislado de alta virulencia (30 DEG). Sin embargo, a pesar de estas diferencias, el cambio de pliegue de genes entre las muestras infectadas y no infectadas mostró una fuerte correlación positiva (R = 0,77, valor de p = 0; Figura 2B), que indica una gran semejanza con los cambios inducidos por el aislamiento en los cotiledones. Durante la última etapa de la infección (20 dpi), se detectaron diferencias en la expresión génica solo entre las novillas infectadas con el aislado de alta virulencia y los animales no infectados (1.363 DEG), en cambio, solo se detectaron 4 DEG entre el aislado de baja virulencia y los animales no infectados (Figura 2A). Una vez más, también se encontró una correlación positiva para los valores de cambio de pliegue de ambos grupos en esta etapa de la infección (R = 0,57, valor de p = 0) (Figura 2B).
Por lo tanto, aunque los análisis de expresión diferencial mostraron diferencias sustanciales entre los aislados en los cotiledones, sus patrones de expresión génica se correlacionaron fuertemente con el estado de la infección. Este hallazgo sugiere que el aislado utilizado para la infección tiene menos influencia en los cambios transcriptómicos en los cotiledones que en las carúnculas. De hecho, no hubo correlación entre los valores de cambio de pliegue del contraste equivalente en la carúncula y el cotiledón (Figura complementaria 2). Este hallazgo está de acuerdo con las diferentes respuestas observadas en la placenta materna y fetal después de la infección por N. caninum. Para evaluar el grado de similitud entre la lista de DEG derivados de cada contraste, se calculó el coeficiente de superposición entre contrastes que eran muy altos en los cotiledones entre aislados según el curso de la infección (Figura 2C). Los resultados detallados de todos los análisis de expresión diferencial en cotiledones se pueden encontrar en la Tabla Suplementaria 3.
3.4 Las señales proinflamatorias inducidas por aislados de baja y alta virulencia son opuestas en las carúnculas y se activan diferencialmente en los cotiledones
Para investigar los mecanismos subyacentes durante las respuestas contra los aislados de alta y baja virulencia de N. caninum, realizamos análisis de enriquecimiento funcional para las anotaciones GO y KEGG. El análisis de sobrerrepresentación de los términos BP GO y las vías KEGG mediante la consulta de las listas de DEG reveló un enriquecimiento en los procesos relacionados con el sistema inmune y la respuesta de defensa, ya sea en carúnculas o cotiledones, provocando diferentes procesos inmunes según el dpi o el aislado. Por ejemplo, el contraste entre los animales infectados por Nc-Spain7 y los no infectados en la carúncula reveló el enriquecimiento de varios términos GO relacionados con la diferenciación de células T a 10 dpi, pero a 20 dpi, se observó un enriquecimiento de los procesos inmunes involucrados en la regulación de la producción y estimulación de citoquinas (Figura complementaria 3A). Aunque no hubo enriquecimiento de términos GO entre los DEG entre las muestras infectadas por Nc-Spain1H y los controles en carúnculas (Figura complementaria 3A), el análisis utilizando anotación de las vías KEGG reveló un enriquecimiento de procesos relacionados con la diferenciación de células T a 20 dpi, como «diferenciación de células Th1 y Th2» y «diferenciación de células Th17» (Figura complementaria 3B). Curiosamente, en los cotiledones, se encontró un enriquecimiento robusto de los procesos proinflamatorios solo con el aislado de alta virulencia a 20 dpi (Figura complementaria 3). No se encontró enriquecimiento significativo en cotiledones de animales infectados con aislado de baja virulencia, lo cual es sorprendente debido al alto número de DEG a 10 dpi.
Para explorar más a fondo las implicaciones funcionales de las diferencias transcripcionales inducidas por la infección por N. caninum en placentomos, realizamos un GSEA utilizando una base de datos de conjuntos de genes distintivos curada. GSEA reveló detalles más profundos de los cambios transcriptómicos encontrados en cada tejido placentario de acuerdo con dpi o aislado. En general, los enriquecimientos funcionales fueron marcadamente diferentes entre los aislados en la carúncula en comparación con el cotiledón. Una de las diferencias más significativas fue la regulación a la baja de los genes distintivos asociados con la respuesta inflamatoria, la señalización de IL-6/JAK/STAT3, la señalización del factor de necrosis tumoral (TNF)-alfa a través de NF-κB y la coagulación en carúnculas infectadas por el aislado de baja virulencia (Nc-Spain1H) (Figura 3A). Por el contrario, estos conjuntos de genes y otros, como las respuestas IFN-γ e IFN-α, la señalización de IL2-STAT5 y el complemento, se regularon al alza temprano en muestras de carúncula infectadas por el aislado de alta virulencia (Figura 3A). Además, estos conjuntos de genes proinflamatorios fueron consistentemente regulados al alza en las carúnculas durante la etapa posterior de la infección por el aislado de alta virulencia. Sin embargo, la respuesta inmune proinflamatoria no fue inducida en ninguna de las etapas de la infección con el aislado de baja virulencia.
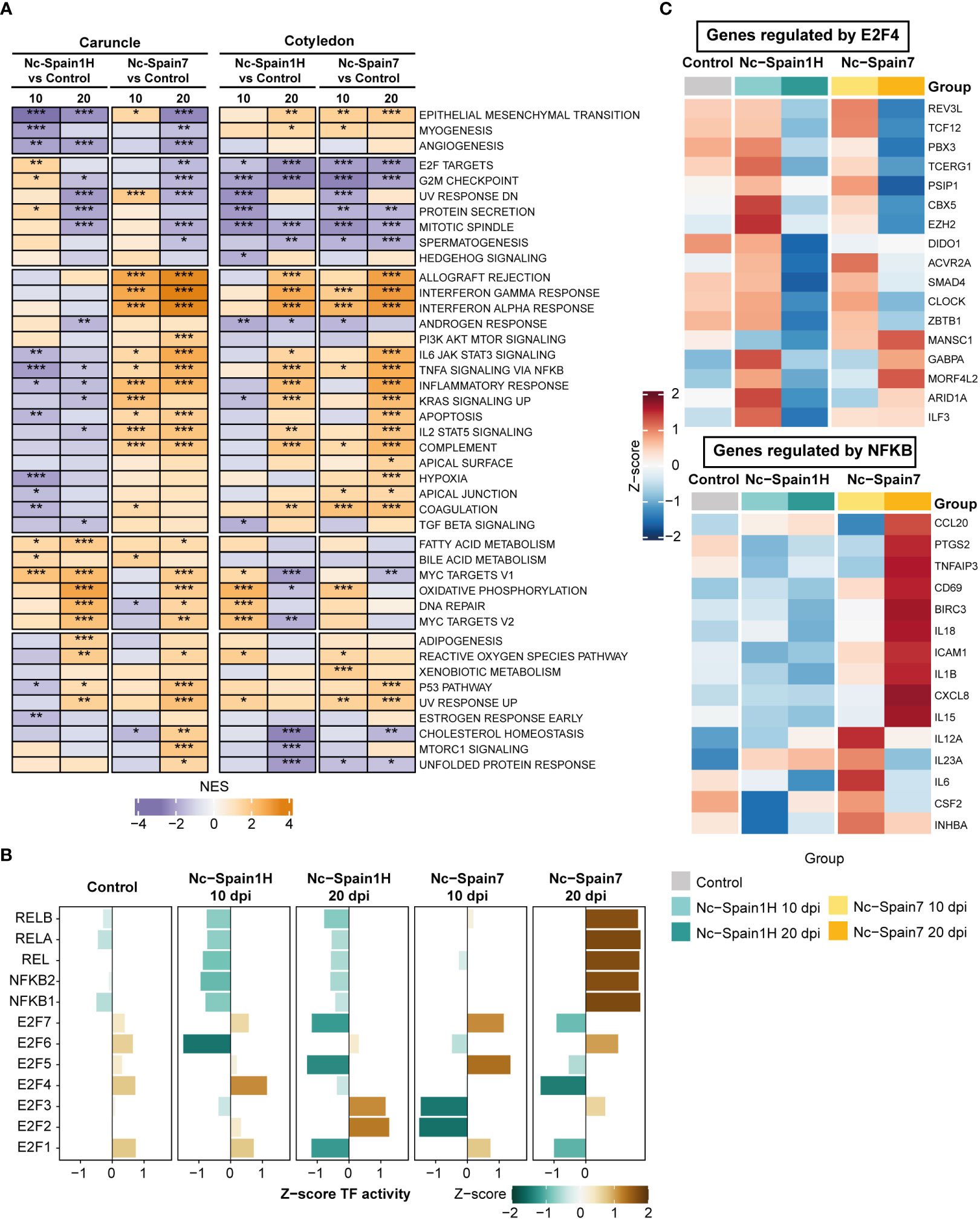 Figura 3 Análisis funcional de muestras infectadas de alta y baja virulencia en comparación con muestras control. (A) Un mapa de calor muestra las puntuaciones de enriquecimiento normalizadas (NES) de GSEA para conjuntos de genes distintivos utilizando la expresión génica diferencial de las muestras infectadas con Nc-Spain1H en comparación con las muestras de control y las muestras infectadas con Nc-Spain7 en comparación con las muestras de control por separado por fecha de sacrificio y tejido. Las vías mejoradas (NES >0) en las muestras infectadas por N. caninum están en naranja, y las vías disminuidas (NES < 0) en las muestras infectadas por N. caninum están en púrpura. Los valores de p ajustados están representados por estrellas, *** p.ajustar < 0,001, ** p.ajustar < 0,01, * p.ajustar < 0,1. (B) Los diagramas de barras representan la actividad del factor de transcripción (TF) de las familias E2F y NF-κB TF calculada utilizando la expresión génica de las muestras infectadas por aislados y control de N. caninum en carúnculas. El eje x representa la puntuación Z de la actividad media de TF por grupo. (C) Los mapas de calor muestran los niveles de expresión de dianas genéticas seleccionadas de E2F4 (superior) y NFKB (inferior) en tejido carúnculo por separado en las muestras control, infectadas con Nc-Spain1H y Nc-Spain7.
Figura 3 Análisis funcional de muestras infectadas de alta y baja virulencia en comparación con muestras control. (A) Un mapa de calor muestra las puntuaciones de enriquecimiento normalizadas (NES) de GSEA para conjuntos de genes distintivos utilizando la expresión génica diferencial de las muestras infectadas con Nc-Spain1H en comparación con las muestras de control y las muestras infectadas con Nc-Spain7 en comparación con las muestras de control por separado por fecha de sacrificio y tejido. Las vías mejoradas (NES >0) en las muestras infectadas por N. caninum están en naranja, y las vías disminuidas (NES < 0) en las muestras infectadas por N. caninum están en púrpura. Los valores de p ajustados están representados por estrellas, *** p.ajustar < 0,001, ** p.ajustar < 0,01, * p.ajustar < 0,1. (B) Los diagramas de barras representan la actividad del factor de transcripción (TF) de las familias E2F y NF-κB TF calculada utilizando la expresión génica de las muestras infectadas por aislados y control de N. caninum en carúnculas. El eje x representa la puntuación Z de la actividad media de TF por grupo. (C) Los mapas de calor muestran los niveles de expresión de dianas genéticas seleccionadas de E2F4 (superior) y NFKB (inferior) en tejido carúnculo por separado en las muestras control, infectadas con Nc-Spain1H y Nc-Spain7.
En los cotiledones, los genes asociados con la respuesta al IFN, la señalización del TNF-alfa a través de NF-κB, el complemento y la coagulación solo fueron estimulados por el aislado de alta virulencia en la etapa más temprana de la infección. Sin embargo, a 20 dpi, ambos aislamientos indujeron una fuerte respuesta proinflamatoria en este tejido (Figura 3A). Sin embargo, la comparación entre aislados mostró que esta respuesta fue más fuerte en los animales infectados con el aislado de alta virulencia (Figura complementaria 4A).
Curiosamente, se observó una ligera pero significativa regulación positiva de los genes diana de los factores de transcripción E2F y las vías relacionadas con el ciclo celular (por ejemplo, el punto de control G2M) solo en carúnculas infectadas con el aislado de baja virulencia (Figura 3A). En particular, aunque los genes diana E2F y el punto de control G2M se regularon negativamente en los cotiledones de todas las muestras infectadas, observamos fuertes diferencias entre las muestras infectadas por los aislados de alta y baja virulencia (Figura suplementaria 4A, Tablas suplementarias 4, 5).
Finalmente, algunos procesos fisiológicos esenciales (por ejemplo, homeostasis del colesterol, transición mesenquimal epitelial, apoptosis e hipoxia) revelaron actividades discordantes entre aislados en muestras de carúncula a lo largo del curso de la infección. Por el contrario, los genes relacionados con la fosforilación oxidativa tuvieron un perfil muy similar en las infecciones por ambos aislados. Todos los resultados derivados del análisis de enriquecimiento funcional se detallan en las Tablas Complementarias 6, 7.
3.5 Aumento de la actividad de E2F4 en muestras infectadas por el aislado de baja virulencia en carúnculas
Dadas las diferencias en las respuestas del huésped a la infección de los dos aislados en la carúncula, realizamos un análisis para inferir la actividad de TF en función de los niveles de expresión de sus genes diana en muestras de carúncula. Centrándonos en las familias de TF enriquecidas, como NF-κB y E2F, identificamos una mayor actividad de la familia NF-κB (por ejemplo, NFKB1, NFKB2, REL, RELA, RELB) en muestras de las novillas infectadas con el aislado de alta virulencia a 20 dpi y menor actividad para las muestras infectadas por Nc-Spain1H a 10 dpi (Figura 3B). Estas muestras también mostraron una mayor actividad de E2F4, que disminuyó en las muestras de los animales infectados por el aislado de alta virulencia (Figura 3B). Para determinar si la actividad de TF varió utilizando los resultados del análisis de expresión diferencial, también calculamos la actividad de TF utilizando valores log2FC a partir de comparaciones entre aislados a 10 dpi en carúnculas y seleccionamos los 15 TF principales con la mayor actividad en cada aislado. Estos resultados también revelaron un aumento en la actividad de TFs con un papel importante en la respuesta inmune frente a patógenos como STAT1, STAT4 y NFKB1 en las muestras infectadas con el aislado Nc-Spain7. Además, las muestras infectadas por Nc-Spain1H mostraron un aumento de la actividad de E2F4, entre otras (Figura complementaria 4B).
Teniendo en cuenta que estudios previos han demostrado una inhibición de la vía NF-κB asociada con la expresión de genes regulados por E2F3/E2F4 tras la infección por Toxoplasma gondii (46), examinamos la expresión de genes diana E2F4 seleccionados y observamos que algunos genes, como EZH2, CBX5, GABPA, ARID1A e ILF3, mostraron una mayor expresión en las muestras infectadas por Nc-Spain1H a 10 dpi (Figura 3C). En particular, se ha demostrado que EZH2 inhibe la expresión de genes diana NF-κB. En consecuencia, encontramos que la expresión de los genes diana NF-κB inhibidos por EZH2 está disminuida en las muestras infectadas por Nc-Spain1H. Sin embargo, su expresión fue elevada en las muestras infectadas por Nc-Spain7, especialmente a 20 dpi (Figura 3C). Estos hallazgos son consistentes con los resultados del análisis de actividad de TF (Figura 3B).
3.6 La PCR cuantitativa en tiempo real (qPCR) recapitula las diferencias transcriptómicas detectadas por RNA-Seq
Los resultados de RNA-Seq se validaron utilizando datos de qPCR de genes relacionados con respuestas inmunes e inflamatorias que se analizaron previamente (5). Más específicamente, el valor de FC de los datos de qPCR en cada gen se calculó como la diferencia entre el valor de Ct de cada muestra y el valor de Ct de las muestras de control. El valor resultante se utilizó como ΔΔCt, y 2–ΔΔCt se utilizó para comparar con el valor de cambio de pliegue obtenido del análisis RNA-Seq. Los valores de RNA-Seq y qPCR se correlacionaron significativamente positivamente en la mayoría de los casos en muestras de carúncula y cotiledón. Sólo el contraste entre las muestras infectadas por Nc-Spain1H y las muestras control a 10 dpi en cotiledones mostró poca correlación y un valor de p no significativo (Figuras complementarias 5A, B).
3.7 El análisis de conglomerados revela firmas genéticas específicamente relacionadas con la muerte fetal durante la infección con el aislado de alta virulencia
Para investigar la relación de la infección con la muerte fetal, analizamos los patrones de firma génica de animales no infectados y infectados por Nc-Spain7 clasificando en grupos independientes aquellas novillas sacrificadas a 10 o 20 dpi y portadoras de fetos viables o no viables.
En la carúncula, un total de 15.430 genes expresados en muestras se dividieron en 6 grupos correspondientes a patrones de expresión específicos (Figuras 4A, B). Se llevó a cabo un análisis de sobrerrepresentación de los términos GO para cada conglomerado para identificar los procesos biológicos que podrían estar involucrados en la progresión y gravedad de la infección hasta la muerte fetal (Figura 4C, Tabla suplementaria 8). El grupo 2 reveló firmas genéticas que disminuyeron su expresión durante la infección hasta que se produjo la muerte fetal, revelando varios genes asociados con el ciclo celular. Por el contrario, el grupo 5 mostró una mayor expresión de genes durante el curso de la infección y un enriquecimiento significativo de los términos relacionados con los procesos del sistema inmune. Varios genes relacionados con la organización mitocondrial y el transporte fueron regulados al alza en la infección por Nc-Spain7 a 20 dpi, como se muestra en el Cluster 4 (Figura 4C). Además, se identificaron 2 patrones divergentes entre las muestras de FV y NVF (Cluster 3 y Cluster 6). Las muestras de las novillas portadoras de fibrilación ventricular mostraron un aumento en los genes relacionados con los procesos biosintéticos de péptidos y esteroles, y estos se inhibieron en muestras de novillas portadoras de NVF (Cluster 3). Además, las muestras de FV mostraron una disminución en los genes relacionados con la organización de la matriz extracelular y el desarrollo de tejidos, y estos genes fueron regulados al alza en las muestras de NVF (Cluster 6).
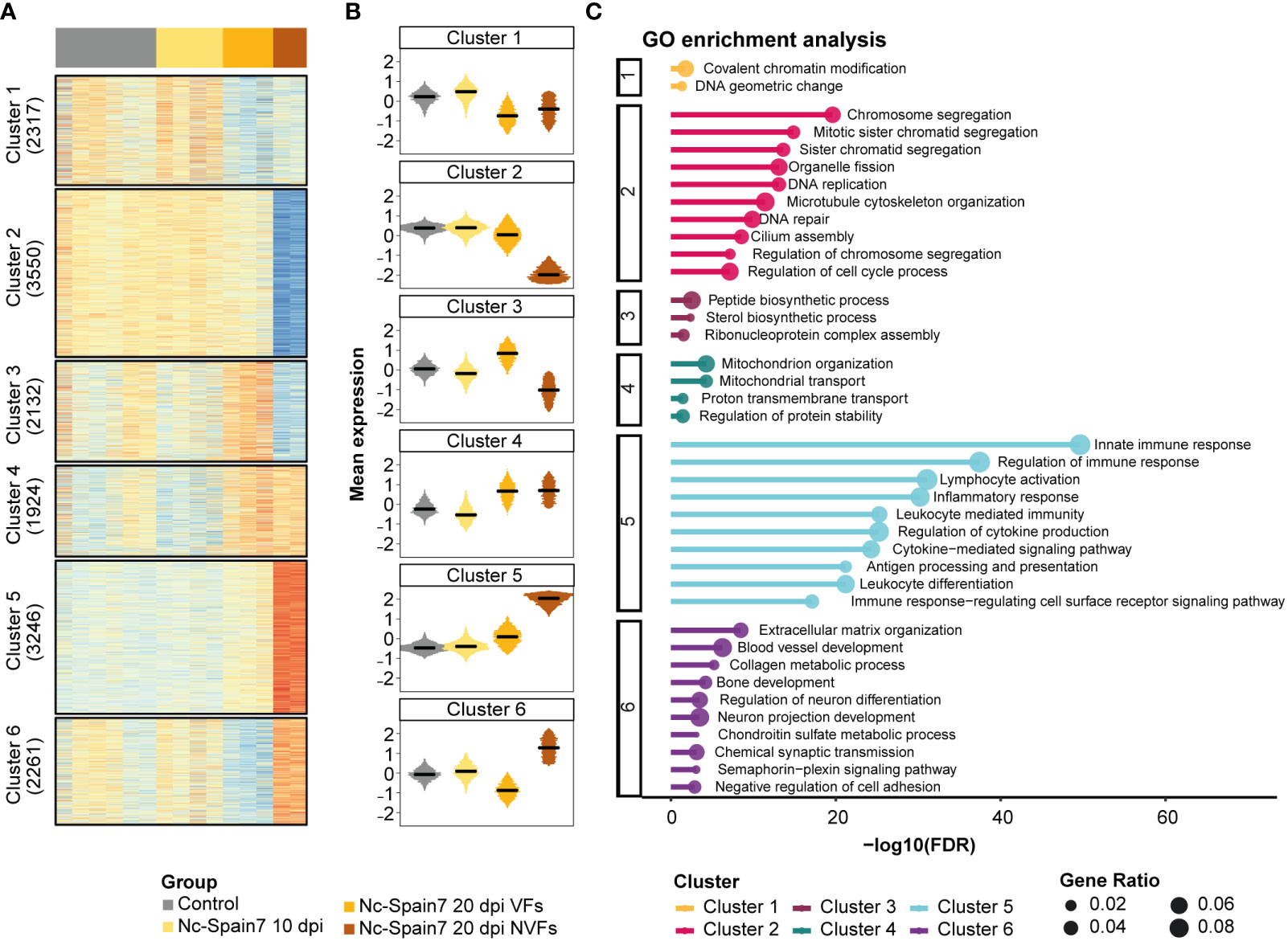 Figura 4 Dinámica de expresión génica desde la no infección hasta la muerte fetal en tejido carúnculo de animales infectados por el aislado de alta virulencia Nc-Spain7. (A) Mapa de calor de la expresión génica normalizada (puntuación Z) para las muestras control e infectadas por Nc-Spain7. Número de genes en cada grupo indicado en la anotación izquierda. (B) Los diagramas de caja representan la expresión media de genes bajo cada condición. (C) Los 10 términos más significativos (valor de p ajustado más bajo) de cada grupo. El eje x representa el valor de p ajustado -log10, y el tamaño del círculo representa la proporción de genes de cada término enriquecido.
Figura 4 Dinámica de expresión génica desde la no infección hasta la muerte fetal en tejido carúnculo de animales infectados por el aislado de alta virulencia Nc-Spain7. (A) Mapa de calor de la expresión génica normalizada (puntuación Z) para las muestras control e infectadas por Nc-Spain7. Número de genes en cada grupo indicado en la anotación izquierda. (B) Los diagramas de caja representan la expresión media de genes bajo cada condición. (C) Los 10 términos más significativos (valor de p ajustado más bajo) de cada grupo. El eje x representa el valor de p ajustado -log10, y el tamaño del círculo representa la proporción de genes de cada término enriquecido.
En los cotiledones, 13.980 firmas genéticas se agruparon en 7 subconjuntos de genes (Figuras 5A, B; Cuadro complementario 9). Los grupos 1 y 2 se regularon negativamente a medida que la infección progresaba hasta que se observó la muerte fetal. Sin embargo, curiosamente, el Cluster 2 reveló una regulación negativa específica de los genes involucrados en la organización de la matriz extracelular y el desarrollo de la vasculatura en las muestras de NVF. En contraste, la progresión de la infección por N. caninum indujo una regulación positiva de los genes asociados con la biogénesis y traducción de los ribosomas (Cluster 3) y la respuesta inmune (Clusters 5 y 6) (Figura 5C). Sin embargo, se observó una disminución de la expresión de genes asociados con la biogénesis y traducción de ribosomas (Cluster 3) y con la activación de la respuesta inmune inflamatoria (Cluster 5) una vez que la muerte fetal fue evidente, y esto también se asoció con una mayor expresión de procesos apoptóticos y necroptóticos (Cluster 6) (Figura 5C).
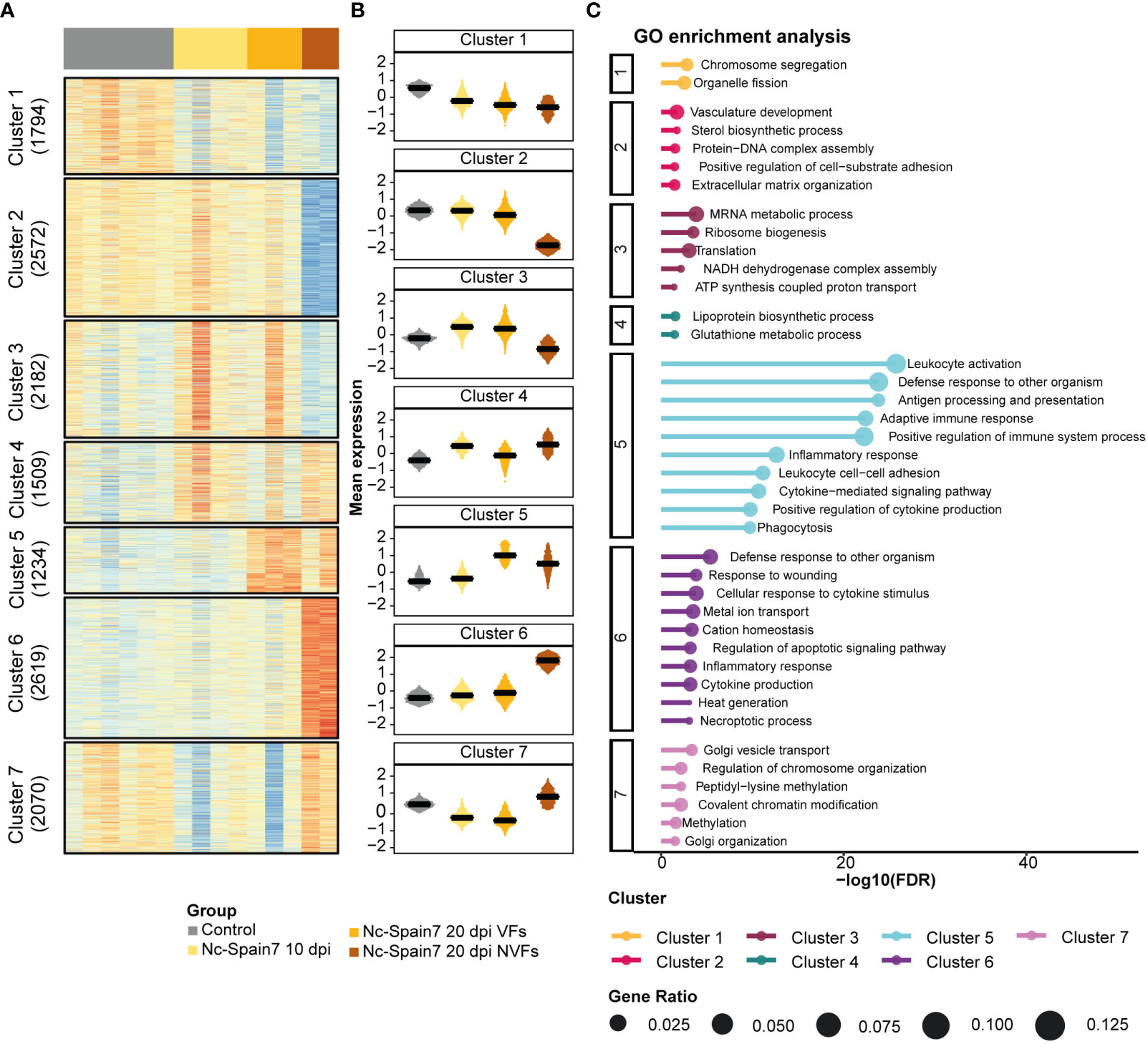 Figura 5 Dinámica de expresión génica desde la no infección hasta la muerte fetal en tejido de cotiledón de animales infectados por el aislado de alta virulencia Nc-Spain7. (A) Un mapa de calor de la expresión génica normalizada (puntuación Z) para las muestras control e infectadas por Nc-Spain7. Número de genes en cada grupo indicado en la anotación izquierda. (B) Los diagramas de caja representan la expresión media de genes bajo cada condición. (C) Los 10 términos más significativos (valor de p ajustado más bajo) de cada grupo. El eje x representa el valor de p ajustado -log10, y el tamaño del círculo representa la proporción de genes de cada término enriquecido.
Figura 5 Dinámica de expresión génica desde la no infección hasta la muerte fetal en tejido de cotiledón de animales infectados por el aislado de alta virulencia Nc-Spain7. (A) Un mapa de calor de la expresión génica normalizada (puntuación Z) para las muestras control e infectadas por Nc-Spain7. Número de genes en cada grupo indicado en la anotación izquierda. (B) Los diagramas de caja representan la expresión media de genes bajo cada condición. (C) Los 10 términos más significativos (valor de p ajustado más bajo) de cada grupo. El eje x representa el valor de p ajustado -log10, y el tamaño del círculo representa la proporción de genes de cada término enriquecido.
3.8 El análisis de deconvolución revela diferentes fracciones de contribuciones de células inmunes durante la infección por N. caninum en tejidos de carúncula y cotiledón
Para dilucidar los tipos de células inmunes involucradas en la infección por N. caninum para cada aislado, empleamos la herramienta CIBERSORTx para realizar un análisis de deconvolución de muestras de ARN a granel (37).
La deconvolución de muestras de NVF de carúncula Nc-Spain7 reveló un aumento en los tipos de células inmunes como monocitos, macrófagos (M0, M1 y M2), células NK activadas, células CD8, células B de memoria, células T foliculares auxiliares y mastocitos activados. La mayoría de estas poblaciones celulares también aumentaron en las muestras de FV Nc-Spain7, excepto en la fracción de células M2, que fue menor en las muestras de FV Nc-Spain7 que en las muestras de animales infectados con el aislado Nc-Spain1H. También encontramos que los eosinófilos y las células B naïve tenían una menor representación en las muestras de NVF Nc-Spain7 (Figura 6A).
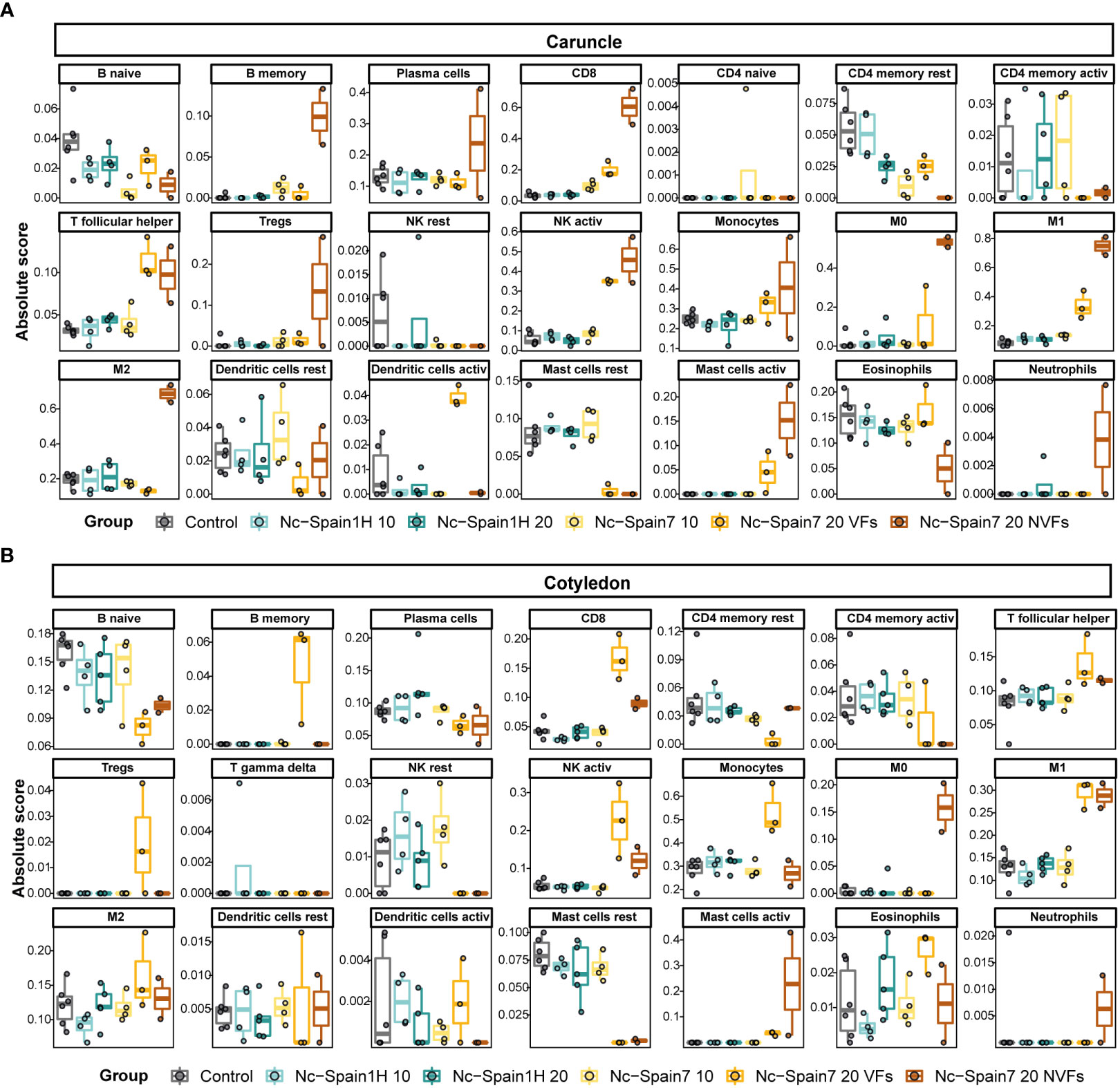 Figura 6 Resultados de la deconvolución del placentome utilizando la herramienta CIBERSORTx. Los diagramas de caja representan la puntuación absoluta de cada tipo de célula inmune en la matriz de firma LM22 obtenida en el análisis de deconvolución en cada grupo de muestra separado para los tejidos de carúncula (A) y cotiledón (B).
Figura 6 Resultados de la deconvolución del placentome utilizando la herramienta CIBERSORTx. Los diagramas de caja representan la puntuación absoluta de cada tipo de célula inmune en la matriz de firma LM22 obtenida en el análisis de deconvolución en cada grupo de muestra separado para los tejidos de carúncula (A) y cotiledón (B).
Los análisis de deconvolución realizados en muestras de cotiledones demostraron que las muestras de FV Nc-Spain7 tuvieron un aumento en la proporción de macrófagos (M1 y M2), células NK activadas, células memoria B, células T foliculares auxiliares, Tregs y células CD8, mientras que las muestras de NVF Nc-Spain7 tuvieron una mayor proporción de macrófagos M0 y mastocitos activados (Figura 6B).
4 Discusión
Aquí, por primera vez, utilizamos un modelo bovino preñado de neosporosis experimental a mediados de la gestación para estudiar las modificaciones del transcriptoma completo en la placenta durante la infección temprana con aislados virulentos y no virulentos de N. caninum. Las novillas preñadas fueron sacrificadas a 10 y 20 dpi tras la inoculación con aislados de virulencia alta (Nc-España7) o baja (Nc-Spain1H) para analizar las interacciones huésped-parásito que ocurren antes del aborto, que a menudo ocurre 30 días después de la infección. Dado que el interactoma huésped-parásito en la placenta puede desempeñar un papel clave durante la patogénesis (47-49), ideamos un análisis comparativo del transcriptoma centrado en la interfaz feto-materna de novillas infectadas por ambos aislados de N. caninum.
Dado que la interfaz feto-materna está compuesta por la interdigitación de las partes fetales (cotiledón) y maternas (carúncula), ambos tejidos se consideraron por separado en nuestro diseño experimental. Como era de esperar, nuestros análisis exploratorios revelaron dos programas transcripcionales bien definidos y diferentes correspondientes a muestras de carúncula y cotiledón. Esta diferencia entre tejidos ha sido corroborada recientemente por Diniz et al. (50), quienes encontraron 2.654 genes y vías, como el transporte de nutrientes, la diferenciación y remodelación tisular, y la tolerancia inmune, que estaban regulados diferencialmente entre carúnculas y cotiledones de novillas de carne nulíparas sanas. Estas diferencias transcripcionales específicas del tejido persisten incluso en presencia de diferentes aislamientos, lo que indica que el principal impulsor de la variación se encuentra dentro de los propios tejidos. Sin embargo, es importante tener en cuenta que nuestros resultados también revelaron procesos específicos de aislado alterados en ambos tejidos, lo que sugiere una interacción entre los programas específicos de tejido y las respuestas inducidas por aislamiento.
El análisis de expresión diferencial entre las muestras de las novillas infectadas y no infectadas reveló correlaciones positivas de sus firmas genéticas en la carúncula y el cotiledón de acuerdo con el dpi, lo que refleja una interacción similar de ambos aislados con el huésped. Esta correlación fue especialmente notable en la parte fetal de la placenta y se había observado previamente in vitro utilizando trofoblasto bovino (7). Estos resultados podrían reflejar la mayor necesidad de N. caninum de regular estrictamente el sistema inmune materno para poder atravesar la placenta y llegar al feto, donde la falta de presión inmune madura permitiría que el parásito se replique fácilmente. Las mayores diferencias transcriptómicas observadas entre los aislados en las carúnculas podrían explicar los diferentes resultados de infección desencadenados por cada aislado. Una firma común de la infección por N. caninum fue la mejora de la fosforilación oxidativa y los procesos relacionados con la mitocondria a 10 dpi en muestras de cotiledón y a 20 dpi en muestras de carúncula. De hecho, la interacción de T. gondii y N. caninum con las mitocondrias ha sido ampliamente demostrada (51, 52) y corroborada en este estudio en el caso de N. caninum. Estos resultados indican que la activación de la cadena respiratoria mitocondrial es parte de las respuestas tempranas del huésped contra la infección, ya que es una fuente importante de especies reactivas de oxígeno (ROS). Esta hipótesis está sólidamente respaldada por las vías mejoradas de especies reactivas de oxígeno que observamos tanto en carúnculas como en cotiledones al mismo tiempo después de la infección. En una comparación de ambos aislados, el proceso de fosforilación oxidativa pareció ser más altamente mejorado por el aislado de baja virulencia a 10 dpi. Este fenómeno puede permitir que el huésped controle la infección por Nc-Spain1H antes, como se sugirió anteriormente (5). Esta observación concuerda con el mayor deterioro de la producción de ROS encontrado en los macrófagos bovinos infectados por el aislado de alta virulencia en comparación con los infectados con el aislado de baja virulencia (53). Estos resultados podrían estar relacionados con la mayor abundancia de la supuesta proteína glucosa-6-fosfato deshidrogenasa (G6PD, NCLIV_000940) de N. caninum en aislados de alta virulencia (9, 54). Esta enzima contribuye a mantener el equilibrio redox (55), lo que podría ayudar a los parásitos a sobrevivir y proliferar en las células huésped. Además, un estudio reciente demostró un nuevo mecanismo de evasión inmune en N. caninum basado en la promoción de la mitofagia del huésped (56). Este mecanismo podría perjudicar la producción de ROS y atenuar la producción de citoquinas proinflamatorias. Este hallazgo abre una nueva línea de investigación para determinar si los aislados que muestran diferencias en la producción de ROS y la activación de las respuestas proinflamatorias también son capaces de modular el proceso de mitofagia de una manera dependiente del aislado.
En cuanto a las diferencias encontradas entre los aislados, la respuesta inmune merece especial atención. Nuestros resultados destacaron que la infección con el aislado de alta virulencia Nc-Spain7 indujo una respuesta inmune más fuerte tanto en carúnculas como en cotiledones, aunque estas diferencias fueron especialmente notables en el lado materno de la placenta. Estos resultados concuerdan con estudios previos que han demostrado una regulación positiva de la respuesta proinflamatoria en placenta, principalmente en la parte materna, después de la infección por N. caninum con aislados de alta virulencia en diferentes etapas de la infección (14, 21, 22, 57). La sobreexpresión de genes relacionados con la señalización de TNF-alfa a través de la señalización NF-κB, IL-6/JAK/STAT3, la señalización IL-2/STAT5, las respuestas IFN-γ e IFN-α en las carúnculas demuestra la activación de una fuerte respuesta proinflamatoria después de la infección con el aislado de alta virulencia, que no se observó en novillas infectadas con el aislado de baja virulencia. De hecho, los GSEA mostraron una regulación a la baja de conjuntos de genes como la señalización de TNF-alfa a través de NF-κB, la señalización de IL-6 / JAK / STAT3 y la respuesta inflamatoria después de la infección con el aislado de baja virulencia en una etapa temprana de la infección. Estos resultados contrastan con la respuesta proinflamatoria más fuerte descrita para el aislado de baja virulencia a 10 dpi cuando algunas citocinas fueron analizadas por qPCR (5). Esta discrepancia entre los estudios probablemente refleja las enormes diferencias entre los enfoques de qPCR y RNA-seq en términos de potencia y resolución. Los resultados obtenidos a través de la secuenciación completa del transcriptoma realizada en este estudio se basan en la expresión de más de trece mil genes, por el contrario, en los estudios basados en qPCR solo se analizan unos pocos genes. Por lo tanto, los resultados del presente estudio muestran una imagen más realista de las interacciones a nivel de placentoma durante la infección temprana. En este estudio, el transcriptoma de las carúnculas obtenido de animales infectados con el aislado de baja virulencia a 10 dpi fue muy similar al observado de los mismos tejidos en animales no infectados, con solo 4 genes que se expresan diferencialmente. Curiosamente, TOLLIP fue regulado a la baja en ambos aislados. Este gen se ha asociado con un efecto antiinflamatorio al amortiguar la inflamación mediada por TLR y, por lo tanto, suprimir la expresión de mediadores inflamatorios como TNF-α, IL-6 e IL-1β, entre otros (58, 59). Este fenómeno puede ser una explicación para la sobreexpresión significativa de IL-6 observada en las muestras infectadas por Nc-Spain7. Se ha sugerido que Leishmania donovani explota TOLLIP para su propio establecimiento (60), y todavía se necesitan estudios específicos en N. caninum para determinar su papel en la biología del parásito. El efecto de la regulación a la baja de este gen estimulando la respuesta inmune no se observó en los animales infectados con Nc-Spain1H. La falta de respuesta proinflamatoria por el aislado Nc-Spain1H en la carúncula a 10 dpi podría estar relacionada con la sobreexpresión de genes regulados por E2F3/E2F4 observada en novillas infectadas. El gen EZH2 regulado por E2F podría ser responsable de la regulación negativa de la actividad de NF-kB durante las infecciones por T. gondii, lo que contribuye al equilibrio inmune del huésped y la persistencia del parásito en ratones (46). Aquí, observamos que EZH2 solo estaba regulado al alza en novillas infectadas por el aislado de baja virulenciae, que podría considerarse una regulación específica de aislado que proporciona una ventaja evolutiva al parásito. Esta regulación evitaría una respuesta inmune exacerbada, evitando el daño placentario y el aborto y promoviendo la persistencia del parásito a través de la transmisión inofensiva a generaciones sucesivas. En T. gondii, la activación del gen EZH2 parece ser inducida por el efector TEEGR (46). Sin embargo, este efector está mal conservado en el genoma de N. caninum (46), lo que indica la necesidad de una futura exploración de los genes responsables de este fenotipo.
En particular, a partir de 10 dpi, los genes relacionados con la apoptosis se sobreexpresan en las carúnculas de los animales infectados con el aislado de alta virulencia, pero se regulan a la baja en aquellos infectados por el aislado de baja virulencia. La modulación de los procesos apoptóticos ha sido ampliamente descrita en N. caninum y T. gondii (6, 61-63), pero su importancia biológica sigue sin estar clara. El examen histológico de placentas de novillas infectadas con el aislado de alta virulencia mostró áreas de necrosis focal que no se encontraron en muestras de animales infectados con el aislado de baja virulencia (17, 20). Aunque ambos procesos son diferentes, podrían estar relacionados, ya que la necroptosis se define como una forma programada de muerte necrótica y puede ser encendida por las mismas señales de muerte que inducen la apoptosis, pero también por receptores de reconocimiento de patógenos, incluidos los receptores tipo Toll y los receptores similares a NOD (64, 65). En cualquier caso, es probable que la muerte celular excesiva tuviera un impacto negativo en el microambiente de la interfaz materno-fetal y podría ayudar a desencadenar la muerte fetal (66).
Las diferencias entre los aislados de alta y baja virulencia son mucho más marcadas a medida que avanza la infección. A 20 dpi, los animales infectados por Nc-Spain7 mostraron una inducción más fuerte de la respuesta inmune y la apoptosis, mientras que la respuesta inmune fue mucho más moderada y controlada en los animales infectados con aislados de baja virulencia. Este hallazgo está en línea con estudios previos que demostraron que el aislado Nc-Spain1H no es capaz de crecer exponencialmente in vitro en una línea celular de carúncula bovina (67) y que solo puede detectarse en la carúncula de un animal después de 20 dpi (20). Estos hallazgos apoyan la idea de que el aislado de baja virulencia tiene una menor tasa de replicación que impide el desarrollo de un ambiente proinflamatorio exacerbado en la placenta, evitando así alteraciones estructurales y funcionales y, en consecuencia, el aborto. También corroboramos la influencia de la respuesta inmune en la ocurrencia de muerte fetal a través de análisis de agrupamiento. Estos análisis demostraron que la progresión de la infección hasta la muerte fetal en las carúnculas dio lugar a una sobreexpresión de genes enriquecidos en procesos biológicos relacionados con la respuesta inmune proinflamatoria. Esto corrobora la hipótesis previamente establecida que propone que el aborto es la consecuencia de un mecanismo inmunomediado (3, 15-17, 21, 68). La sobreexpresión de genes relacionados con respuestas proinflamatorias en novillas infectadas por el aislado de alta virulencia podría desencadenarse por la expresión de TF, como STAT1, STAT4 y NFKB1, que son necesarios para montar una respuesta inmune contra patógenos. De hecho, la subversión ejercida por los efectores de T. gondii sobre las vías gobernadas por estos TF ha sido ampliamente definida (69). A la vista de nuestros resultados, sería interesante determinar qué factores de N. caninum están implicados en este tipo de modulación del huésped y cómo influye la virulencia del aislado del parásito en este factor.
En la parte fetal de la placenta, los resultados fueron marcadamente diferentes de los observados en las carúnculas. Ambos aislados indujeron una respuesta inflamatoria similar, siendo más tempranos y más fuertes en los animales infectados con el aislado de alta virulencia. Como se ha comentado anteriormente para muestras de carúnculas, estos resultados contrastan con la propuesta de una mayor estimulación inmune por el aislado Nc-Spain1H (5), estas discrepancias serán investigadas más a fondo. En general, encontramos una correlación positiva entre los datos de RNA-seq presentados aquí y nuestros resultados previos de qPCR, excepto para los animales infectados por Nc-Spain1H en comparación con los animales no infectados a 10 dpi en cotiledones, en los que se encontraron resultados no significativos, y este contraste merece especial atención. Aunque encontramos un alto número de DEG (2962) entre los animales infectados por Nc-Spain1H y no infectados en cotiledones, este resultado no se asoció con un enriquecimiento de ninguna función biológica específica. Sin embargo, este hallazgo podría reflejar una interacción temprana de Nc-Spain1H con el huésped que posteriormente permite el control local contra la replicación del parásito. Sin embargo, a pesar de las diferencias observadas en los análisis de expresión diferencial, encontramos una alta correlación entre el transcriptoma de animales infectados con ambos aislados en cotiledones, lo que se traduce en una interacción similar de los aislados Nc-Spain7 y Nc-Spain1H con la parte fetal de la placenta.
Los análisis de deconvolución de genes expresados demostraron que las carúnculas infectadas con el aislado de alta virulencia (FV y NVF) presentaron una mayor proporción de diferentes poblaciones inmunes, incluidos monocitos, macrófagos M1, células NK activadas, células T CD8 +, células T foliculares auxiliares y mastocitos activados. Estudios previos han demostrado la presencia de un infiltrado inflamatorio basado en células como CD3+, CD4+, CD8+, γδTCR+ y NKp46+ en carúnculas de animales infectados en diferentes etapas de infección (15, 21, 68, 70). Además, algunas de estas células se han asociado a la muerte fetal en la gestación temprana (15, 21, 68). Estos trabajos también han mostrado marcadas diferencias en la infiltración de poblaciones de células inmunes dependiendo de la etapa de gestación en la que se produce la infección. Por lo tanto, centrándose en la mitad de la gestación, de acuerdo con trabajos anteriores, las células T CD4 no serían responsables de los perfiles transcriptómicos descritos aquí (15). Sin embargo, y a diferencia de nuestros resultados, Cantón et al. (2014) (15) no observaron diferencias en las puntuaciones CD8+ o macrófagos entre madres desafiadas y animales control a mitad de gestación. Sin embargo, primero mostramos por inmunohistoquímica que los animales infectados con el aislado Nc-Spain7 presentaban una mayor proporción de linfocitos T y células fagocíticas en los tejidos placentarios, que fue más acentuada a 20 dpi (5). Posteriormente, mediante citometría de flujo, detectamos un aumento de linfocitos T CD8+ en células mononucleares de sangre periférica de las novillas infectadas con el aislado Nc-Spain7 a 9, 13 y 20 dpi (71). Las células T CD8+ están involucradas en el reconocimiento y la destrucción de células infectadas con patógenos intracelulares. Por lo tanto, el aumento de CD8+ sería un intento de reducir la multiplicación del parásito como consecuencia de la mayor invasión y replicación del aislado Nc-Spain7. Sin embargo, los aumentos de CD8+ podrían aumentar el daño tisular in vivo debido a los efectos citotóxicos (72). Además, al igual que en este estudio, un estudio in vitro con macrófagos derivados de monocitos bovinos mostró que la infección por N. caninum induce la polarización de los macrófagos hacia el fenotipo M1 (6). Por lo tanto, planteamos la hipótesis de que estas poblaciones celulares podrían ser responsables de la respuesta inmune exacerbada y el daño tisular que ocurre en la placenta durante la infección con el aislado de alta virulencia, como también se ha sugerido para las infecciones en la gestación temprana (15, 21, 68). Por otro lado, se sabe que los mediadores de los mastocitos también aumentaron en las novillas infectadas por Nc-Spain7, desempeñando un papel clave en la inflamación y en la patogénesis de otras enfermedades parasitarias protozoarias (73, 74), pero hasta donde sabemos, no hay estudios sobre su importancia en la neosporosis bovina, y este tema debe investigarse ya que hay algunos estudios en humanos que relacionan estas células con trastornos del embarazo (56, 75). Además, como se describe para T. gondii y N. caninum, algunas de estas poblaciones inmunes pueden ser secuestradas por el parásito, que puede usarlas como un caballo de Troya para propagarse sistémicamente, cruzar barreras biológicas y alcanzar órganos inmunes privilegiados (53, 76). De hecho, experimentos previos realizados con células dendríticas humanas infectadas con dLos aislados de N. caninum han mostrado variaciones significativas en su capacidad para inducir la migración a través de las membranas (77). En este sentido, nuestro trabajo previo mostró que el aislado de alta virulencia llega a los cotiledones antes (a 10 dpi) que el aislado de baja virulencia, lo que indica que Nc-Spain7 tiene la capacidad de propagarse de manera más eficiente a la parte fetal (5, 20). En relación con esto, cabe destacar la fuerte inducción transcriptómica de genes relacionados con la transición mesenquimal epitelial en carúnculas de las novillas infectadas con el aislado de alta virulencia a 10 dpi (Figura 3A). Esta transición puede ser ventajosa para el aislado Nc-Spain7, potenciando sus capacidades celulares invasivas y favoreciendo el acceso de transmisión a la parte fetal de la placenta, ya que la activación de la transición mesenquimal epitelial provoca defectos en las células epiteliales, pérdida de su polaridad apical-basal y de las uniones célula-célula y aumento de la motilidad celular y la invasión celular (78). Sorprendentemente, los macrófagos M2 tuvieron una mayor representación en las carúnculas de los animales infectados con el aislado de baja virulencia que los de los animales infectados con aislado de alta virulencia. En el ganado vacuno, en condiciones fisiológicas, los macrófagos experimentan diferenciación M2 durante el período de gestación (79). Este fenotipo de macrófagos es responsable del microambiente normal en la interfaz materno-fetal (80) y está involucrado en respuestas antiinflamatorias (81). De hecho, cualquier sesgo en el equilibrio de macrófagos M1/M2 puede conducir a trastornos del embarazo (82, 83). Este aumento de macrófagos M2 en las novillas infectadas con el aislado Nc-Spain1H podría ser clave para prevenir el aborto, y dado su papel en las respuestas antiinflamatorias, su presencia podría explicar la falta de estimulación inmune que observamos en las carúnculas infectadas con el aislado de baja virulencia. En un contexto con presencia predominante de macrófagos M2 en la placenta, se promovería un ambiente antiinflamatorio, contrarrestando así el fenotipo proinflamatorio que desencadenaría una infección parasitaria. Este fenómeno mantendría el delicado equilibrio inmunológico en la placenta que asegura el mantenimiento del embarazo (48). En línea con este hallazgo, estudios previos realizados con T. gondii han demostrado que la virulencia de la cepa determina la polarización de los macrófagos (84), y teniendo en cuenta nuestros hallazgos, sería razonable extrapolar esta situación a N. caninum. De hecho, el descubrimiento de factores parasitarios que gobiernan dicha polarización de macrófagos en diferentes aislados podría sentar las bases para el desarrollo de medidas efectivas para prevenir el aborto inducido por N. caninum. Esta polarización no se observó en los cotiledones infectados por Nc-Spain1H, aunque faltan estudios centrados en macrófagos fetales. Sin embargo, según un estudio reciente que utiliza placentas termino, los macrófagos fetales y maternos son diferentes y muestran fenotipos diferentes (85). Este estudio plantea preguntas interesantes sobre el fenotipo mostrado por los macrófagos fetales durante la gestación.
5 Conclusiones
Este estudio muestra que los aislados de diferente virulencia difieren en su interacción con la placenta huésped en las primeras etapas de la infección, lo que podría mediar el resultado de la infección. El aislado de alta virulencia desencadena una respuesta inmune proinflamatoria tan pronto como 10 dpi que aumenta más adelante en la infección. Sin embargo, el aislado de baja virulencia no activa tal respuesta inflamatoria, e incluso hubo indicios de inhibición, lo que podría facilitar su transmisión sin causar daños intensos a la carúncula. La respuesta inmune exacerbada desencadenada por el aislado de alta virulencia podría ser un contribuyente clave a la muerte fetal como se sugirió anteriormente. Los cambios transcripcionales inducidos por ambos aislados en la parte fetal fueron más similares, y ambos desencadenaron una respuesta proinflamatoria, aunque fue más fuerte y más temprana en novillas infectadas con el aislado de alta virulencia. Estos resultados indican que la modulación estrecha de la respuesta del huésped en el lado materno, la carúncula, puede ser decisiva para determinar el resultado de la infección; Sin embargo, una vez que el parásito llega al feto, esta regulación puede no ser tan importante.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres del repositorio / repositorios y los números de acceso se pueden encontrar a continuación: PRJEB58430 (European Nucleotide Archive-ENA).
Declaración ética
El estudio en animales fue revisado y aprobado por el Comité Ético de Investigación Animal del Principado de Asturias, España (número de referencia PROAE 25/2016).
Contribuciones del autor
AR-P, LO-M, MC y PH concibieron el estudio y participaron en su diseño experimental. IP-F, LR-S y PH prepararon todas las muestras para la secuenciación del transcriptoma. AR-P y MC desarrollan todos los análisis bioinformáticos y estadísticos. PH y MC escribieron el manuscrito con interpretación de los resultados y aportes de discusión de AR-P, EC-F, IP-F y LO-M. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Financiación
Este trabajo ha contado con el apoyo del Ministerio de Ciencia, Innovación y Universidades (PID2019-104713RB-C21) y la Comunidad de Madrid, España (PLATESA2-CM P2018/BAA-4370). AR-P e IP-F fueron otorgados por el Programa de Investigación «Atracción de Talento de la Comunidad de Madrid» (2017-T2/BMD-5532 y 2018T2/BIO10170, respectivamente). AR-P y MC han sido apoyados por el Programa Regional de Investigación e Innovación Tecnológica para Jóvenes Doctores UCM-CAM (PR65/19-22460).
Reconocimientos
Queremos agradecer los recursos informáticos y de software proporcionados por el Centro SCBI (Supercomputación y Bioinformática) de la Universidad de Málaga.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fimmu.2023.1198609/full#supplementary-material
Referencias
1. Pastor-Fernández I, Collantes-Fernández E, Jiménez-Pelayo L, Ortega-Mora LM, Horcajo P. Modeling the rumiant placenta-pathogen interactions in apicomplexan parasites: current and future perspectives. Front Vet Sci (2021) 7:634458. DOI: 10.3389/FVETS.2020.634458
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Reichel MP, Alejandra Ayanegui-Alcérreca M, Gondim LFP, Ellis JT. ¿Cuál es el impacto económico global de Neospora caninum en el ganado: la pregunta del billón de dólares? Int J Parasitol (2013) 43:133–42. doi: 10.1016/j.ijpara.2012.10.022
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Almería S, Serrano-Pérez B, López-Gatius F. Respuesta inmune en neosporosis bovina: protección o contribución a la patogénesis del aborto. Microb Pathog (2017) 109: 177–82. doi: 10.1016/j.micpath.2017.05.042
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Horcajo P, Regidor-Cerrillo J, Aguado-Martínez A, Hemphill A, Ortega-Mora LM. Vacunas para la neosporosis bovina: estado actual y aspectos clave para el desarrollo. Parasite Immunol (2016) 38:709–23. doi: 10.1111/pim.12342
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Jiménez-Pelayo L, García-Sánchez M, Collantes-Fernández E, Regidor-Cerrillo J, Horcajo P, Gutiérrez-Expósito D, et al. La diafonía entre Neospora caninum y el huésped bovino en la interfaz materno-fetal determina el resultado de la infección. Vet Res (2020) 51:83. DOI: 10.1186/S13567-020-00803-Y
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. García-Sánchez M, Jiménez-Pelayo L, Horcajo P, Collantes-Fernández E, Ortega-Mora LM, Regidor-Cerrillo J. La infección por Neospora caninum induce un perfil de expresión génica proinflamatoria dependiente de virulencia aislada en macrófagos derivados de monocitos bovinos. Parásitos vectores (2020) 13:374. DOI: 10.1186/S13071-020-04239-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Horcajo P, Jiménez-Pelayo L, García-Sánchez M, Regidor-Cerrillo J, Collantes-Fernández E, Rozas D, et al. Modulación del transcriptoma de células trofoblásticas bovinas in vitro por Neospora caninum. Int J Parasitol (2017) 47:791–9. doi: 10.1016/j.ijpara.2017.08.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Jiménez-Pelayo L, García-Sánchez M, Regidor-Cerrillo J, Horcajo P, Collantes-Fernández E, Gómez-Bautista M, et al. Perfil de respuesta inmune de líneas celulares carunculares y trofoblastas infectadas por aislados de Neospora caninum de alta (Nc-Spain7) y baja virulencia (Nc-Spain1H). Vectores parásitos (2019) 12:218. DOI: 10.1186/S13071-019-3466-Z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Regidor-Cerrillo J, Xia D, Jiménez-Pelayo L, García-Sánchez M, Collantes-Fernández E, Randle N, et al. Caracterización proteómica de las interacciones huésped-patógeno durante la infección de la línea celular del trofoblasto bovino por Neospora caninum. Patógenos (2020) 9:E749. doi: 10.3390/pathogens9090749
10. Abe C, Tanaka S, Ihara F, Nishikawa Y. El agotamiento de macrófagos antes de la infección por Neospora caninum produce neosporosis severa en ratones. Clin Vaccine Immunol (2014) 21: 1185-8. doi: 10.1128/CVI.00082-14
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Macaldowie C, Maley SW, Wright S, Bartley P, Esteban-Redondo I, Buxton D, et al. Patología placentaria asociada a muerte fetal en bovinos inoculados con Neospora caninum por dos vías diferentes en el precogón del embarazo. J Comp Pathol (2004) 131:142–56. doi: 10.1016/j.jcpa.2004.02.005
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Gibney EH, Kipar A, Rosbottom A, Guy CS, Smith RF, Hetzel U, et al. La extensión de la necrosis asociada al parásito en la placenta y los tejidos fetales del ganado después de la infección por Neospora caninum en la gestación temprana y tardía se correlaciona con la muerte fetal. Int J Parasitol (2008) 38:579–88. doi: 10.1016/j.ijpara.2007.09.015
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Caspe SG, Moore DP, Leunda MR, Cano DB, Lischinsky L, Regidor-Cerrillo J, et al. El aislado de Neospora caninum-Spain 7 induce daño placentario, muerte fetal y aborto en bovinos cuando se inocula en gestación temprana. Vet Parasitol (2012) 189:171–81. doi: 10.1016/j.vetpar.2012.04.034
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Regidor-Cerrillo J, Arranz-Solís D, Benavides J, Gómez-Bautista M, Castro-Hermida JA, Mezo M, et al. Neospora caninum infection during early pregnancy in cattle: how the isolate influence infection dynamics, clinical outcome and peripheral and local immune responses. Vet Res (2014) 45:10. doi: 10.1186/1297-9716-45-10
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Cantón GJ, Katzer F, Maley SW, Bartley PM, Benavides-Silván J, Palarea-Albaladejo J, et al. Expresión de citoquinas en la placenta de bovinos preñados después de la inoculación con Neospora caninum. Vet Immunol Immunopathol (2014) 161: 77–89. doi: 10.1016/j.vetimm.2014.07.004
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Almería S, Araujo R, Tuo W, López-Gatius F, Dubey JP, Gasbarre LC. Muerte fetal en vacas infectadas experimentalmente con Neospora caninum a los 110 días de gestación. Vet Parasitol (2010) 169:304–11. doi: 10.1016/j.vetpar.2009.12.044
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Almería S, Serrano-Perez B, Darwich L, Domingo M, Mur-Novales R, Regidor-Cerrillo J, et al. Muerte fetal en novillas ingenuas inoculadas con aislado de Neospora caninum nc-Spain7 a los 110 días de embarazo. Exp Parasitol (2016) 168:62–9. doi: 10.1016/j.exppara.2016.06.009
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. López-Gatius F, Pabón M, Almería S. La infección por Neospora caninum no afecta el embarazo temprano en ganado lechero. Theriogenology (2004) 62:606–13. doi: 10.1016/j.theriogenology.2003.11.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Lindsay DS, Dubey JP. Neosporosis, toxoplasmosis y sarcocistosis en rumiantes: una actualización. Vet Clin N Am Food Anim Pract (2020) 36: 205–22. doi: 10.1016/j.cvfa.2019.11.004
20. Jiménez-Pelayo L, García-Sánchez M, Vázquez P, Regidor-Cerrillo J, Horcajo P, Collantes-Fernández E, et al. Dinámica temprana de la infección por Neospora caninum en bovinos después de la inoculación a mediados de la gestación con aislados de virulencia alta (Nc-Spain7)- o baja (Nc-Spain1H). Vet Res (2019) 50:72. DOI: 10.1186/S13567-019-0691-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Rosbottom A, Gibney EH, Guy CS, Kipar A, Smith RF, Kaiser P, et al. La regulación positiva de las citoquinas se detecta en las placentas del ganado infectado con Neospora caninum y es más marcada al principio de la gestación cuando se observa la muerte fetal. Infect Immun (2008) 76:2352–61. doi: 10.1128/IAI.01780-06
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Almería S, Araujo RN, Darwich L, Dubey JP, Gasbarre LC. Expresión génica de citoquinas en la interfaz materno-fetal después de la infección experimental por Neospora caninum de novillas a los 110 días de gestación. Parasite Immunol (2011) 33:517–23. doi: 10.1111/j.1365-3024.2011.01307.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Chen S, Zhou Y, Chen Y, Gu J. Fastp: un preprocesador FASTQ todo en uno ultrarrápido. Bioinformática (2018) 34:i884–90. DOI: 10.1093/Bioinformatics/BTY560
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Andrews S. FastQC: a quality control tool for high throughput sequence data (2010). Disponible en: http://www.bioinformatics.babraham.ac.uk/projects/fastqc.
25. Dobin A, Davis CA, Schlesinger F, Drenkow J, Zaleski C, Jha S, et al. STAR: ultrafast universal RNA-seq aligner. Bioinformática (2013) 29:15–21. DOI: 10.1093/Bioinformatics/BTS635
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Love MI, Huber W, Anders S. Estimación moderada del cambio de pliegue y dispersión para datos de RNA-seq con DESeq2. Genoma Biol (2014) 15:550. DOI: 10.1186/S13059-014-0550-8
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Blighe K, Lun A. PCAtools: PCAtools: everything main components analysis. En: Paquete R versión 2.2.0 (2020). Disponible en: https://github.com/kevinblighe/PCAtools.
28. Marini F, Ludt A, Linke J, Strauch K. GeneTonic: an R/Bioconductor package for streamlining the interpretation of RNA-seq data. BMC Bioinf (2021) 22:610. DOI: 10.1186/S12859-021-04461-5
29. Yu G, Wang L-G, Han Y, He Q-Y. clusterProfiler: un paquete r para comparar temas biológicos entre grupos de genes. OMICS (2012) 16:284–7. doi: 10.1089/omi.2011.0118
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Durinck S, Moreau Y, Kasprzyk A, Davis S, De Moor B, Brazma A, et al. BioMart y bioconductor: un poderoso vínculo entre las bases de datos biológicas y el análisis de datos de microarrays. Bioinformática (2005) 21:3439–40. DOI: 10.1093/Bioinformatics/BTI525
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Carlson M. org. Bt.eg.db: anotación de todo el genoma para bovinos. En: Paquete R versión 3.12.0 (2020). Disponible en: https://bioconductor.org/packages/3.12/data/annotation/html/org.Bt.eg.db.html.
32. Sayols S. Rrvgo: un paquete de bioconductores para reducir y visualizar términos de ontología génica (2020). Disponible en: https://ssayols.github.io/rrvgo.
33. Subramanian A, Tamayo P, Mootha VK, Mukherjee S, Ebert BL, Gillette MA, et al. Análisis de enriquecimiento de conjuntos de genes: un enfoque basado en el conocimiento para interpretar perfiles de expresión de todo el genoma. Proc Natl Acad Sci (2005) 102:15545–50. doi: 10.1073/PNAS.0506580102
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Liberzon A, Birger C, Thorvaldsdóttir H, Ghandi M, Mesirov JP, Tamayo P. La colección de conjuntos de genes de sello distintivo de la base de datos de firmas moleculares (MSigDB). Cell Syst (2015) 1:417–25. doi: 10.1016/j.cels.2015.12.004
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Dolgalev I. Msigdbr: Conjuntos de genes MSigDB para múltiples organismos en un formato de datos ordenado. En: Paquete R versión 7.4.1 (2021). Disponible en: https://igordot.github.io/msigdbr/.
36. Kassambara A, Mundt F. Factoextra: extract and visualize the results of multivariate data analyses. En: Paquete R versión 1.0.7 (2020). Disponible en: https://CRAN.R-project.org/package=factoextra.
37. Newman AM, Steen CB, Liu CL, Gentles AJ, Chaudhuri AA, Scherer F, et al. Determinación de la abundancia y expresión del tipo celular a partir de tejidos a granel con citometría digital. Nat Biotechnol (2019) 37:773–82. DOI: 10.1038/S41587-019-0114-2
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Badia-I-Mompel P, Vélez Santiago J, Braunger J, Geiss C, Dimitrov D, Müller-Dott S, et al. decoupleR: ensemble of computational methods to infer biological activities from omics data. Bioinform Adv (2022) 2:vbac016. doi: 10.1093/bioadv/vbac016
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. García-Alonso L, Holland CH, Ibrahim MM, Turei D, Saez-Rodriguez J. Benchmark and integration of resources for the estimation of human transcription factor activities. Genoma Res (2019) 29:1363–75. doi: 10.1101/gr.240663.118
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Equipo central de R. R: un lenguaje y entorno para la computación estadística. Viena, Austria: R Foundation for Statistical Computing (2020). Disponible en: https://www.R-project.org/.
41. Gu Z, Eils R, Schlesner M. Los mapas de calor complejos revelan patrones y correlaciones en datos genómicos multidimensionales. Bioinformática (2016) 32:2847–9. DOI: 10.1093/Bioinformatics/BTW313
Resumen de PubMed | Texto completo de CrossRef | Google Académico
43. Lex A, Gehlenborg N, Strobelt H, Vuillemot R, Pfister H. UpSet: visualización de conjuntos de intersección. IEEE Trans Vis Comput Graph (2014) 20:1983–92. doi: 10.1109/TVCG.2014.2346248
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Wei T, Simko V. R package «corrplot»: visualización de una matriz de correlación (Versión 0.92) (2021). Disponible en: https://github.com/taiyun/corrplot.
45. Wickham H. ggplot2: gráficos elegantes para el análisis de datos. Nueva York: Springer-Verlag (2016). Disponible en: https://ggplot2.tidyverse.org.
46. Braun L, Brenier-Pinchart M-P, Hammoudi P-M, Cannella D, Kieffer-Jaquinod S, Vollaire J, et al. El efector de Toxoplasma TEEGR promueve la persistencia del parásito mediante la modulación de la señalización NF-κB a través de EZH2. Nat Microbiol (2019) 4:1208–20. DOI: 10.1038/S41564-019-0431-8
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. La inmunidad de tipo Raghupathy R. Th 1 es incompatible con el éxito del embarazo. Immunol Today (1997) 18:478–82. doi: 10.1016/S0167-5699(97)01127-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
48. Entrican G. Regulación inmune durante el embarazo e interacciones huésped-patógeno en el aborto infeccioso. J Comp Pathol (2002) 126:79–94. DOI: 10.1053/JCPA.2001.0539
Resumen de PubMed | Texto completo de CrossRef | Google Académico
49. Quinn HE, Ellis JT, Smith NC. Neospora caninum: ¿una causa del fracaso del embarazo mediado por el sistema inmunitario? Tendencias Parasitol (2002) 18:391–4. doi: 10.1016/S1471-4922(02)02324-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
50. Diniz WJS, Reynolds LP, Ward AK, Borowicz PP, Sedivec KK, McCarthy KL, et al. Desenredando la red genética placentome de novillas de carne en gestación temprana. Genómica (2022) 114:110274. doi: 10.1016/j.ygeno.2022.110274
Resumen de PubMed | Texto completo de CrossRef | Google Académico
51. Nolan SJ, Romano JD, Luechtefeld T, Coppens I. Neospora caninum recluta estructuras de células huésped para su vacuola parasitófora y salva lípidos de orgánulos. Eukaryot Cell (2015) 14:454–73. doi: 10.1128/EC.00262-14
Resumen de PubMed | Texto completo de CrossRef | Google Académico
52. Pernas L, Adomako-Ankomah Y, Shastri AJ, Ewald SE, Treeck M, Boyle JP, et al. El efector de toxoplasma MAF1 media el reclutamiento de las mitocondrias del huésped e impacta la respuesta del huésped. PloS Biol (2014) 12:e1001845. doi: 10.1371/journal.pbio.1001845
Resumen de PubMed | Texto completo de CrossRef | Google Académico
53. García-Sánchez M, Jiménez-Pelayo L, Horcajo P, Regidor-Cerrillo J, Ólafsson EB, Bhandage AK, et al. Respuestas diferenciales de macrófagos derivados de monocitos bovinos a la infección por aislados de Neospora caninum de alta y baja virulencia. Frente Immunol (2019) 10:915. doi: 10.3389/fimmu.2019.00915
Resumen de PubMed | Texto completo de CrossRef | Google Académico
54. Rico-San Román L, Horcajo P, Regidor-Cerrillo J, Fernández-Escobar M, Collantes-Fernández E, Gutiérrez-Blázquez D, et al. Análisis comparativos del proteoma de taquizoíto entre seis aislados de Neospora caninum con diferente virulencia. Intl J Parasitol (2020) 50:377–88. doi: 10.1016/j.ijpara.2020.02.003
55. Ho H, Cheng M, Chiu DT. Glucosa-6-fosfato deshidrogenasa: desde el estrés oxidativo hasta las funciones celulares y las enfermedades degenerativas. Redox Rep (2007) 12:109–18. doi: 10.1179/135100007X200209
Resumen de PubMed | Texto completo de CrossRef | Google Académico
56. Zhang N, Schumacher A, Fink B, Bauer M, Zenclussen AC, Meyer N. Insights into early-pregnancy mechanisms: mast cells and chymase CMA1 shape the phenotype and modulate the functionality of human trophoblast cells, vascular smooth-muscle cells and endothelial cells. Celdas (2022) 11:1158. doi: 10.3390/cells11071158
Resumen de PubMed | Texto completo de CrossRef | Google Académico
57. Almería S, Serrano-Pérez B, Darwich L, Araujo RN, Lopez-Gatius F, Dubey JP, et al. Patrones de respuesta inmune materna y fetal en novillas infectadas experimentalmente con Neospora caninum en el segundo trimestre del embarazo: un estudio descriptivo. Veterinario Parasitol (2014) 204:146–52. doi: 10.1016/j.vetpar.2014.05.006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
58. Kowalski EJA, Li L. Toll-interact protein in solving and non-solving inflammation. Frente Immunol (2017) 8:511. doi: 10.3389/fimmu.2017.00511
Resumen de PubMed | Texto completo de CrossRef | Google Académico
59. Zhang G, Ghosh S. Negative regulation of toll-like receptor-mediated signaling by tollip. J Biol Chem (2002) 277:7059–65. DOI: 10.1074/JBC. M109537200
Resumen de PubMed | Texto completo de CrossRef | Google Académico
60. Parmar N, Chandrakar P, Vishwakarma P, Singh K, Mitra K, Kar S. Leishmania donovani explota tollip, una proteína multitarea, para perjudicar la señalización TLR/IL-1R para su supervivencia en el huésped. J Immunol (2018) 201:957–70. doi: 10.4049/jimmunol.1800062
Resumen de PubMed | Texto completo de CrossRef | Google Académico
61. Du K, Lu F, Xie C, Ding H, Shen Y, Gao Y, et al. La infección por Toxoplasma gondii induce la apoptosis celular a través de múltiples vías reveladas por el análisis del transcriptoma. J Zhejiang Univ Sci B (2022) 23:315–27. DOI: 10.1631/JZUS. B2100877
Resumen de PubMed | Texto completo de CrossRef | Google Académico
62. Rahimi HM, Nemati S, Alavifard H, Baghaei K, Mirjalali H, Zali MR. Soluble total antigen derived from Toxoplasma gondii RH strain prevent apoptosis, but induces anti-apoptosis in human monocyte cell line. Folia Parasitol (Praga) (2021) 68:2021. DOI: 10.14411/FP.2021.026
63. Wang X, Li X, Gong P, Zhang N, Li L, Ouyang H, et al. El verdugo de la piroptosis gasdermin d contribuye a la defensa del huésped y promueve la respuesta inmune Th 1 durante la infección por Neospora caninum. Veterinario Parasitol (2020) 286:109254. doi: 10.1016/j.vetpar.2020.109254
Resumen de PubMed | Texto completo de CrossRef | Google Académico
64. Nikoletopoulou V, Markaki M, Palikaras K, Tavernarakis N. Diafonía entre apoptosis, necrosis y autofagia. Biochim Biophys Acta (2013) 1833:3448–59. doi: 10.1016/j.bbamcr.2013.06.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
65. Vandenabeele P, Galluzzi L, Vanden Berghe T, Kroemer G. Mecanismos moleculares de necroptosis: una explosión celular ordenada. Nat Rev Mol Cell Biol (2010) 11:700–14. DOI: 10.1038/NRM2970
Resumen de PubMed | Texto completo de CrossRef | Google Académico
66. Reister F, Frank H-G, Kingdom JCP, Heyl W, Kaufmann P, Rath W, et al. La apoptosis inducida por macrófagos limita la invasión endovascular del trofoblasto en la pared uterina de las mujeres preeclámpticas. Lab Invest (2001) 81:1143–52. doi: 10.1038/labinvest.3780326
Resumen de PubMed | Texto completo de CrossRef | Google Académico
67. Jiménez-Pelayo L, García-Sánchez M, Regidor-Cerrillo J, Horcajo P, Collantes-Fernández E, Gómez-Bautista M, et al. Susceptibilidad diferencial de líneas celulares carunculares y trofoblastas bovinas a la infección con aislados de alta y baja virulencia. Neospora caninum. Vectores parásitos (2017) 10:463. DOI: 10.1186/S13071-017-2409-9
68. Maley SW, Buxton D, Macaldowie CN, Anderson IE, Wright SE, Bartley PM, et al. Caracterización de la respuesta inmune en la placenta de bovinos infectados experimentalmente con Neospora caninum en gestación temprana. J Comp Pathol (2006) 135:130–41. doi: 10.1016/j.jcpa.2006.07.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
69. Hakimi M-A, Olias P, Sibley LD. Efectores de toxoplasma dirigidos a la señalización y transcripción del huésped. Clin Microbiol Rev (2017) 30: 615–45. doi: 10.1128/CMR.00005-17
Resumen de PubMed | Texto completo de CrossRef | Google Académico
70. Cantón GJ, Katzer F, Benavides-Silván J, Maley SW, Palarea-Albaladejo J, Pang Y, et al. Caracterización fenotípica del infiltrado inmune celular en placentas de bovinos tras inoculación experimental con Neospora caninum en gestación tardía. Vet Res (2013) 44:60. doi: 10.1186/1297-9716-44-60
Resumen de PubMed | Texto completo de CrossRef | Google Académico
71. García-Sánchez M, Jiménez-Pelayo L, Vázquez P, Horcajo P, Regidor-Cerrillo J, Jiménez-Meléndez A, et al. Respuestas inmunes celulares maternas y fetales en madres infectadas con aislados de alta y baja virulencia de Neospora caninum a mediados de la gestación. Célula frontal infectar microbiol (2021) 11:684670. DOI: 10.3389/fcimb.2021.684670
Resumen de PubMed | Texto completo de CrossRef | Google Académico
72. Correia A, Ferreirinha P, Botelho S, Belinha A, Leitão C, Caramalho Í, et al. Papel predominante del interferón-γ en el efecto protector del huésped de las células T CD8+ contra la infección por Neospora caninum. Sci Rep (2015) 5:14913. doi: 10.1038/srep14913
Resumen de PubMed | Texto completo de CrossRef | Google Académico
73. Lu F, Huang S. El papel de los mastocitos en las infecciones parasitarias por protozoos. Frente Immunol (2017) 8:363. doi: 10.3389/fimmu.2017.00363
Resumen de PubMed | Texto completo de CrossRef | Google Académico
74. Park E-A, Han I-H, Kim J-H, Park S-J, Ryu J-S, Ahn M-H. Producción de citoquinas inflamatorias y óxido nítrico por mastocitos humanos incubados con lisado de Toxoplasma gondii. Korean J Parasitol (2019) 57:201–6. doi: 10.3347/kjp.2019.57.2.201
Resumen de PubMed | Texto completo de CrossRef | Google Académico
75. Schoots MH, Bezemer RE, Dijkstra T, Timmer B, Scherjon SA, Erwich JJHM, et al. Distribución de mastocitos deciduales en la restricción del crecimiento fetal y la muerte fetal a término (cercano). Placenta (2022) 129:104–10. doi: 10.1016/j.placenta.2022.10.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
76. Lambert H, Hitziger N, Dellacasa I, Svensson M, Barragan A. Induction of dendritic cell migration upon Toxoplasma gondii infection potentiates parasite diseminion. Cell Microbiol (2006) 8:1611–23. doi: 10.1111/j.1462-5822.2006.00735.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
77. Collantes-Fernández E, Arrighi RBG, Álvarez-García G, Weidner JM, Regidor-Cerrillo J, Boothroyd JC, et al. Las células dendríticas infectadas facilitan la diseminación sistémica y el paso transplacentario del parásito intracelular obligado Neospora caninum en ratones. PloS One (2012) 7:e32123. doi: 10.1371/journal.pone.0032123
Resumen de PubMed | Texto completo de CrossRef | Google Académico
78. Lamouille S, Xu J, Derynck R. Mecanismos moleculares de la transición epitelial-mesenquimal. Nat Rev Mol Cell Biol (2014) 15:178–96. DOI: 10.1038/NRM3758
Resumen de PubMed | Texto completo de CrossRef | Google Académico
79. Oliveira LJ, McClellan S, Hansen PJ. Diferenciación del macrófago endometrial durante el embarazo en la vaca. PloS One (2010) 5:e13213. doi: 10.1371/journal.pone.0013213
Resumen de PubMed | Texto completo de CrossRef | Google Académico
80. Nagamatsu T, Schust DJ. La contribución de los macrófagos a embarazos normales y patológicos. Am J Reprod Immunol (2010) 63:460–71. doi: 10.1111/j.1600-0897.2010.00813.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
81. Muraille E, Leo O, Moser M. Th1/Th2 paradigm extended: macrophage polarization as an unappreciated pathogen-driven escape mechanism? Frente Immunol (2014) 5:603. doi: 10.3389/fimmu.2014.00603
Resumen de PubMed | Texto completo de CrossRef | Google Académico
82. Brown MB, von Chamier M, Allam AB, Reyes L. M1/M2 macrophage polarity in normal and complicated pregnancy. Frente Immunol (2014) 5:606. doi: 10.3389/fimmu.2014.00606
Resumen de PubMed | Texto completo de CrossRef | Google Académico
83. Renaud SJ, Graham CH. El papel de los macrófagos en las interacciones utero-placentarias durante el embarazo normal y patológico. Immunol Invest (2008) 37:535–64. doi: 10.1080/08820130802191375
Resumen de PubMed | Texto completo de CrossRef | Google Académico
84. Jensen KDC, Wang Y, Wojno EDT, Shastri AJ, Hu K, Cornel L, et al. Los efectores polimórficos de Toxoplasma determinan la polarización de los macrófagos y la inflamación intestinal. Cell Host Microbe (2011) 9:472–83. doi: 10.1016/j.chom.2011.04.015
Resumen de PubMed | Texto completo de CrossRef | Google Académico
85. Hooshmandabbasi R, Kazemian A, Zerbe H, Kowalewski MP, Klisch K. Macrófagos en placenta bovina a término: un estudio ultraestructural y molecular. Reprod Domest Anim (2021) 56:1243–53. DOI: 10.1111/RDA.13983
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: Neospora caninum, transcriptoma, placenta bovina, virulencia, respuesta inmune
Cita: Horcajo P, Coronado M, Pastor-Fernández I, Collantes-Fernández E, Rico-San Román L, Reyes-Palomares A y Ortega-Mora L-M (2023) El análisis del transcriptoma completo revela interacciones patógeno-huésped específicas de virulencia en la placenta en la neosporosis bovina. Frente. Immunol. 14:1198609. doi: 10.3389/fimmu.2023.1198609
Recibido: 01 Abril 2023; Aprobado: 27 de junio de 2023;
Publicado: 14 julio 2023.
Editado por:
José Roberto Mineo, Universidad Federal de Uberlandia, Brasil
Revisado por:
Sonia Almeria, Administración de Alimentos y Medicamentos de los Estados Unidos, Estados Unidos
Musa Hassan, Universidad de Edimburgo, Reino Unido
Copyright © 2023 Horcajo, Coronado, Pastor-Fernández, Collantes-Fernández, Rico-San Román, Reyes-Palomares y Ortega-Mora. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Luis-Miguel Ortega-Mora, luis.ortega@ucm.es; Armando Reyes-Palomares, armandorp@ucm.es
†Estos autores han contribuido igualmente a este trabajo y comparten la primera autoría
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

