El análisis del transcriptoma de las secuencias de adenoma y carcinoma
El análisis del transcriptoma de las secuencias de adenoma y carcinoma identifica nuevos biomarcadores asociados con el desarrollo del cáncer colorrectal canino
 Zixiang Lin1,2†
Zixiang Lin1,2†  Jiatong Zhang1†
Jiatong Zhang1†  Qi Chen1
Qi Chen1  Xiaohu Zhang1
Xiaohu Zhang1  Di Zhang1*
Di Zhang1*  Jiahao Lin1*
Jiahao Lin1*  Degui Lin1*
Degui Lin1*- 1Laboratorio Nacional Clave de Seguridad de la Salud Pública Veterinaria, Facultad de Medicina Veterinaria, Universidad Agrícola de China, Beijing, China
- 2Facultad de Ciencia y Tecnología Animal, Universidad de Agricultura de Pekín, Pekín, China
El concepto de transformación de adenoma a cáncer en cáncer en cáncer colorrectal humano (CCR) es ampliamente aceptado. Sin embargo, la relación entre las características del transcriptoma y la transformación del adenoma al carcinoma en caninos no está clara. Recolectamos datos del transcriptoma de 8 tejidos normales de colon, 4 tejidos de adenoma y 15 tejidos cancerosos. El análisis diferencial no pudo determinar los cambios dinámicos de los genes, pero reveló que PFKFB3 puede desempeñar un papel clave en este proceso. El análisis de enriquecimiento explicó la desregulación metabólica, la inmunosupresión y las vías típicas del cáncer en los tumores colorrectales caninos. MFuzz generó patrones específicos de expresión dinámica de cinco genes expresados diferencialmente (DEGs). El análisis de la red de correlación ponderada mostró que los DEG en el grupo 3 estaban asociados con tejidos malignos, lo que revela el papel clave de las vías inflamatorias e inmunitarias en el CCR canino, y también se encontró que la familia de proteínas S100A está involucrada en la transformación maligna de los tumores colorrectales caninos. Al comparar estrategias entre humanos y perros, encontramos cinco marcadores novedosos que pueden ser impulsores del CCR. Entre ellos, GTBP4 mostró una excelente capacidad diagnóstica y pronóstica. Este estudio fue la primera exploración sistemática de la transformación en el CCR canino, complementó las características moleculares del desarrollo y la progresión del CCR canino y proporcionó nuevos biomarcadores potenciales y evidencia oncológica comparativa para estudios de biomarcadores en el cáncer colorrectal humano.
1 Introducción
La tercera causa de muerte relacionada con el cáncer en todo el mundo es el cáncer colorrectal (CCR) (1). En pacientes humanos, la mayoría de los casos de cáncer colorrectal fueron esporádicos, originados por pólipos dentro de recesos anormales (2, 3), y el CCR canino mostró un patrón similar (4-6). La tumorigénesis comenzó con la mutación de las células madre epiteliales intestinales en el colon o la mucosa rectal, luego el adenoma y finalmente el cáncer colorrectal. Durante un período de más de 10 a 15 años, el adenoma tubular se transformó en adenocarcinoma, y las alteraciones en las vías Wnt, RAS, TP53 y PI3K-AKT ocurrieron secuencialmente (7). Los efectos de las vías de señalización Wnt, P53 y TGF-β se han identificado en estudios de CCR canino (8-11). El desarrollo de modelos genéticos de carcinogénesis de múltiples impactos, junto con una comprensión más profunda de la inestabilidad cromosómica, la inestabilidad de los microsatélites y el fenotipo metilador de la isla CpG del CCR (12, 13), ayudará a aclarar las diversas alteraciones genéticas y epigenéticas que subyacen a la secuencia adenoma-carcinoma (14). En el CCR canino, se encontró que los Jack Russell Terriers tenían la misma mutación genética APC de la línea germinal que los humanos, y esta raza se caracterizó por una alta incidencia de cáncer colorrectal hereditario (15, 16). Todos los estudios realizados en esta etapa mostraron un alto grado de similitud entre el CCR en perros y humanos. Y el CCR canino puede revelar nuevos mecanismos moleculares en humanos (11, 17). El cáncer colorrectal canino tiene anomalías en el número de copias similares a las del cáncer colorrectal humano, lo que revela un fuerte grado de homología genética entre los CCR caninos y humanos esporádicos (18). Al igual que en el cáncer colorrectal humano, la sobreactivación de la vía de señalización WNT también está presente en el cáncer colorrectal canino, acompañada de mutaciones repetidas en genes asociados con las vías de señalización CTNNB1 y TGF-β (17). APC fue el gen mutado más significativamente tanto en adenomas caninos como en adenocarcinomas (Frequent Alteration of the Tumor Suppressor Gene APC in Sporadic Canine Colorectal Tumors). La aparición y el desarrollo de tecnologías ómicas de alto rendimiento han facilitado los estudios sistemáticos que comparan tejidos normales y tumorales a nivel de genes, ARNm y proteínas. Cuatro secuencias moleculares de consenso (CMS) de CRC-CMS1 (inmunorasgo, inestabilidad de microsatélites), CMS2 (rasgo canónico), CMS3 (rasgo metabólico) y CMS4 (rasgo mesenquimatoso), y combinaciones transicionales de clasificación de rasgos en distintos subtipos, se establecieron utilizando datos de expresión génica de 4.151 muestras tumorales (19). El análisis del transcriptoma del CCR canino también dilucidó las firmas moleculares específicas de los tumores caninos proliferativos y agresivos, revelando que el cáncer de colon humano CMS4 consistía en dos subtipos, EMT e invasión similar a una cripta, con diferencias en las vías de señalización de TGF-β y el contenido microbiano (17).
Aunque se ha encontrado una variedad de genes expresados diferencialmente (DEG) en el CCR, los patrones de expresión específicos de estos genes en la patogénesis del cáncer colorrectal siguen sin estar claros. Además, los mecanismos por los cuales surgió y evolucionó el CCR no se comprenden completamente. Por lo tanto, es esencial identificar los patrones de expresión génica asociados con el desarrollo y la progresión del CCR.
En este estudio, el análisis del transcriptoma del cáncer normal de colon, adenoma y colorrectal identificó cambios en las vías de señalización durante la formación del tumor colorrectal canino, y el análisis dinámico reveló cinco patrones específicos de expresión dinámica génica. Estos hallazgos se correlacionaron con tres marcadores conocidos de cáncer: la desregulación del metabolismo celular, la evitación de la alteración inmunitaria y la activación de las vías relacionadas con el cáncer. El análisis de WGCNA identificó los módulos genéticos significativamente relacionados con el fenotipo maligno del tumor. Se encontró que los genes en el módulo maligno estaban principalmente regulados al alza, y estos genes afectaron principalmente la inflamación tisular y el proceso inmunológico, revelando así el importante papel de la inmunosupresión en la transformación maligna del CCR canino. Mediante el uso de una estrategia de comparación humano-canina, comparamos genes con un patrón de regulación positiva persistente en el CCR humano. Se identificaron cinco genes principales a través del análisis, y GTPBP4 se examinó adicionalmente como un marcador de pronóstico adverso para el cáncer de colon con una capacidad diagnóstica óptima. Ninguno de estos genes se ha estudiado mucho en tumores humanos y tampoco en tumores caninos. Nuestro análisis y estrategias comparativas identificaron estos nuevos marcadores potenciales que deben ser monitoreados y explorados continuamente durante la formación del tumor.
2 Materiales y métodos
2.1 Adquisición de datos del transcriptoma y análisis diferencial
Se descargaron los datos transcriptómicos (número PRJNA418842, PRJNA396033) de la base de datos de la SRA para tumores colorrectales caninos y tejido normal del colon canino. Para obtener lecturas limpias de alta calidad, se utilizó el software fastp (v0.20.0) para recortar el adaptador y eliminar las lecturas de baja calidad. Utilizamos el software STAR (v2.7.9a) para comparar lecturas limpias de alta calidad con el genoma de referencia Canis_lupus_familiaris (CanFam3.1). Los recuentos de lectura sin procesar de los genes de ARNm se obtuvieron como valores de expresión de ARNm utilizando featurecount (v2.0.2). La normalización se realizó mediante DESeq2 (v1.30.1), conservando solo las lecturas asignadas de forma única para el recuento de HTseq. Se utilizó el método ComBat-seq de R Package sva para eliminar el efecto discontinuo de RNAseq (20).
DEG se identificó utilizando el paquete R DESeq2 con una tasa de falso descubrimiento (FDR) <0.05 y un cambio absoluto log2|fold| > 1. Para la anotación de ARNm se utilizó la base de datos de anotación GTF (Ensembl v104). El análisis de la ontología génica y el enriquecimiento de la vía KEGG de ARNm expresados diferencialmente se realizó utilizando el paquete clusterProfiler R (v3.18.1).
2.2 Análisis de enriquecimiento de conjuntos de genes
El análisis de enriquecimiento de conjuntos genéticos (GESA) se realizó utilizando el software GSEA versión 3.01 con el conjunto de genes de la base de datos de firmas moleculares versión 6.2. Todos los datos de expresión génica se ordenaron fenotípicamente con un número de 1.000. Utilice el umbral de valor FDR (p < 0,05) para identificar las vías correspondientes a los genes enriquecidos en la parte superior o inferior del conjunto de genes y ordenarlos de acuerdo con las puntuaciones de enriquecimiento normalizadas.
2.3 Análisis del modelo de expresión dinámica
El algoritmo central del paquete R «Mfuzz» se basa en Fuzzy C-Means Clustering (FCM), que se utiliza para analizar la tendencia temporal de la expresión génica en los datos del transcriptoma con características de series temporales, y agrupar los genes con patrones de expresión similares. Ayudar a comprender los patrones dinámicos de expresión de estas moléculas biológicas y su conexión con la función. De acuerdo con las instrucciones del paquete R Mfuzz, el análisis de conglomerados blandos se realiza en datos de transcriptoma (21). El agrupamiento de C-means se realizó utilizando R-pack Mfuzz para mapear lecturas por millón (FPKM) de fragmentos de grados identificados por LRT para evaluar los cambios dinámicos en el patrón de expresión.
2.4 Establecimiento de redes de coexpresión e identificación de módulos centrales relacionados con el fenotipo patológico mediante el análisis de redes de coexpresión génica ponderada
El análisis de coexpresión se realizó utilizando el paquete R Weighted Gene Correlation Network Analysis (WGCNA), guiado por tutoriales publicados. Se examinaron genes con FPKM < 1 de 22 muestras y las muestras (tipo de muestra y número de muestras) se agruparon jerárquicamente en función de las distancias euclidianas calculadas a partir de los datos de expresión génica y combinadas con (información clínica del paciente e información de rasgos). El análisis de la topología de la red garantizó una red topológica libre de escala y definió una potencia de umbral suave de (8). Se identificó un total de módulos (número de módulos sin contar los módulos grises) en función de un algoritmo dinámico de tala de árboles con un parámetro minModuleSize de 30 y un parámetro mergeCutHeight de 0,25. Para cada módulo, se identificó el gen de firma (la expresión del primer componente del gen en el módulo) y, a continuación, se calculó la correlación entre el gen de firma y la descripción relacionada con el fenotipo clínico, y se calcularon características clínicas como la estadificación y el grado del tumor. Los genes con alta conectividad en cada módulo se consideraron genes fundamentales. Las relaciones de co-expresión de cada módulo fueron analizadas y visualizadas por Metascape (v3.5.20230101).
2.5 Análisis de la infiltración de células inmunitarias en el CCR
En este estudio, aplicamos el algoritmo CIBERSORT para determinar los subconjuntos de células inmunes de los tejidos caninos. Dado que el algoritmo CIBERSORT es capaz de analizar datos inespecíficos y ruido, es superior a los enfoques convencionales de deconvolución para evaluar la inmunidad infiltrante y para determinar la abundancia de células especializadas en la matriz mixta.
2.6 Validación cruzada con conjuntos de datos humanos
Debido a la falta de bases de datos veterinarias que contengan información sobre tumores colorrectales en perros, seleccionamos las proteínas más relevantes para el CCR humano y las validamos utilizando muestras humanas de la base de datos Bioinformática. Los datos del transcriptoma del cáncer colorrectal humano se obtuvieron de la base de datos GEO (GSE164541). Los datos del transcriptoma para el análisis de ROC y la verificación de la expresión diferencial se derivaron de la base de datos TCGA.2
2.7 Características de funcionamiento del receptor
El paquete pROC se utilizó para el análisis de las características operativas del receptor (ROC) de los datos, y los resultados se visualizaron con ggplot2. El área bajo la curva (AUC) se utiliza a menudo para evaluar las pruebas diagnósticas, y el rango de valores de AUC suele estar entre 0,5 y 1. Cuanto más se acerque el AUC a 1, mejor será el efecto diagnóstico de esta variable en la predicción del resultado.
2.8 Análisis de supervivencia
La curva de Kaplan-Meier es un método gráfico utilizado para presentar los resultados de los análisis de supervivencia y se utiliza comúnmente para analizar la relación entre el tiempo de supervivencia y la incidencia de eventos en los pacientes. Se calcularon automáticamente los puntos de corte óptimos para clasificar a los pacientes en grupos de alta y baja expresión génica y se trazaron curvas de supervivencia empleando el paquete de supervivencia y el paquete survminer en R3.5.3.
2.9 Análisis estadístico
Para evaluar la eficacia pronóstica de los biomarcadores, se generaron las curvas de características operativas del receptor (ROC) en función de los niveles de expresión génica y los tipos de muestra. Se calcularon los valores de p de la prueba de rango logarítmico y se consideró estadísticamente significativo un valor de p de 0,05 o menos. Para realizar el análisis estadístico se utilizaron paquetes de software como GraphPad Prism versión 8.0 o SPSS versión 20.0. La significación estadística se definió como una p de dos colas 0,05.
3 Resultados
3.1 Identificación de DEGs durante la formación del CCR canino
En total, se identificaron 2.578 DEG comparando el adenoma con el colon normal (AN), 3.701 DEG se identificaron comparando el carcinoma con un colon normal (CN), mientras que 14 DEG se identificaron comparando el carcinoma con el adenoma (AC), lo que sugiere que los adenomas eran más similares al carcinoma que a los normales. Se encontraron 3.750 DEG después de las comparaciones de los tres tipos de tejido (ACN) por LRT (DEseq2) (Tablas suplementarias S1-S4). En la figura 1A se muestran los DEG superpuestos entre los tejidos normales de colon, adenoma y carcinoma. PFKFB3 fue el único gen que se cruzó. Aunque se pueden realizar comparaciones entre dos tipos de tejido para identificar los DEG que exhiben una regulación relativa al alza o a la baja, es difícil identificar cambios en los patrones de expresión a lo largo de la secuencia adenoma-carcinoma.
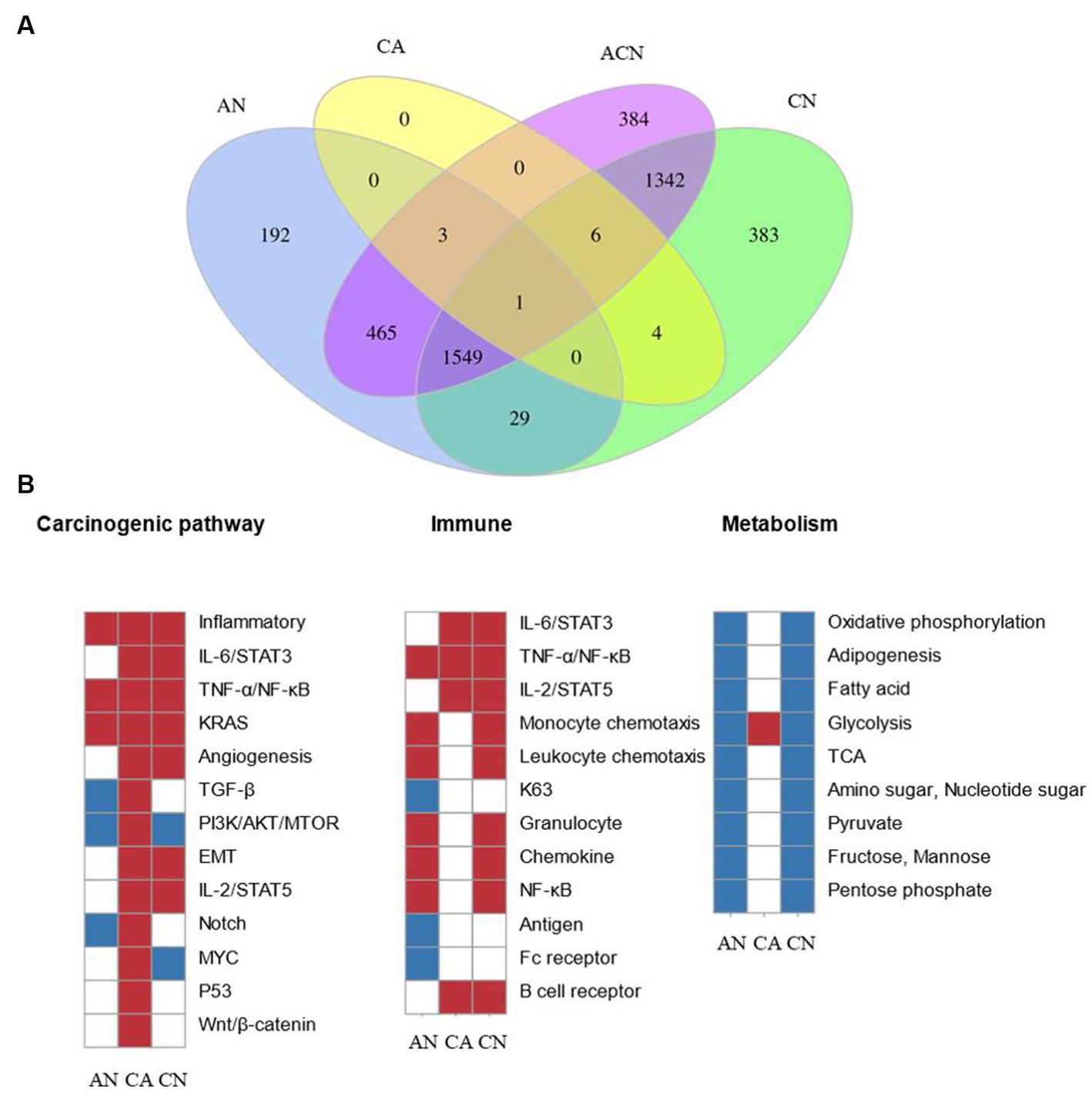 Figura 1. Comparación de los datos del transcriptoma en tres tipos de DEG identificados por tumor y características moleculares de la formación de CCR. N: tejido normal; A: tejido de adenoma; C: tejido carcinoma; AN: adenoma vs. colon normal; AC: carcinoma vs. adenoma; CN: carcinoma vs. colon normal; ACN, tres tipos de tejidos. (A) Diagrama de Venn que muestra la superposición de DEG identificados por comparaciones AN (azul), CA (amarillo), CN (verde) y ACN (morado). (B) Mapa de calor de GSEA que muestra cambios significativos en la actividad de la vía canónica, las actividades metabólicas e inmunológicas durante la formación del CCR.
Figura 1. Comparación de los datos del transcriptoma en tres tipos de DEG identificados por tumor y características moleculares de la formación de CCR. N: tejido normal; A: tejido de adenoma; C: tejido carcinoma; AN: adenoma vs. colon normal; AC: carcinoma vs. adenoma; CN: carcinoma vs. colon normal; ACN, tres tipos de tejidos. (A) Diagrama de Venn que muestra la superposición de DEG identificados por comparaciones AN (azul), CA (amarillo), CN (verde) y ACN (morado). (B) Mapa de calor de GSEA que muestra cambios significativos en la actividad de la vía canónica, las actividades metabólicas e inmunológicas durante la formación del CCR.
3.2 Las etapas tempranas y tardías de la aparición del CCR canino se relacionaron con tres características distintivas
Se utilizó GSEA para comparar los patrones del transcriptoma de cada uno de los tres tejidos diferentes. Después de revisar los conjuntos de genes publicados anteriormente relacionados con la actividad de la vía canónica y los procesos metabólicos e inmunológicos, se eligieron conjuntos de genes enriquecidos (p < 0,05) para producir mapas de calor, como se muestra en la Figura 1B.
Las etapas de formación tumoral generalmente se dividieron en la etapa temprana o la etapa avanzada. La etapa temprana se refería a la transformación de tejido normal en un adenoma, y la etapa tardía se refería a la transformación de tejido de adenoma en carcinoma. La mayoría de las vías típicas se activaron durante la transición de las células epiteliales normales del colon a los adenomas y al carcinoma, pero solo las vías TGF-β, PI3K/AKT/mTOR y Notch se regularon a la baja durante la transición normal a adenoma y se activaron durante la transición del adenoma al carcinoma. Las vías inflamatoria, TNF-α/NF-κB, así como KRAS, se activaron tempranamente durante la transformación de normal a adenoma, y la vía IL-6/STAT3, angiogénesis, EMT, IL-2/STAT5, p53 y Wnt/β-catenina se activaron tardíamente durante la transformación del adenoma en carcinoma.
La actividad metabólica desregulada fue una característica de la formación tumoral. GSEA mostró que la actividad de la mayoría de las vías relacionadas con el metabolismo disminuyó durante la formación del tumor. En particular, la glucólisis aumentó durante la transformación de los adenomas en cancerosos.
Durante la transición de adenoma a adenocarcinoma, los genes que afectan la transición epitelio-mesenquimal (EMT) se regularon significativamente al alza. En segundo lugar, los genes relacionados con la angiogénesis se regularon significativamente al alza, lo que fue consistente con las características biológicas del tumor. Además, muchas vías clásicas fueron reguladas al alza, incluida la respuesta de los genes regulados por NF-κB al TNF-α, la regulación al alza del gen STAT5 bajo la estimulación de IL2 y la regulación al alza de los genes después de la activación de la vía de señalización Notch, p53 y Wnt.
En el desarrollo y propagación de las neoplasias malignas, el efecto del sistema inmunológico era indispensable. Durante la transición se activaron vías de señalización estrechamente relacionadas con la inmunidad, como IL-6/STAT3, TNF-α/NF-κB e IL-2/STAT5.
Los resultados de la GSEA mostraron que las vías típicas, el metabolismo y las respuestas inmunitarias variaban con el tiempo, pero no siempre eran consistentes. Las diferentes vías sufrieron una variedad de modificaciones en varias transiciones, algunas de las cuales se involucraron durante el cambio de normal a adenoma y otras involucradas en la transición de adenoma a carcinoma. Posteriormente, investigamos los patrones de expresión de DEG durante la formación del tumor para arrojar más luz sobre estos hallazgos.
3.3 Identificación de patrones dinámicos de expresión de procesos de adenoma a carcinoma
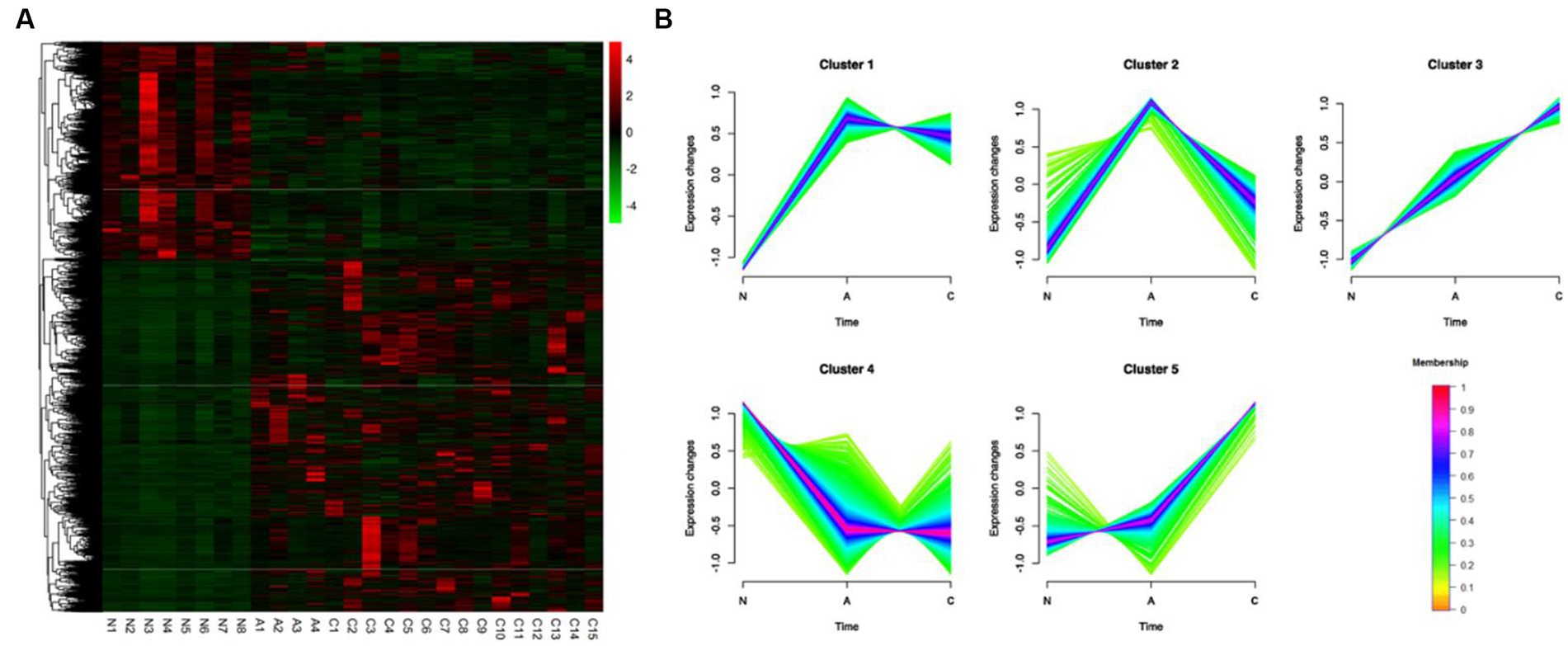
A partir de la comparación de los DEGs descubiertos por los tres tipos de tejidos, se llevó a cabo un agrupamiento jerárquico no supervisado y se crearon mapas de calor de 3.750 DEGs en 27 muestras para comprender los cambios dinámicos de los DEGS en las tres fases de formación del CCR. Como se ve en la Figura 2A, varios tipos de tejidos se enriquecieron para grupos de genes con varios grados de expresión. Utilizando Mfuzz, los 3.750 DEG se dividieron en cinco grupos con el fin de evaluar los patrones dinámicos de expresión de los DEG durante el inicio del CCR (Figura 2B) (Tabla Suplementaria S5). El patrón de expresión génica del grupo 5 cambió significativamente en la etapa posterior de la formación del tumor, mientras que la regulación positiva de los DEG solo ocurrió durante la transformación del adenoma en neoplasia maligna. Mientras que los DEG solo se regulaban al alza o a la baja durante la transición de los adenomas, el patrón de expresión de los DEG en los grupos 1 y 4 cambió considerablemente durante las primeras etapas de la formación del tumor. Durante el cambio de tejido normal a tejido canceroso, los DEG en el grupo 3 continuaron aumentando de manera constante (Figura 2B).
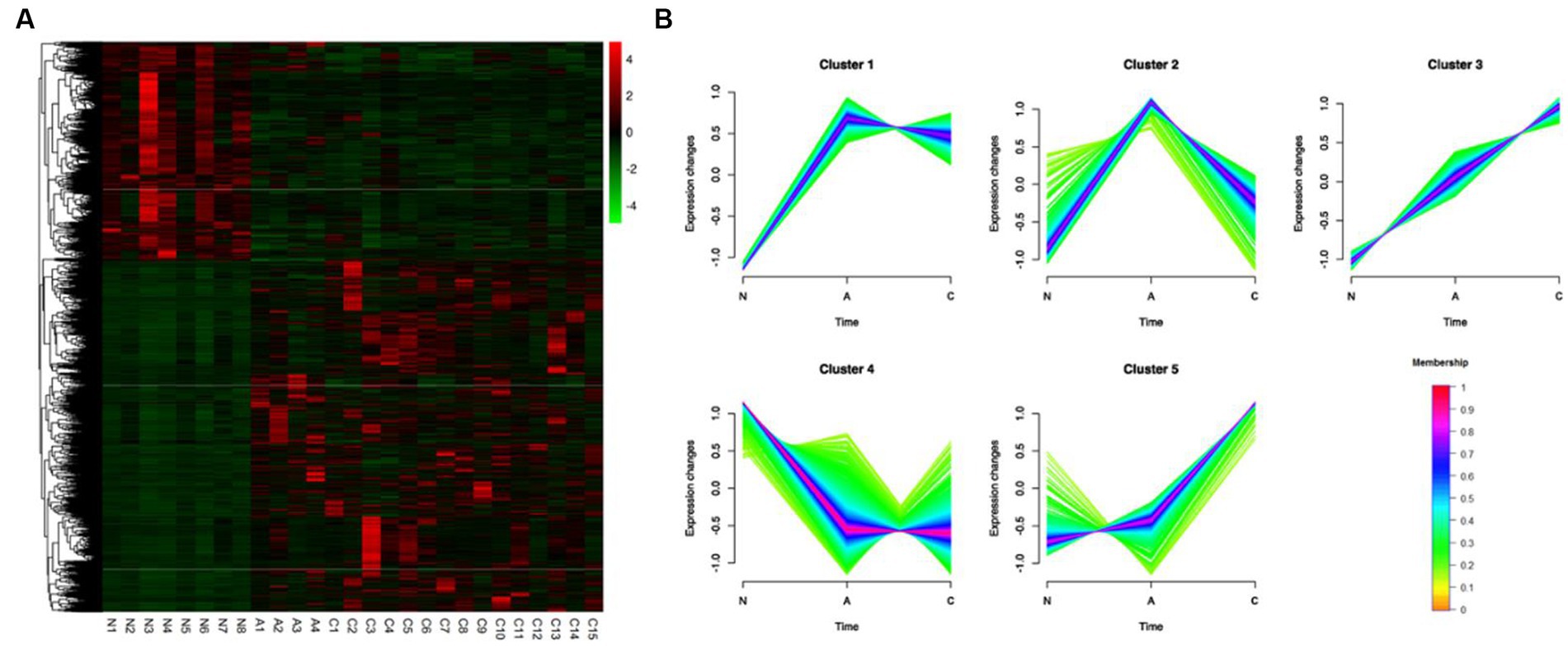 Figura 2. Identificación de 3.750 DEGs, perfiles de expresión dinámica y características químicas a partir de la comparación de ACN. (A) Se mapearon 3.750 DEG y se agruparon jerárquicamente. N, normal; A: adenoma; C: carcinoma. (B) Los diagramas mostraron los patrones de cambios dinámicos en los DEG que se descubrieron utilizando Mfuzz durante el desarrollo del CCR.
Figura 2. Identificación de 3.750 DEGs, perfiles de expresión dinámica y características químicas a partir de la comparación de ACN. (A) Se mapearon 3.750 DEG y se agruparon jerárquicamente. N, normal; A: adenoma; C: carcinoma. (B) Los diagramas mostraron los patrones de cambios dinámicos en los DEG que se descubrieron utilizando Mfuzz durante el desarrollo del CCR.
3.4 Análisis de enriquecimiento asociado a patrones de expresión dinámica
Planteamos la hipótesis de que los diferentes patrones de expresión de los genes sirvieron para diversos propósitos a lo largo del desarrollo de los CCR. El análisis GO y KEGG de los cinco grupos reveló rasgos distintivos de los genes en cada grupo.
El análisis de GO reveló que los genes del grupo 1, que estaban regulados al alza durante la transición normal a adenoma, se asociaron con la inflamación y la inmunidad, incluida la quimiotaxis de neutrófilos, la respuesta inflamatoria, la vía de señalización mediada por quimiocinas, la respuesta celular a la interleucina-1 y la vía de señalización mediada por la interleucina-17. Se encontraron resultados similares mediante el análisis KEGG, que mostró que los genes estaban principalmente involucrados en la interacción del receptor de citocinas a citocinas, las vías en el cáncer, la vía de señalización de quimiocinas, la enfermedad inflamatoria intestinal y la vía de señalización MAPK (Figura suplementaria S1).
Los genes del grupo 4, que se regulaban a la baja durante la transición normal a adenoma, se asociaron principalmente con el metabolismo (los términos GO incluían el transporte transmembrana de iones de hidrógeno, el ciclo del ácido tricarboxílico, el proceso metabólico de los lípidos, la betaoxidación de ácidos grasos, la respiración aeróbica, la síntesis mitocondrial de ATP acoplada al transporte de protones, el proceso biosintético del colesterol). El análisis de KEGG indicó que los genes del grupo 4 estaban involucrados en vías asociadas con las vías metabólicas, el metabolismo del carbono, el ciclo del citrato (ciclo del TCA), la fosforilación oxidativa, la carcinogénesis química – especies reactivas de oxígeno, entre otras (Figura suplementaria S4).
Como se muestra en la Figura 2D, los genes del grupo 5, que se regularon al alza durante la transición del adenoma al carcinoma, estaban principalmente implicados en las vías canónicas asociadas con el cáncer, incluidas las vías de señalización Ras, PI3K-Akt, Proteoglicanos en el cáncer, MAPK e Hippo, entre otras (Figura suplementaria S5).
El análisis de GO reveló que los genes del grupo 3, que se regulaban monótonamente al alza durante la secuencia normal-adenoma-carcinoma, se asociaron con la inmunidad y el cáncer, incluida la respuesta inflamatoria, la regulación positiva de la producción de citocinas, la transducción de señales, la vía de señalización mediada por citocinas de quimiotaxis, las respuestas inflamatorias reguladas positivamente, la producción de IL-6 y la señalización célula-célula. Se extrajeron conclusiones similares del análisis de KEGG, que reveló que los genes estaban involucrados principalmente en las vías de señalización de NF-κB, señalización de IL-17, señalización de TNF, vías en el cáncer y señalización de JAK-STAT. La Figura Suplementaria S3 muestra los resultados del análisis GO y KEGG de los otros conglomerados (Figura Suplementaria S3).
3.5 Construcción de la red de coexpresión e identificación de módulos centrales relacionados con el fenotipo patológico mediante el análisis de la red de coexpresión génica ponderada
Se aplicó WGCNA a 12.866 genes con una expresión de al menos 0,1 FPKM con el fin de investigar la asociación entre los DEG y la fisiopatología de los tejidos. Este proceso produjo 33 módulos de genes altamente coexpresados. Tres de los DEG de los módulos tuvieron una fuerte correlación con el fenotipo tisular. Los DEG se relacionaron estrechamente con la malignidad en el módulo verde oscuro (cor = 0,57, p = 0,002) (Tabla suplementaria S6), benigna en el módulo amarillo claro (cor = 0,65, p = 3e-04) y los tejidos normales en el módulo turquesa (cor = 0,75, p = 6e-06) (Figura 3A). Los patrones de expresión de DEG fueron dinámicos en estos tres módulos. Los grupos 4 (1,261/2684) en el módulo turquesa se enriquecieron en DEG que se unieron a tejidos de adenomas. Los genes en el módulo verde oscuro que se unieron a tejidos malignos fueron más prevalentes en los grupos 1 (15/150), 3 (44/150) y 5 (25/150).
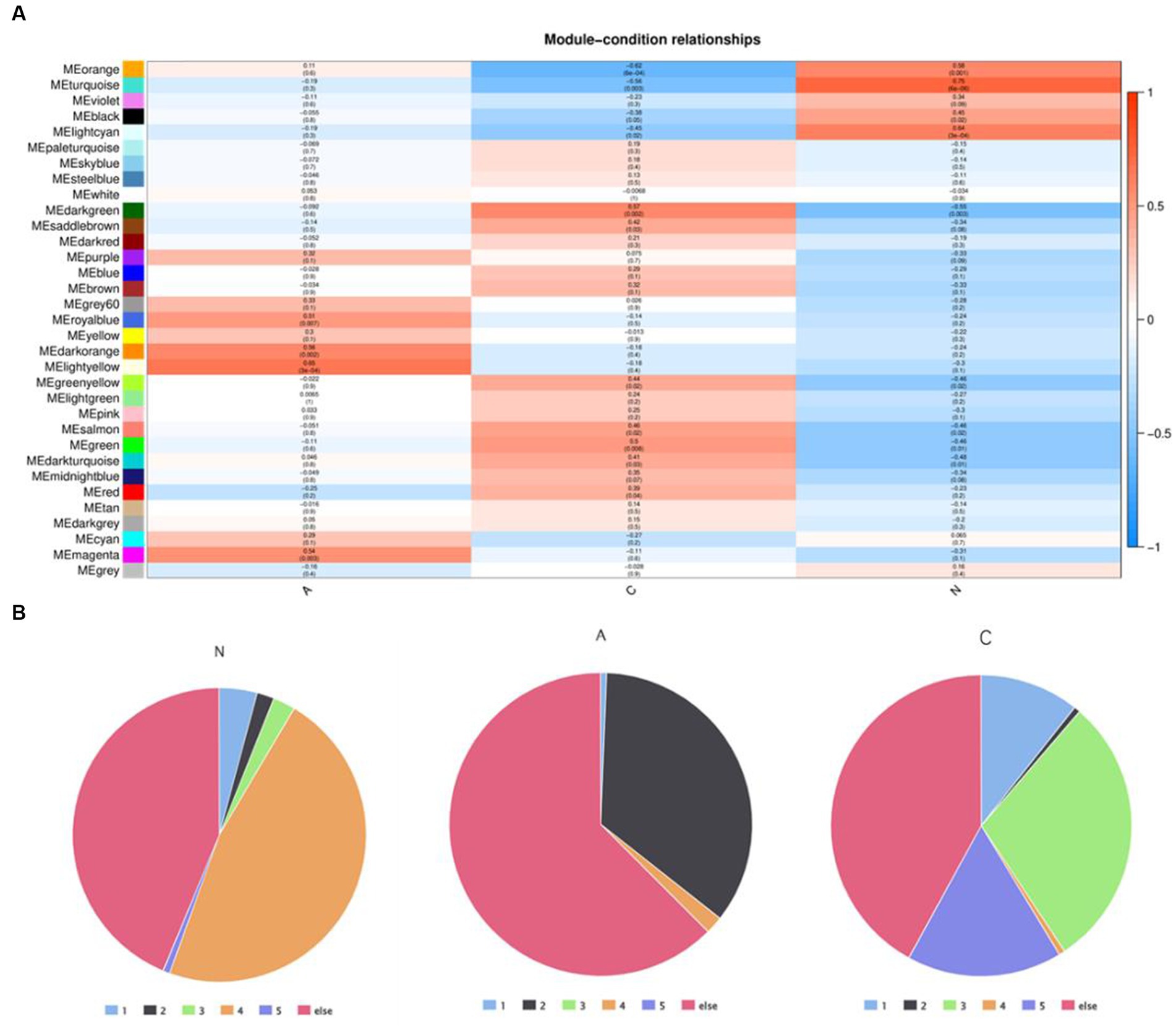 Figura 3. (A) El análisis de la red de coexpresión génica ponderada identificó los genes del módulo estrechamente relacionados con los fenotipos patológicos del tumor y (B) la distribución de DEGS en los genes del módulo central en cinco modos de expresión dinámica.
Figura 3. (A) El análisis de la red de coexpresión génica ponderada identificó los genes del módulo estrechamente relacionados con los fenotipos patológicos del tumor y (B) la distribución de DEGS en los genes del módulo central en cinco modos de expresión dinámica.
Los genes expresados diferencialmente en el grupo 4 pueden desempeñar un papel clave en la progresión del tejido normal a adenoma. La proporción de genes expresados diferencialmente en el grupo 4 es mayor en el módulo normal, pero la proporción tanto en los módulos benignos como en los malignos es muy pequeña. Mientras tanto, la proporción de genes en el grupo 2 es muy pequeña tanto en los módulos normales como en los malignos, pero la proporción en el módulo benigno es muy alta. Esto sugiere que los genes de los grupos 2 y 4 podrían estar involucrados en los eventos tempranos esenciales de la formación tumoral, pero pueden no ser los grupos clave que afectan la progresión tumoral.
Los genes de los grupos 3 y 5 presentaron la mayor proporción en el módulo maligno y una baja proporción tanto en el módulo normal como en el benigno, lo que sugiere que estos genes podrían ser cruciales para el eventual deterioro de la organización.
A través del análisis de este enriquecimiento génico, este hallazgo implica que en el desarrollo del cáncer colorrectal en perros, el primero es la desregulación de la actividad metabólica (reprogramación metabólica), combinada con la pérdida de proliferación celular y conexiones estrechas, y el desarrollo de tejidos normales en adenomas. En el desarrollo posterior, la vía de señalización Wnt, la vía de señalización IL-6/STAT3, los cambios en el microambiente inmunitario tumoral y la transformación epitelio-mesenquimal desempeñan un papel importante en el proceso de transformación maligna de los tejidos.
3.6 Patrones dinámicos de expresión asociados al fenotipo patológico en la secuencia adenoma-carcinoma
Los cambios monótonos que siguieron a la formación del CCR indicaron que estos genes pueden ser adecuados como biomarcadores de seguimiento específicos o factores determinantes. Los DEG del grupo 3 se seleccionaron para su posterior análisis como marcadores de formación de CCR. El diagrama de Venn identificó 44 genes que estaban consistentemente regulados al alza en los módulos significativamente relacionados con la malignidad de los tumores, y las proteínas representadas por estos genes tenían interacciones complejas (Figuras 4A, B). El análisis de enriquecimiento encontró que estos genes estaban relacionados principalmente con la inflamación y la inmunidad, lo que sugiere que durante la aparición y el desarrollo del cáncer colorrectal en perros, los cambios en el microambiente inmunológico del tumor pueden desempeñar el papel más importante. A través de los módulos centrales identificados por MCODE, encontramos dos comunidades principales, una de las cuales es la comunidad de la función inmune con IL6 como núcleo, y la otra es la comunidad con la familia de proteínas S100 como núcleo (Figura 4C). Las funciones biológicas de estos genes centrales se han estudiado ampliamente en las enfermedades tumorales humanas, pero es necesario explorarlas más a fondo en los tumores colorrectales humanos y caninos. Se utilizó Cibersort para analizar la infiltración de células inmunitarias en los tres tipos de tejidos. Se encontró que la infiltración de monocitos y neutrófilos en los tejidos tumorales aumentó, y la disminución de las células NK activadas, la disminución de las células CD8T y el aumento de las células T reguladoras sugirieron el fenómeno de inmunosupresión en los tumores colorrectales caninos (Figura suplementaria S6).+
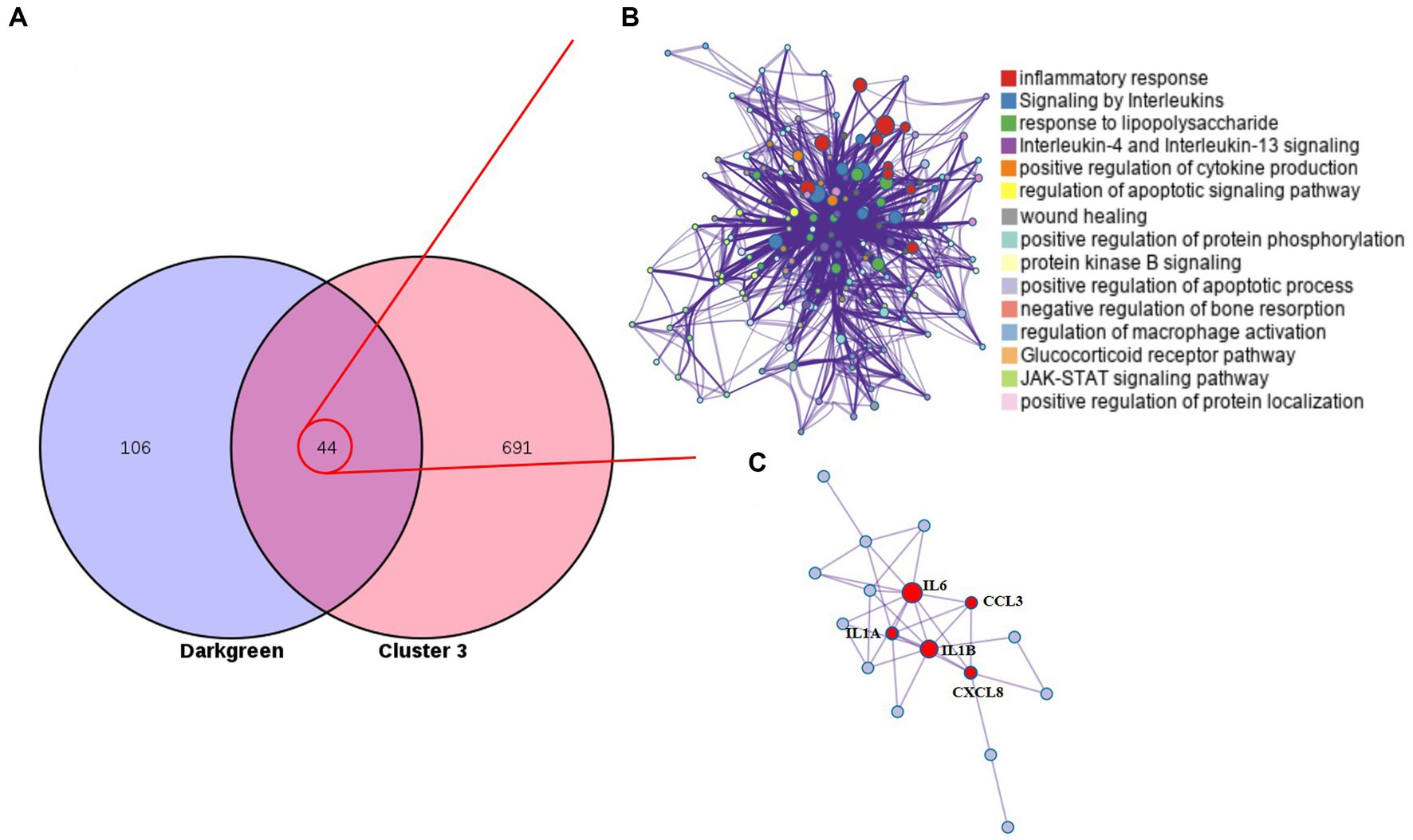 Figura 4. Agrupe 3 genes en el módulo central. (A) El diagrama de Venn encontró 44 genes superpuestos. (B) Red PPI y análisis de enriquecimiento de 44 genes. (C) Los genes centrales identificados por MCODE.
Figura 4. Agrupe 3 genes en el módulo central. (A) El diagrama de Venn encontró 44 genes superpuestos. (B) Red PPI y análisis de enriquecimiento de 44 genes. (C) Los genes centrales identificados por MCODE.
3.7 La estrategia de comparación entre perros y humanos revela genes centrales
Basado en una nueva estrategia para comparar humanos y perros entre especies, podría ayudar a aclarar un objetivo central de la investigación del cáncer, a saber, las distinciones entre conductor y pasajero del cáncer. Comparamos el conglomerado 3 con conglomerados con el mismo patrón de expresión en el patrón dinámico del adenoma colorrectal humano y obtuvimos 42 genes diferenciales (Figura 5A). Estos genes diferenciales se basan en comparaciones entre especies y es más probable que impulsen el desarrollo del cáncer. El análisis de enriquecimiento de 42 genes diferenciales reveló una variedad de procesos biológicos estrechamente relacionados con el cáncer. A nivel metabólico, implica la biosíntesis, descomposición y metabolismo de los ácidos grasos. Está implicado en la diferenciación de TH17, la vía de señalización de IL17 y la vía de señalización de citoquinas a nivel inmunitario. Además, están implicadas varias vías de señalización clásicas relacionadas con el cáncer, como el receptor tipo toll, la quimiocina, el Wnt, el TGF-β y las vías de señalización MAPK (Figura 5B). Se construyó una red PPI de 42 genes diferenciales de acuerdo con la base de datos STRING, y se obtuvieron 5 genes centrales que pueden desempeñar un papel clave mediante un análisis adicional de MCODE (Figura 5C).
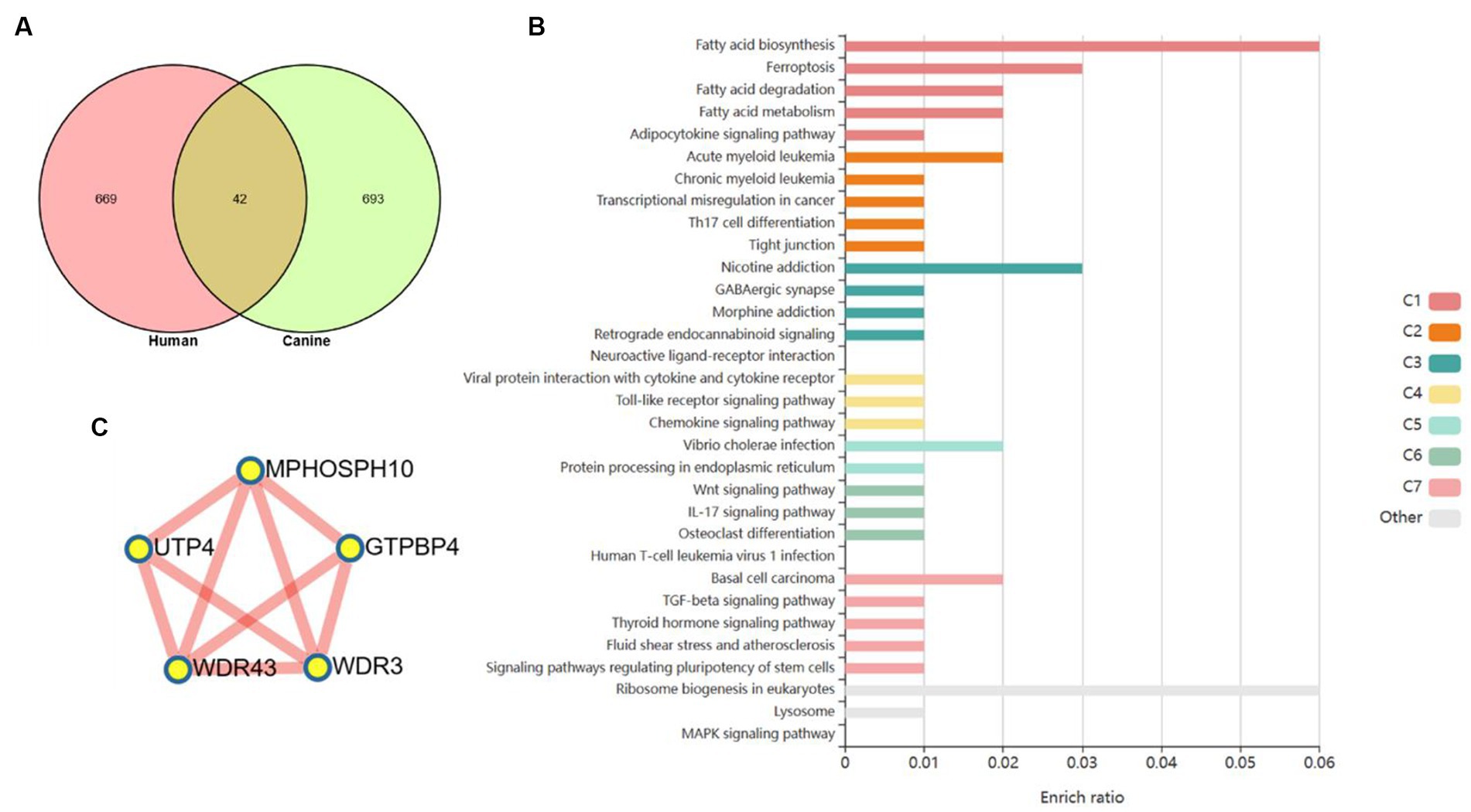 Figura 5. Los genes centrales se examinaron en función de la estrategia comparativa entre humanos y perros. (A) Diagrama de Venn para obtener los genes centrales. (B) El análisis de enriquecimiento revela una variedad de vías y procesos biológicos. (C) La red PPI se utilizó para descartar más genes diferenciales centrales.
Figura 5. Los genes centrales se examinaron en función de la estrategia comparativa entre humanos y perros. (A) Diagrama de Venn para obtener los genes centrales. (B) El análisis de enriquecimiento revela una variedad de vías y procesos biológicos. (C) La red PPI se utilizó para descartar más genes diferenciales centrales.
3.8 Los análisis diagnósticos y de supervivencia revelaron la eficacia de cinco marcadores potenciales
Utilizando los datos de expresión y los datos de supervivencia de la base de datos TCGA, evaluamos la utilidad diagnóstica y pronóstica de los cinco DEG. La probabilidad de SG se calculó mediante un análisis de Kaplan-Meier y se compararon los grupos con diversos grados de expresión génica mediante la prueba de rango logarítmico. Las altas expresiones de GTPBP4 se correlacionaron con una mala supervivencia (Figura 6A). El análisis de la curva ROC mostró que todos ellos podrían usarse como biomarcadores precisos para ayudar en la identificación de tumores colorrectales. Y GTPBP4 tuvo la puntuación más alta de AUC y el mejor efecto diagnóstico (Figura 6B).
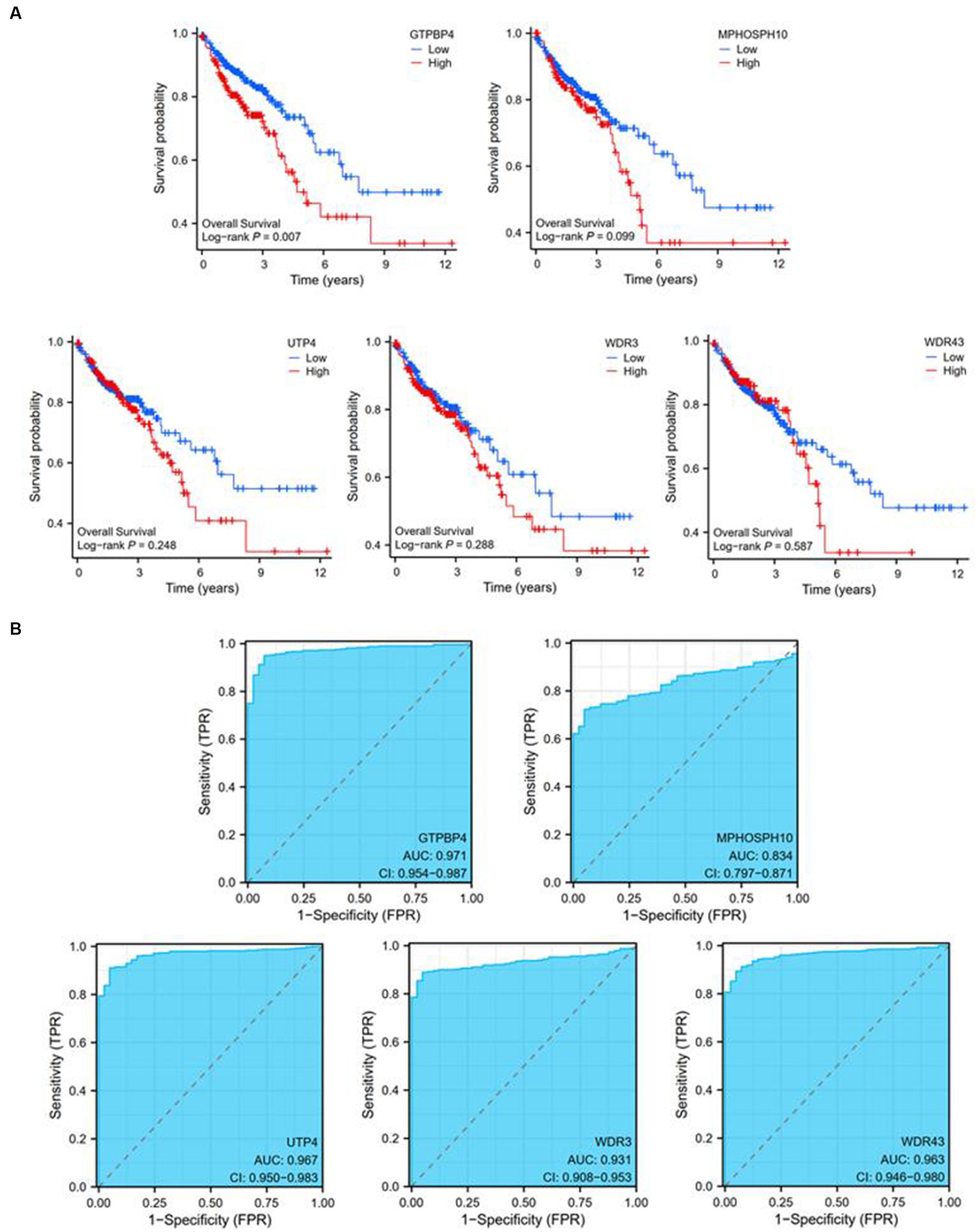 Figura 6. Pruebas de capacidad diagnóstica y pronóstica de cinco genes. (A) Curva de supervivencia de GTPBP4, MPHOSPH10, UTP4, WDR3 y WDR43. (B) Curva ROC de GTPBP4, MPHOSPH10, UTP4, WDR3 y WDR43.
Figura 6. Pruebas de capacidad diagnóstica y pronóstica de cinco genes. (A) Curva de supervivencia de GTPBP4, MPHOSPH10, UTP4, WDR3 y WDR43. (B) Curva ROC de GTPBP4, MPHOSPH10, UTP4, WDR3 y WDR43.
4 Discusión
Este proceso evolutivo del adenoma-carcinoma es ampliamente aceptado en los estudios de cáncer colorrectal humano (22). Sobre la base de las numerosas homologías moleculares y las características clínicas de los tumores colorrectales en humanos y perros, es probable que los tumores colorrectales caninos sigan este proceso evolutivo (23). El patrón de expresión dinámica del cáncer colorrectal humano se ha estudiado ampliamente, pero no se ha informado en perros.
Encontramos que en adenomas y carcinomas, la expresión del número de activación génica es generalmente mayor que el número de supresión de la expresión génica y cuatro tipos de DEG eventualmente involucran PFKFB3, lo que despertó nuestro interés. Dirigirse a los flujos glucolíticos mediante el metabolismo de PFKFB3 para el tratamiento de cánceres dependientes de glucosa. PFKFB3 aumentó la IL-1β y el TNF-α en las células epiteliales intestinales para promover la tumorigénesis del cáncer colorrectal relacionado con la colitis. La interleucina-6 estimuló la glucólisis aeróbica mediante la regulación de PFKFB3 en las primeras etapas del cáncer colorrectal (24). Mir-488 alivió la resistencia química y la glucólisis en el cáncer colorrectal al dirigirse a PFKFB3 (25). No hay estudios que exploren el papel biológico de PFKFB3 en los tumores colorrectales caninos, ni siquiera un solo artículo sobre temas caninos, lo cual es una buena dirección para futuras exploraciones.
Los genes del grupo 5 de los cinco patrones de expresión dinámica solo se alteraron cuando un adenoma se convirtió en un carcinoma, y se enriquecieron especialmente en la vía habitual del cáncer. Cualquier gen que exhiba el patrón de expresión dinámica del grupo 5 durante el desarrollo de un tumor puede ser crucial para el proceso de transformación maligna. Esta metodología podría facilitar la búsqueda de nuevas dianas para la transformación cancerosa. El análisis de GO y KEGG encontró que la desregulación inmunitaria desempeñaba un papel clave en el desarrollo de tumores. Muchas vías inmunitarias se relacionaron principalmente con la inmunidad innata. En las últimas décadas, la investigación sobre la inmunidad tumoral ha ignorado la contribución de la inmunidad innata frente a la adaptativa. Además, cada vez más datos indican que la inmunidad innata puede ser crucial para los efectos cancerígenos del cáncer de intestino (26). Por lo tanto, la aplicación de sistemas inmunes adaptativos e innatos para luchar contra las células del CCR podría superar el problema de la especificidad, que fue una preocupación importante en la quimioterapia y la radioterapia (27).
Entre los cinco patrones de expresión dinámica, los genes del grupo 5 solo se modificaron durante la transición del adenoma al cáncer y se enriquecieron en la vía típica del cáncer. Cualquier gen que siga el patrón de expresión dinámica del grupo 5 durante la formación del tumor puede desempeñar un papel clave en la transformación maligna. Este modelo puede ayudar a facilitar el descubrimiento de nuevas dianas para la transformación maligna. El análisis de GO y KEGG encontró que la desregulación inmunitaria desempeñaba un papel clave en el desarrollo de tumores. Muchas vías inmunitarias se relacionaron principalmente con la inmunidad innata. En las últimas décadas, la investigación sobre la inmunidad tumoral ha ignorado la contribución de la inmunidad innata frente a la adaptativa. Cada vez hay más pruebas que sugieren que la inmunidad innata puede desempeñar un papel importante en los efectos cancerígenos del cáncer de colon (26). Por lo tanto, la aplicación de sistemas inmunes adaptativos e innatos para luchar contra las células del CCR podría superar el problema de la especificidad, que fue una preocupación importante en la quimioterapia y la radioterapia (27).
En combinación con el análisis de WGCNA, encontramos que los genes relacionados con el sistema inmunitario pueden desempeñar un papel clave en la progresión maligna de los tumores colorrectales. Como objetivo ponderado, la IL-6 se ha estudiado ampliamente en el cáncer colorrectal humano. También nos sorprendió descubrir que la familia de proteínas S100A desempeñaba un papel importante en el desarrollo maligno de los tumores colorrectales caninos.
Las estrategias basadas en comparaciones entre especies tienen más probabilidades de identificar los factores que impulsan el cáncer, y seguimos la nueva estrategia de comparaciones entre humanos y perros. De hecho, existe un patrón de expresión génica similar en el cáncer colorrectal humano, y es más probable que la comunidad de genes regulada al alza desempeñe un papel importante en el cáncer. A través de un cribado comparativo, encontramos 42 genes diferenciales comunes, y el análisis de enriquecimiento encontró que estos genes tienen una amplia gama de funciones biológicas relacionadas con las vías clásicas del cáncer, el metabolismo y la inmunidad están cubiertos.
Se identificaron cinco genes principales mediante un análisis más detallado de los 42 genes, que eran GTPBP4, MPHOSPH10, UTP4, WDR3 y WDR43.
Un regulador crítico de la progresión del ciclo celular y la activación de MAPK es la proteína de unión a trifosfato de guanosina 4 (GTPBP4), un miembro de la familia GTPBP que está altamente conservado en todos los eucariotas, desde la levadura hasta los humanos (28). Se demostró que GTPBP4 promovió el crecimiento y la metástasis del carcinoma hepatocelular (CHC) tanto in vivo como in vitro y promovió la glucólisis aeróbica al inducir la formación de piruvato quinasa dimérica M2 (PKM2) (29). Se detectó un nivel más alto de GTPBP4 en los tejidos metastásicos del CCR, y se ha demostrado que GTPBP4 promueve la metástasis del CCR al interrumpir la actividad de RhoA (30). GTPBP4 ha demostrado efectos superiores en el diagnóstico y la supervivencia a través del análisis de supervivencia del cáncer colorrectal humano y las pruebas diagnósticas.
La fosfoproteína 10 (MPHOSPH10) en fase M pertenecía a categorías de respuesta fisiológica celular y transducción de señales (31), sin embargo, su papel en el progreso tumoral aún ha sido poco estudiado. Se encontró que la hipoxia regulaba marcadamente a la baja los genes relacionados con la supervivencia celular, como MPHOSPH10, IMP-3, ITGA2, SDCBP e IGBP3 en células SK-N-MC (32).
Uno de los subcomplejos en el processoma de subunidad pequeña (SSU), las proteínas U tres (Utps), fue esencial para la formación de la cromatina ribosómica (r-cromatina) y para la producción eficiente de ADNr (33). Siete proteínas formaban el subcomplejo t-Utp: Utp4, Utp5, Utp8, Utp9, Utp10, Utp15 y Utp17 (34). Utp4 era un complejo ribonucleoproteico necesario para el procesamiento del ARN ribosómico y el ensamblaje de subunidades pequeñas (35).
El dominio de repetición de triptófano-aspartato (WDR) participó en un gran número de procesos celulares, como el sistema ubiquitina-proteasoma (UPS), la vía de señalización del receptor acoplado a la proteína G, la percepción y reparación del daño en el ADN, la regulación epigenética de la cromatina y el sistema inmunológico (36). El dominio de repetición WD 3 (WDR3), también conocido como DIP2 o UTP1, un miembro de la familia de repeticiones WD, participó en una serie de procesos celulares, incluida la transmisión de señales, la apoptosis, la regulación génica y la progresión del ciclo celular (37, 38). Al interactuar con GATA4, WDR3 activó la vía de señalización de Hippo, demostrando que era crucial para promover el avance del cáncer de páncreas (39). Además, se encontró que los tejidos de cáncer de próstata (CaP) tenían un nivel sustancialmente más alto de WDR3, y la sobreexpresión de WDR3 aumentaba los marcadores de características similares a las células madre (40). Estos estudios anteriores mostraron que WDR3 puede ayudar a algunos cánceres malignos a crecer y proliferar, sin embargo, la función biológica de WDR3 en el CCR y su mecanismo asociado aún se desconocen.
El dominio de repetición 43 de triptófano-aspartato (WDR43), el ortólogo de la levadura Utp5, podría interactuar con la maquinaria Pol II en células madre embrionarias (ESC) (41, 42). Al examinar los datos del conjunto de datos Gene Expression Omnibus (GEO) y la base de datos The Cancer Genome Atlas (TCGA), se identificó WDR43 como un factor oncogénico potencialmente significativo en la patogénesis del CCR y un marcador para predecir la eficacia de la quimioterapia (43), lo que fue consistente con nuestros resultados. Además, las células cancerosas con mayor expresión de WDR43 fueron más resistentes a la muerte celular mediada por quimioterapia y, por lo tanto, la sobreexpresión de WDR43 se relacionó con el mal pronóstico de los pacientes con CCR. Los estudios in vitro han revelado que la eliminación de WDR43 aumentó la apoptosis, inhibió la proliferación, migración e invasión de las células del CCR y ralentizó la carcinogénesis en modelos animales (44).
En el análisis pancancerígeno, estos genes se expresaron significativamente de manera diferente en múltiples cánceres (Figura suplementaria S7), lo que reforzó nuestra creencia en la importancia de este hallazgo. Se ha descubierto el importante papel de GTPBP4, MPHOSPH10, UTP4, WDR3 y WDR43 en tumores humanos, pero aún falta una investigación más profunda sobre el mecanismo. Sin embargo, en el estudio del CCR canino, no se han demostrado las funciones moleculares ni la predictividad diagnóstica clínica de estos marcadores, lo que merecía una mayor exploración.
5 Conclusión
Nuestro estudio fue el primero en explorar este patrón de expresión dinámica del carcinoma de adenoma normal en el CCR canino, explicando las características moleculares del desarrollo de tumores colorrectales en perros. En conclusión, GTPBP4, MPHOSPH10, UTP4, WDR3 y WDR43 podrían servir como biomarcadores valiosos de valores entre especies y proporcionar nuevas selecciones para el futuro diagnóstico y tratamiento del CCR humano y canino.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio están incluidas en el artículo/Material complementario, las consultas adicionales pueden dirigirse a los autores correspondientes.
Declaración ética
No se requirió aprobación ética para el estudio con animales de acuerdo con la legislación local y los requisitos institucionales porque este estudio solo analizó los datos de secuenciación disponibles públicamente y no involucró pruebas con animales.
Contribuciones de los autores
ZL diseñó experimentos y analizó los datos. JZ redactó el manuscrito. QC y XZ llevaron a cabo los experimentos. JL revisó el manuscrito. JL y DL supervisaron el proyecto. ZL y DZ concibieron el proyecto. Todos los autores contribuyeron al artículo y aprobaron la versión enviada.
Financiación
Esta investigación fue financiada por el 14º Programa Nacional de Investigación y Desarrollo Clave del Plan Quinquenal de China (2022YFD1801104).
Reconocimientos
Agradecemos a Yeyizhou-Fu de la Universidad de Pekín por su ayuda desinteresada.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1192525/full#supplementary-material
Notas
Referencias
1. Bazzi, ZA, Sneddon, S, Zhang, PGY, y Tai, IT. Caracterización del panorama de las células inmunitarias en el CCR: implicaciones clínicas de los leucocitos infiltrantes de tumores en el CCR en estadio temprano y tardío. Frente Immunol. (2023) 13:978862. doi: 10.3389/fimmu.2022.978862
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Dekker, E, Tanis, PJ, Vleugels, JLA, Kasi, PM, y Wallace, MB. Cáncer colorrectal. Lanceta. (2019) 394:1467–80. doi: 10.1016/S0140-6736(19)32319-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Vogelstein, B, Fearon, ER, Hamilton, SR, Kern, SE, Preisinger, AC, Leppert, M, et al. Alteraciones genéticas durante el desarrollo del tumor colorrectal. N Engl J Med. (1988) 319:525–32. doi: 10.1056/NEJM198809013190901
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. McEntee, MF, y Brenneman, KA. La desregulación de la betacatenina es común en los tumores colorrectales esporádicos caninos. Veterinario Pathol. (1999) 36:228–36. doi: 10.1354/vp.36-3-228
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Valerius, KD, Powers, BE, McPherron, MA, Hutchison, JM, Mann, FA y Withrow, SJ. Pólipos adenomatosos y carcinoma in situ de colon y recto caninos: 34 casos (1982-1994). J Am Anim Hosp Assoc. (1997) 33:156–60. doi: 10.5326/15473317-33-2-156
6. Youmans, L, Taylor, C, Shin, E, Harrell, A, Ellis, AE, Séguin, B, et al. Alteración frecuente del gen supresor tumoral APC en tumores colorrectales caninos esporádicos. PLoS Uno. (2012) 7:E50813. doi: 10.1371/journal.pone.0050813
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Kuipers, EJ, Grady, WM, Lieberman, D, Seufferlein, T, Sung, JJ, Boelens, PG, et al. Colorectal cancer. Nat Rev Dis Primers. (2015) 1:15065. doi: 10.1038/nrdp.2015.65
8. Aresu, L, Pregel, P, Zanetti, R, Caliari, D, Biolatti, B, y Castagnaro, M. Expresión de E-cadherina y β-catenina en el adenocarcinoma colorrectal canino. Res Vet Sci. (2010) 89:409–14. doi: 10.1016/j.rvsc.2010.04.008
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Cho, SH, Seung, BJ, Kim, SH, Lim, HY y Sur, JH. Sobreexpresión y mutación de los exones 53-4 de p8 en adenocarcinoma intestinal canino. J Comp Pathol. (2020) 175:79–84. doi: 10.1016/j.jcpa.2019.12.008
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Restucci, B, Martano, M, de Vico, G, LoMuzio, L, y Maiolino, P. Expresión de E-cadherina, beta-catenina y proteína APC en tumores colorrectales caninos. Res. Anticancerígeno (2009) 29:2919–25.
11. Tang, J, Li, Y, Lyon, K, Camps, J, Dalton, S, Ried, T, et al. Distinción entre el conductor y el pasajero del cáncer a través de la comparación esporádica del cáncer humano y canino: un estudio de prueba de principio con el cáncer colorrectal. Oncogén. (2014) 33:814–22. doi: 10.1038/onc.2013.17
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Nojadeh, JN, Behrouz Sharif, S, y Sakhinia, E. Inestabilidad de microsatélites en el cáncer colorrectal. EXCLI J. (2018) 17:159–68. doi: 10.17179/excli2017-948
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Pino, MS, y Chung, DC. La vía de inestabilidad cromosómica en el cáncer de colon. Gastroenterología. (2010) 138:2059–72. doi: 10.1053/j.gastro.2009.12.065
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Fearon, ER, y Vogelstein, B. Un modelo genético para la tumorigénesis colorrectal. Células. (1990) 61:759–67. doi: 10.1016/0092-8674(90)90186-I
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Yoshizaki, K, Hirata, A, Matsushita, H, Sakaguchi, M, Yoneji, W, Owaki, K, et al. Estudio epidemiológico molecular de la variante de la línea germinal de APC asociada a poliposis gastrointestinal hereditaria en perros: frecuencia actual en Jack Russell terriers en Japón y distribución de la raza. BMC Vet Res. (2022) 18:230. DOI: 10.1186/s12917-022-03338-W
16. Yoshizaki, K, Hirata, A, Nishii, N, Kawabe, M, Goto, M, Mori, T, et al. Poliposis adenomatosa familiar en perros: poliposis gastrointestinal hereditaria en Jack Russell terriers con mutaciones de la línea germinal en APC. Carcinogénesis. (2021) 42:70–9. doi: 10.1093/carcin/bgaa045
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Wang, J, Wang, T, Sun, Y, Feng, Y, Kisseberth, WC, Henry, CJ, et al. Los tumores colorrectales proliferativos e invasivos en perros domésticos proporcionan información única sobre el cáncer colorrectal humano. Cánceres. (2018a) 10:330. doi: 10.3390/cancers10090330
18. Tang, J, Le, S, Sun, L, Yan, X, Zhang, M, Macleod, J, et al. Anomalías en el número de copias en cánceres colorrectales caninos esporádicos. Genoma Res. (2010) 20:341–50. doi: 10.1101/gr.092726.109
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Guinney, J, Dienstmann, R, Wang, X, de Reyniès, A, Schlicker, A, Soneson, C, et al. Los subtipos moleculares de consenso de cáncer colorrectal. Nat Med. (2015) 21:1350–6. doi: 10.1038/nm.3967
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Leek, JT, Johnson, WE, Parker, HS, Jaffe, AE y Storey, JD. El paquete sva para eliminar efectos de lote y otras variaciones no deseadas en experimentos de alto rendimiento. Bioinformática. (2012) 28:882–3. doi: 10.1093/bioinformatics/bts034
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Kumar, L, y Futschik, ME. Mfuzz: un paquete de software para la agrupación en clústeres de datos de microarrays. Bioinformación. (2007) 2:5–7. doi: 10.6026/97320630002005
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Schmitt, M, y Greten, FR. La patogenia inflamatoria del cáncer colorrectal. Nat Rev Immunol. (2021) 21:653–67. doi: 10.1038/s41577-021-00534-x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Inamura, K . Cánceres colorrectales: una actualización sobre su patología molecular. Cánceres. (2018) 10:26. doi: 10.3390/cancers10010026
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Han, J, Meng, Q, Xi, Q, Zhang, Y, Zhuang, Q, Han, Y, et al. La interleucina-6 estimula la glucólisis aeróbica mediante la regulación de PFKFB3 en la etapa temprana del cáncer colorrectal. Int J Oncol. (2016) 48:215–24. doi: 10.3892/ijo.2015.3225
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Deng, X, Li, D, Ke, X, Wang, Q, Yan, S, Xue, Y, et al. Mir-488 alivia la quimiorresistencia y la glucólisis del cáncer colorrectal al dirigirse a PFKFB3. J Clin Lab Anal. (2021) 35:e23578. doi: 10.1002/jcla.23578
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Smaglik, P . Secuencia genómica, análisis comparativo y estructura haplotípica del perro doméstico. Naturaleza. (2005) 438:1187. DOI: 10.1038/NJ7071-1187A
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Johdi, NA, y Sukor, NF. Inmunoterapia contra el cáncer colorrectal: opciones y estrategias. Frente Immunol. (2020) 11:1624. doi: 10.3389/fimmu.2020.01624
28. Kim, Y-I, Bandyopadhyay, J, Cho, I, Lee, J, Park, DH y Cho, JH. La GTPasa nucleolar NOG-1 regula el desarrollo, el almacenamiento de grasa y la longevidad a través de la señalización de insulina/IGF en C. elegans. Células molares. (2014) 37:51–7. doi: 10.14348/molcells.2014.2251
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Zhou, Q, Yin, Y, Yu, M, Gao, D, Sun, J, Yang, Z, et al. GTPBP4 promueve la progresión y la metástasis del carcinoma hepatocelular a través del metabolismo de la glucosa dependiente de PKM2. Redox Biol. (2022) 56:102458. doi: 10.1016/j.redox.2022.102458
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Yu, H, Jin, S, Zhang, N y Xu, Q. La regulación positiva de GTPBP4 en el carcinoma colorrectal es responsable de la metástasis tumoral. Biochem Biophys Res Commun. (2016) 480:48–54. doi: 10.1016/j.bbrc.2016.10.010
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Siitonen, A, Nalls, MA, Hernández, D, Gibbs, JR, Ding, J, et al. Genética de la enfermedad de Parkinson de inicio temprano en Finlandia: secuenciación del exoma y estudio de asociación de todo el genoma. Envejecimiento neurobiol. (2017) 53:195.e7–195.e10. doi: 10.1016/j.neurobiolaging.2017.01.019
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Park, H, Kim, H, Ha, E, Yoon, S, Kim, M-J, Hong, M, et al. Panax ginseng aumenta los genes relacionados con la respuesta celular regulada a la baja inducida por hipoxia en células de neuroblastoma humano, SK-N-MC. Neurol Res. (2013) 23:38. doi: 10.1179/016164107X172338
33. Pérez-Fernández, J, Román, A, De las Rivas, J, Bustelo, XR, y Dosil, M. El preribosoma 90S es una estructura multimodular que se ensambla a través de un mecanismo jerárquico. Mol Cell Biol. (2007) 27:5414–29. doi: 10.1128/MCB.00380-07
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Freed, EF, y Baserga, SJ. El extremo C-terminal de Utp4, mutado en la cirrosis infantil, es esencial para la biogénesis del ribosoma. Nucl Acids Res. (2010) 38:4798–806. doi: 10.1093/nar/gkq185
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Wilkins, BJ, Lorent, K, Matthews, RP y Pack, M. Defectos biliares mediados por p53 causados por la eliminación de cirh1a, el homólogo de pez cebra del gen responsable de la cirrosis infantil de los indios norteamericanos. PLoS Uno. (2013) 8:e77670. doi: 10.1371/journal.pone.0077670
Resumen de PubMed | Texto completo de CrossRef | Google Académico
36. Schapira, M, Tyers, M, Torrent, M y Arrowsmith, CH. Proteínas de dominio repetido WD40: ¿una nueva clase diana? Nat Rev Droga Discov. (2017) 16:773–86. doi: 10.1038/nrd.2017.179
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Neer, EJ, Schmidt, CJ, Nambudripad, R, y Smith, TF. La antigua familia de proteínas reguladoras de las proteínas de repetición WD. Naturaleza. (1994) 371:297–300. doi: 10.1038/371297a0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Zhao, W, Shen, B, Cheng, Q, Zhou, Y y Chen, K. Funciones de la vía de señalización TSP1-CD47 en la senescencia de las células endoteliales: ciclo celular, inflamación y metabolismo. Mol Biol Rep. (2023) 23:8357. DOI: 10.1007/s11033-023-08357-w
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Su, W, Zhu, S, Chen, K, Yang, H, Tian, M, Fu, Q, et al. La sobreexpresión de WDR3 induce la activación de la vía del hipopótamo al interactuar con GATA4 en el cáncer de páncreas. J Exp Clin Cáncer Res. (2021) 40:88. DOI: 10.1186/s13046-021-01879-w
40. Liu, W, Xie, A, Xiong, J, Li, S, Yang, L y Liu, W. WDR3 promueve propiedades similares a las de las células madre en el cáncer de próstata al inhibir la transcripción de RASSF2A mediada por USF1. J Gene Med. (2023) 2:3598. doi: 10.1002/jgm.3598
Resumen de PubMed | Texto completo de CrossRef | Google Académico
41. Bi, X, Xu, Y, Li, T, Li, X, Li, W, Shao, W, et al. El ARN se dirige al factor de ribogénesis wdr43 a la cromatina para la transcripción y el control de la pluripotencia. Célula molar. (2019) 75:102–116.e9. doi: 10.1016/j.molcel.2019.05.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
42. Zhao, C, Andreeva, V, Gibert, Y, LaBonty, M, Lattanzi, V, Prabhudesai, S, et al. Funciones específicas de los tejidos para el factor de biogénesis del ribosoma Wdr43 en el desarrollo del pez cebra. PLoS Genet. (2014) 10:e1004074. doi: 10.1371/journal.pgen.1004074
Resumen de PubMed | Texto completo de CrossRef | Google Académico
43. Di, Y, Jing, X, Hu, K, Wen, X, Ye, L, Zhang, X, et al. El eje de señalización c-MYC-WDR43 promueve la quimiorresistencia y el crecimiento tumoral en el cáncer colorrectal mediante la inhibición de la actividad de p53. Resistencia a los medicamentos Updat. (2023) 66:100909. doi: 10.1016/j.drup.2022.100909
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: secuencias adenoma-carcinoma, cáncer colorrectal canino, modelo de expresión dinámica, PFKFB3, GTPBP4
Cita: Lin Z, Zhang J, Chen Q, Zhang X, Zhang D, Lin J y Lin D (2023) El análisis del transcriptoma de las secuencias de adenoma y carcinoma identifica nuevos biomarcadores asociados con el desarrollo del cáncer colorrectal canino. Frente. Vet. Sci. 10:1192525. doi: 10.3389/fvets.2023.1192525
Recibido: 23 de marzo de 2023; Aceptado: 11 de octubre de 2023;
Publicado: 29 Noviembre 2023.
Editado por:
Carlos Eduardo Fonseca-Alves, Universidad Paulista, Brasil
Revisado por:
Biswajit Bhowmick, Universidad de Tennessee, Estados
Unidos Alexandre Cavalca, Universidade Estadual Paulista, Brasil
Derechos de autor © 2023 Lin, Zhang, Chen, Zhang, Zhang, Lin y Lin. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Di Zhang, dzhangdvm@cau.edu.cn; Jiahao Lin, jiahaolin@cau.edu.cn; Degui Lin, ldgcau@sina.com
†Estos autores han contribuido igualmente a este trabajo y comparten la primera autoría
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía