Identificación de mutaciones en STAT5A porcino que contribuyen a la transcripción de CISH
Identificación de mutaciones en STAT5A porcino que contribuyen a la transcripción de CISH
 Diwen Yao1
Diwen Yao1  Dongchun Guo2
Dongchun Guo2  Yingkun Zhang1
Yingkun Zhang1  Zhihua Chen1
Zhihua Chen1  Xiaowen Gao1
Xiaowen Gao1  Guiling Xing1
Guiling Xing1  Xiuqin Yang1
Xiuqin Yang1  Xibiao Wang1
Xibiao Wang1  Shengwei Di1
Shengwei Di1  Jiancheng Cai3
Jiancheng Cai3  Buyue Niu1*
Buyue Niu1*- 1Facultad de Ciencia y Tecnología Animal, Universidad Agrícola del Noreste, Harbin, China
- 2Laboratorio Estatal Clave de Biotecnología Veterinaria, Instituto de Investigación Veterinaria de Harbin, Academia China de Ciencias Agrícolas (CAAS), Harbin, China
- 3Granja de cría de Lanxi, Lanxi, China
La identificación de genes causales o variantes genéticas asociadas a rasgos fenotípicos beneficia la mejora genética de los animales. El CISH desempeña un papel en la inmunidad y el crecimiento, sin embargo, los factores transcripcionales ascendentes del CISH porcino y las variaciones genéticas de estos factores siguen sin estar claros. En este estudio, en primer lugar, identificamos el promotor central mínimo de CISH porcino y confirmamos la existencia de sitios de unión a STATx. La sobreexpresión y la RT-qPCR demostraron que STAT5A aumentó la actividad transcripcional de CISH (P < 0,01) y la expresión de ARNm (P < 0,01), mientras que GATA1 inhibió la actividad transcripcional de CISH (P < 0,01) y la posterior expresión de ARNm (P < 0,05 o P < 0,01). A continuación, se examinaron las variaciones genéticas funcionales putativas de STAT5A porcino y se estableció una PCR-SSCP para los genotipos g.508A>C y g.566C>T. El análisis genético poblacional mostró que la frecuencia del alelo A de g.508A>C y la frecuencia del alelo C de g.566C>T fue de 0,61 y 0,94 en cerdos Min, respectivamente, mientras que estos dos alelos se fijaron en la población Landrace. El análisis estadístico mostró que los lechones Min con genotipo CC en g.566C>T o Hap1: AC tenían mayor peso corporal a los 28 días, peso corporal a los 35 días y GMD que los animales TC o Hap3: CT (P < 0,05, P < 0,05 ). Un ensayo de actividad de luciferasa adicional demostró que la actividad de g.508A>C en el alelo C era menor que la del alelo A (P < 0,05). En conjunto, el presente estudio demostró que STAT5A reguló positivamente la transcripción de CISH porcino, y el SNP g.566C>T en el STAT5A se asoció con el rasgo de crecimiento del lechón Min.
1. Introducción
La mejora de la salud y el peso de los lechones, especialmente antes del destete, beneficia la producción porcina. La cría selectiva, que cambia las tendencias genéticas de las poblaciones ganaderas, se ha convertido en un método eficaz para mejorar la salud y el rendimiento del crecimiento porcino. La identificación de genes candidatos o variantes genéticas causantes asociadas con el fenotipo animal está involucrada principalmente en la cría basada en la genética.
La proteína SH2 inducible por citocinas (CISH) es un miembro de las proteínas supresoras de señalización de citocinas (SOCS) que funcionan en el desarrollo de la sangre y las células inmunitarias (1), el crecimiento celular y el metabolismo (2-5). Los estudios iniciales de ratones knockout de CISH demostraron sus funciones en la regulación de la inmunidad y la enfermedad (6). A partir de entonces, se demostró que las variaciones genéticas dentro de CISH se asocian con múltiples enfermedades en humanos, incluida la malaria (7), la bacteriemia (7), la tuberculosis (7, 8), la hepatitis B persistente (9), el síndrome de disfunción orgánica múltiple (MODS) (10) y la sepsis (11). Recientemente, se ha descrito la regulación de CISH sobre la adiposidad del ratón, la ingesta de alimentos y el metabolismo de la glucosa (12). Mientras tanto, el CISH de pollo se ha identificado como un gen clave involucrado en la deposición excesiva de grasa inducida por la mutación del receptor de la hormona del crecimiento (GHR), lo que redujo la calidad de la canal y la inmunidad de los pollos de engorde a las enfermedades (13). Aunque se han identificado variaciones genéticas en el CISH porcino asociadas con diarrea o rasgos de crecimiento en lechones (14, 15), aún se desconocen las características del promotor del CISH porcino y sus factores transcripcionales ascendentes.
La integración de los datos de RNA-seq y ChIP-seq en líneas celulares eritroides murinas sugiere la existencia de un transductor de señal y activador de la transcripción 5 (STAT5) y de la proteína de unión a GATA 1 (GATA1) en el promotor de CISH (16). La función de GATA1 en la inmunidad intestinal o el crecimiento individual es limitada, mientras que STAT5 es fundamental para mediar la señalización impulsada por citocinas y ejerce efectos generalizados sobre la inmunidad, la enfermedad y el crecimiento (17-19). Por ejemplo, la infección por el virus de la diarrea epidémica porcina (PEDV) regula al alza la expresión de STAT3 y STAT5A en las células intestinales porcinas (IPEC-J2) (20), y las variantes genéticas en la región STAT3/STAT5A/STAT5B se asocian con la enfermedad inflamatoria intestinal (EII), una enfermedad gastrointestinal compleja (21-23). Aunque se ha informado que las variaciones genéticas en STAT5A se asocian con la producción de leche bovina (24-26), el rendimiento reproductivo bovino (24, 27, 28), el rendimiento de la leche de cabra y oveja (29, 30) y las variaciones genéticas de STAT5A porcino aún no se conocen bien.
En general, el polimorfismo de un solo nucleótido no sinónimo (nsSNP), que conduce a la sustitución de un solo aminoácido en la proteína codificada, es de particular interés en la investigación genética de mamíferos. Por ejemplo, nsSNP rs342747498 (T/G) dentro de la 17β-hidroxiesteroide deshidrogenasa porcina tipo 14 (HSD17B14) provoca la sustitución de fenilalanina (Phe) a valina (Val), lo que conduce a la alteración de HSD17B14 función en la conversión de estrógenos primerizos y apoptosis de células granulares de ovario (31). Mientras tanto, utilizando el enfoque in silico integrado, se han identificado nsSNPs asociados con rasgos económicos animales que incluyen la resistencia a la mastitis bovina (32), el contenido de grasa abdominal de pollo (33) y el fenotipo gris clásico de alpacas (34). Además del nsSNP, el SNP en la región no codificante del cuerpo del gen es igualmente importante, lo que afecta al fenotipo al alterar la expresión génica funcional. Por ejemplo, rs55618224 (T>C) en la 3’UTR del ciclo de división celular 42 (CDC42) causa un polimorfismo del sitio de unión miR-18a, que afecta la expresión de CDC42 en placentas porcinas (35). g.32828A>G en el intrón del retinoblastoma 1 (RB1) altera la expresión génica, lo que a su vez influye en la deposición de grasa abdominal de pollo (36).
Dado el papel vital de CISH en la enfermedad y el crecimiento, el propósito del presente estudio fue (1) identificar los factores transcripcionales de la CISH porcina y (2) explorar las mutaciones funcionales en el factor de transcripción identificado. En este trabajo, caracterizamos el promotor central de la CISH porcina, confirmamos que STAT5A es un factor transactivador de la CISH porcina, examinamos las variaciones genéticas en STAT5A y prospectamos sus funciones, en un intento de proporcionar nuevos marcadores para el mejoramiento molecular.
2. Materiales y métodos
2.1. Los animales y la declaración ética
Los animales utilizados para la identificación de la variación genética fueron proporcionados por la granja de cría de Lanxi (Lanxi, Heilongjiang, China). Se utilizaron un total de 226 ADN genómico de cerdos de raza autóctona de 226 Min y 186 para el genotipado y análisis de asociación de SNPs, y los rasgos fenotípicos de todos estos animales se han descrito previamente (14). En resumen, se observaron las heces de los lechones y se les asignó una puntuación diaria de diarrea de acuerdo con el siguiente estándar: heces sólidas normales 0, diarrea leve con heces blandas y sueltas 1, diarrea moderada con heces semilíquidas 2 y diarrea severa con heces líquidas y no formadas 3 (37). La puntuación de diarrea de los lechones se calculó como la suma de la puntuación diaria de diarrea durante el experimento. Además, se registró el peso corporal al nacer, a los 21 días, a los 28 días y a los 35 días y se calculó la ganancia media diaria (GMD) de la siguiente manera, GMD = (peso a los 35 días – peso al nacer)/35.
La experimentación con animales se llevó a cabo de acuerdo con las directrices para el cuidado y uso de animales aprobadas por el Comité de Manejo de Animales de Laboratorio de la Universidad Agrícola del Noreste (Harbin, Heilongjiang, China).
2.2. Análisis in silico
Los promotores centrales del gen CISH porcino se analizaron utilizando el software de predicción de promotores de redes neuronales en línea (BDGP) (http://www.fruitfly.org/seqtools/promoter.html). Se utilizaron el software JASPAR (http://jaspar.binf.ku.dk/cgi-bin/jaspar_db.pl) y PROMO (http://www.urogene.org/cgi-bin/methprimer/methprimer.cgi) para buscar los sitios putativos de unión al factor de transcripción. Los SNPs funcionales en la secuencia codificante (CDS) de STAT5A porcino se predijeron mediante el método in silico. La información sobre los nsSNPs en el gen STAT5A porcino se recopiló por primera vez de la base de datos de SNP de Ensembl (http://www.ensembl.org/). Cinco herramientas bioinformáticas que incluyen PhD-SNP (https://snps.biofold.org/phd-snp/), SIFT (http://sift.jcvi.org/), SNAP (http://www.rostlab.org/services/SNAP), Meta-SNP (http://snps.biofold.org/meta-snp/) y PolyPhen-2.0 (http://genetics.bwh.harvard.edu/pph2/uses), que fueron descritas con más detalle por Emadi et al. (38) y Li et al. (33), se emplearon para predecir los NSSNPs más nocivos en función de la incorporación de las puntuaciones de todos los servidores. Los SNPs en la 3’UTR de STAT5A porcino se analizaron mediante miRBase (http://www.mirbase.org) para cribar los SNPs funcionales putativos que podrían alterar los elementos de respuesta de microARN (miARN). Del mismo modo, los SNPs en la UTR 5′ y el intrón 1 fueron analizados por JASPAR y PROMO con una puntuación umbral de 80.
2.3. Construcciones de la luciferasa reportera y mutagénesis dirigida al sitio
Para obtener el promotor de CISH porcino, se recuperó de Ensembl la secuencia genómica ascendente de 2,5 kb de CISH porcino y se diseñó el par de cebadores CISH-C utilizando el software Primer 5 (Tabla Suplementaria S1). La reacción de PCR contenía 100 ng de plantillas de ADN, 0,5 μM de cada cebador como en la Tabla Suplementaria S1, y 10 μL 2 × Taq Master Mix (TaKaRa, Dalian, China). Las condiciones de PCR fueron de 4 min a 94 °C; Se purificaron, secuenciaron y ligaron 35 ciclos de 30 s a 94 °C, 30 s a temperatura de recocido (Tabla suplementaria S1), 2 min a 72 °C y un paso final a 72 °C durante 8 min. Los productos de PCR se purificaron, secuenciaron y ligaron en PMD18-T (TaKaRa, Dalian, China) para producir PMD18-T-CISH. A continuación, utilizando PMD18-T-CISH como plantilla, se amplificaron una serie de fragmentos de deleción 5′ del promotor de CISH utilizando los cebadores directos CISH-C (C1-C5), CISH-P (P1-P3) y el cebador inverso CISH-R (Tabla suplementaria S1). Para explorar el efecto de GATA1 en la transcripción de CISH, se diseñó el cebador CISH-G y se produjo un fragmento específico que contenía un elemento de unión putativo a GATA1 mediante PCR (Tabla suplementaria S1). Todos estos productos de PCR se purificaron, se digierieron con Xho I y Kpn I o Hind III (TaKaRa, Dalian, China) y se insertaron en el vector pGL3-Basic (Promega, Madison, Wisconsin, EE. UU.) para producir plásmidos reporteros de luciferasa pGL3-CISH-C (C1-C5), pGL3-CISH-P (P1-P3) y pGL3-CISH-G.
Utilizando el plásmido pGL3-CISH-P1 como plantilla y los cebadores mutagénicos CISH-Mut1 y CISH-Mut2 (Tabla suplementaria S1), se generaron tres mutantes de los sitios de unión a STATx predichos mediante PCR. A continuación, estos productos de PCR se purificaron, se digierieron mediante endonucleasas de restricción y se insertaron entre los sitios Xho I y Kpn I del vector pGL3-básico. Todos los plásmidos reconstruidos se confirmaron mediante secuenciación de ADN y se denominaron pGL3-CISH-mut1, pGL3-CISH-mut2 y pGL3-CISH-mut3.
Para construir pGL3-STAT5A-Luc y pGL3-STAT5A-SNP1, se amplificaron fragmentos específicos que contenían diferentes variaciones en el intrón 1 de STAT5A porcino con el par de cebadores STAT5A-Luc (Tabla suplementaria S1) y el ADN genómico genotipado, insertado en el vector pGL3-básico. A continuación, utilizando el plásmido pGL3-STAT5A-Luc como plantilla y los cebadores mutagénicos STAT5A-Mut (Tabla Suplementaria S1), se produjo el tercer fragmento y se construyó el correspondiente pGL3-STAT5A-SNP2 como se ha descrito anteriormente. Todos los plásmidos positivos fueron confirmados por digestión endonucleasa y secuenciación de ADN.
2.4. Cultivo celular, transfección y ensayo de informe de luciferasa
Hela e IPEC-J2 fueron seleccionados como sistemas modelo in vitro para explorar los promotores centrales de CISH porcinos. Las células se cultivaron en placas de 24 pocillos con DMEM suplementado con FBS al 10% (Gibco, Carlsbad, CA, EE.UU.), mantenido a 37 °C en la atmósfera de CO al 5%2 durante 18–24 h. A continuación, las células se transfectaron con 0,5 μg de plásmido promotor específico de luciferasa, 0,005 μg de pRL-TK utilizado como control interno (Promega, Madison, Wisconsin, EE.UU.) y 1,5 μL de reactivo de transfección de ADN X-treme GENE HP (Roche, EE.UU.). Simultáneamente, el plásmido pGL3-Basic mezclado con pRL-TK se transfectó a las células correspondientes como el control negativo del ensayo de informe de luciferasa. Después de 24-48 h, todas las células se recolectaron y lisaron con el tampón de ensayo de luciferasa (Promega, Madison, Wisconsin, EE. UU.), y se examinó la actividad enzimática de luciérnaga y renilla de acuerdo con el Sistema de ensayo de reportero de doble luciferasa (Promega, Madison, Wisconsin, EE. UU.). La transfección se realizó por triplicado y se repitió dos o tres veces, y la actividad luciferasa se calculó como la relación entre luciérnaga y renilla.
2.5. Sobreexpresión de STAT3, STAT5A y GATA1 porcinos
La región codificante completa de STAT3 (NM_001044580.1), STAT5A (NM_214290.1) y GATA1 (NM_001278767.1) porcinos se amplificaron utilizando ADNc porcino y pares de cebadores (Tabla suplementaria S1) respectivamente. Los productos de PCR purificados se insertaron entre los sitios Xho I y EcoR I o Not I del vector pCMV-HA (Promega, Madison, WI, EE. UU.) para construir pCMV-HA-STAT3, pCMV-HA-STAT5A y pCMV-HA-GATA1, respectivamente. Estos plásmidos de expresión se confirmaron mediante secuenciación de ADN y análisis de Western blot. A continuación, las células IPEC-J2 se transfectaron con 0,25 μg de plásmido indicador de luciferasa promotor de CISH (pGL3-CISH-C4, pGL3-CISH-P1 o pGL3-CISH-G), 0,25 μg de plásmido de expresión correspondiente (pCMV-HA-STAT3, pCMV-HA-STAT5A, pCMV-HA-GATA1 o pCMV-HA) y 0,005 μg de pRL-TK. La actividad relativa de la luciferasa se calculó como se describió anteriormente.
To validate the role of transcriptional factors on porcine CISH mRNA expression, IPEC-J2 cells were transfected with 2 μg of pCMV-HA-STAT3, pCMV-HA-STAT5A, pCMV-HA-GATA1, or pCMV-HA, respectively. All the cells were collected, and RT-qPCR was performed.
2.6. Western blot, RNA extraction, and RT-qPCR
Para el Western blot, las células IPEC-J2 transfectadas con pCMV-HA-STAT3, pCMV-HA-STAT5A, pCMV-HA-GATA1 o pCMV-HA se lisaron con tampón RIPA (SEVEN, Pekín, China), se hirvieron con tampón de carga desnaturalizante de 5 ×, se resolvieron en SDS-PAGE al 12% y se transfirieron a la membrana de fluoruro de polivinilideno (PVDF) Immuno-Blot (Millipore, Billerica, MA, EE. UU.). A continuación, la membrana se bloqueó con BSA al 5%, se lavó con TBST y se incubó con anticuerpo HA-tag (ABclonal, Wuhan, China) en dilución 1:1000 durante 12 h a 4 °C, y un anticuerpo secundario a temperatura ambiente durante 2 h. Por último, esta membrana incubada se lavó con TBST y se utilizó el kit Super ECL (SEVEN, Beijing, China) para mostrar las bandas.
Para la extracción de ARN y RT-qPCR, se extrajeron ARN totales de células IPEC-J2 utilizando TRIzol (Takara, Dalian, China) y se transcribieron inversamente en ADNc utilizando PrimerScript RT Master Mix (Takara, Dalian, China). De acuerdo con SYBR Premix Ex Taq (Takara, Dalian, China), la RT-qPCR se realizó en un sistema ABI 7500 (Applied Biosystems, Foster City, CA, EE. UU.) con la siguiente reacción: 100 ng ADNc, 0,2 μM de cada cebador (Tabla suplementaria S1) y 10 μL de mezcla SYBR (Takara, Dalian, China). Las condiciones de qPCR fueron de 95 °C durante 30 s, 40 ciclos con 95 °C durante 5 s y 60 °C durante 35 s, con las curvas de fusión construidas al mismo tiempo. De acuerdo con el 2−ΔΔCT (35), se seleccionó la gliceraldehído-3-fosfato deshidrogenasa (GAPDH) (36, 37) como gen de mantenimiento para normalizar la expresión de los genes diana y se calculó la expresión relativa del ARNm de un gen específico en las células.
2.7. Identificación y genotipado de variaciones en STAT5A porcino
Los valores 5’UTR y 3’UTR de STAT5A porcino se amplificaron utilizando el par de cebadores STAT5A-5’UTR o STAT5A-3’UTR (Tabla Suplementaria S1) y los grupos mixtos de ADN genómico que consistieron en cinco cerdos Min sanos, cinco Min diarreicos, cinco Landrace sanos y cinco Landrace diarreicos descritos anteriormente. Todos los productos de PCR fueron purificados, secuenciados comercialmente (Sangon, Shanghái, China) y alineados con Clustal Omega. Los SNPs funcionales predichos mediante herramientas in silico fueron genotipados en poblaciones Min y Landrace a través de polimorfismo de conformación monocatenario basado en PCR (PCR-SSCP). En resumen, los fragmentos que contenían SNPs se amplificaron mediante PCR utilizando el par de cebadores STAT5A-SNP (Tabla suplementaria S1). A continuación, se desnaturalizaron 1 μL de productos de PCR mezclados con 9 μL de tampón de desnaturalización durante 10 min a 98 °C, se colocaron en agua helada durante 5 min, se separaron en un gel PAGE al 14% y se resolvieron mediante tinción de plata. Los productos de PCR con diversos genotipos se purificaron y secuenciaron comercialmente (Sangon, Shanghái, China).
2.8. Análisis estadístico
La diferencia significativa se evaluó mediante la prueba t en el software GraphPad Prism 5 (GraphPad, La Jolla, CA, USA) o ANOVA de una vía. La heterocigosidad observada (Ho), la heterocigosidad esperada (He), el número de alelos efectivos (Ne) y la prueba de chi-cuadrado para el equilibrio de Hardy-Weinberg de los polimorfismos de variación genética se calcularon utilizando el software Popgene (versión 1.32). Se utilizó el software Haploview 4.2 para analizar el desequilibrio de ligamiento (LD) entre g.508A>C y g.566C>T y se construyó el haplotipo.
Se utilizó un procedimiento de modelo lineal mixto de SAS versión 8.0 para realizar el análisis de asociación entre SNP o haplotipo y el rasgo fenotipo en cerdos Min.
Donde YIj son los rasgos fenotípicos observados, μ es la media poblacional, GYo es el efecto fijo de la genética, Sj es el efecto aleatorio de la cerda, y eIj es el residuo aleatorio.
3. Resultados
3.1. Identificación del promotor central de la CISH porcina
Un fragmento de 2071 pb que contenía una región flanqueante de 5′ y una secuencia codificante de 106 pb fue producido por PCR, y cuatro promotores de núcleo putativos en la región de −1.807/−1.758, −1.358/−1.309, −460/−411 y −201/−152 fueron predichos por el software en línea BDGP (Figura 1A). Posteriormente, se amplificaron cinco deleciones 5′ y se insertaron en el plásmido pGL3 para producir los plásmidos del gen indicador de luciferasa promotor de CISH. Como se muestra en la Figura 1B, todos estos plásmidos tuvieron actividad luciferasa tanto en células IPEC-J2 como en células Hela. Un análisis posterior mostró que el fragmento CISH-C4 (secuencia de −412 a 106 pb) tenía mayor actividad que el fragmento CISH-C5 (secuencia de −258 a 106 pb), lo que sugiere la existencia de importantes elementos de transcripción reguladores positivos en −412/−258 pb. Sin embargo, la actividad de CISH-C2 porcino (secuencia de −1.498 a 106 pb) fue menor que la de CISH-C3 (de −528 a 106 pb), lo que indica que había elementos en la región de −1.498/−528 que podrían regular negativamente la transcripción de CISH porcina.
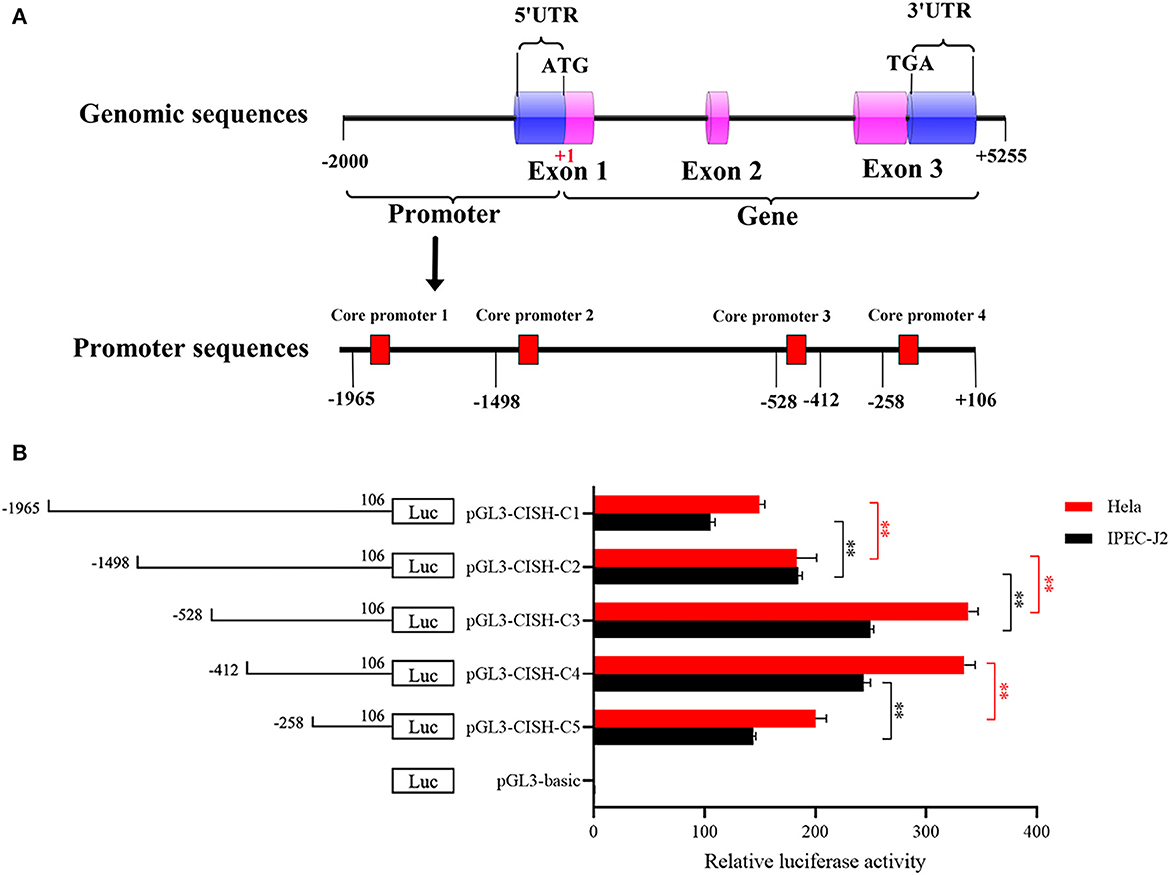 Figura 1. 5′-Análisis de deleción del promotor CISH. (A) Diagrama esquemático de las regiones promotoras centrales predichas en el promotor CISH. (B) Actividades promotoras de una serie de constructos eliminados determinados por el ensayo de luciferasa. Panel izquierdo, representación esquemática de las deleciones 5′ unidas con el gen de la luciferasa en el vector pGL3. Los nucleótidos se numeran a partir del sitio de inicio de la traducción potencial (ATG) al que se le asignó +1. En el panel derecho, las actividades relativas de cinco constructos deledos de 5′ al pGL3-básico determinado por ensayos de luciferasa. La actividad relativa de la luciferasa se representó mediante la normalización a pRL-TK y luego se normalizó frente a la actividad de pGL3-basic en células IPEC-J2 o Hela. Los valores se muestran como medias ± SE de tres réplicas. Se analizaron las diferencias estadísticas en las actividades relativas en las mismas celdas. **P < 0,01.
Figura 1. 5′-Análisis de deleción del promotor CISH. (A) Diagrama esquemático de las regiones promotoras centrales predichas en el promotor CISH. (B) Actividades promotoras de una serie de constructos eliminados determinados por el ensayo de luciferasa. Panel izquierdo, representación esquemática de las deleciones 5′ unidas con el gen de la luciferasa en el vector pGL3. Los nucleótidos se numeran a partir del sitio de inicio de la traducción potencial (ATG) al que se le asignó +1. En el panel derecho, las actividades relativas de cinco constructos deledos de 5′ al pGL3-básico determinado por ensayos de luciferasa. La actividad relativa de la luciferasa se representó mediante la normalización a pRL-TK y luego se normalizó frente a la actividad de pGL3-basic en células IPEC-J2 o Hela. Los valores se muestran como medias ± SE de tres réplicas. Se analizaron las diferencias estadísticas en las actividades relativas en las mismas celdas. **P < 0,01.
3.2. Identificación de los elementos reguladores del CISH porcino
El análisis in silico mostró que el fragmento CISH-C4 contenía un promotor central y sitios de unión al factor de transcripción STATx tetrámero (Figura 2A). Para verificar esta predicción, se construyeron tres plásmidos de deleción 5′. El análisis de la actividad de la luciferasa mostró que la deleción de los dos primeros sitios putativos de unión a STATx (−412/−338) no alteró la actividad de la luciferasa del fragmento CISH-C4 (Figura 2B). Sin embargo, la deleción de los sitios de unión tercero y cuarto de STATx (−338/−227) redujo significativamente la actividad luciferasa de CISH-C4 (Figura 2B). Además, como se muestra en la Figura 2B, la desaparición de la actividad luciferasa del fragmento CISH-P3 indicó la existencia de un promotor central mínimo en la región de −227/−106. Para verificar la existencia de sitios de unión a STATx, se mutaron los terceros o cuartos sitios de unión de STATx predichos dentro del fragmento CISH-P1 (Figura 2C). El análisis de la actividad de la luciferasa reveló que las mutaciones redujeron significativamente la actividad de la luciferasa de CISH-P1 (Figura 2D).
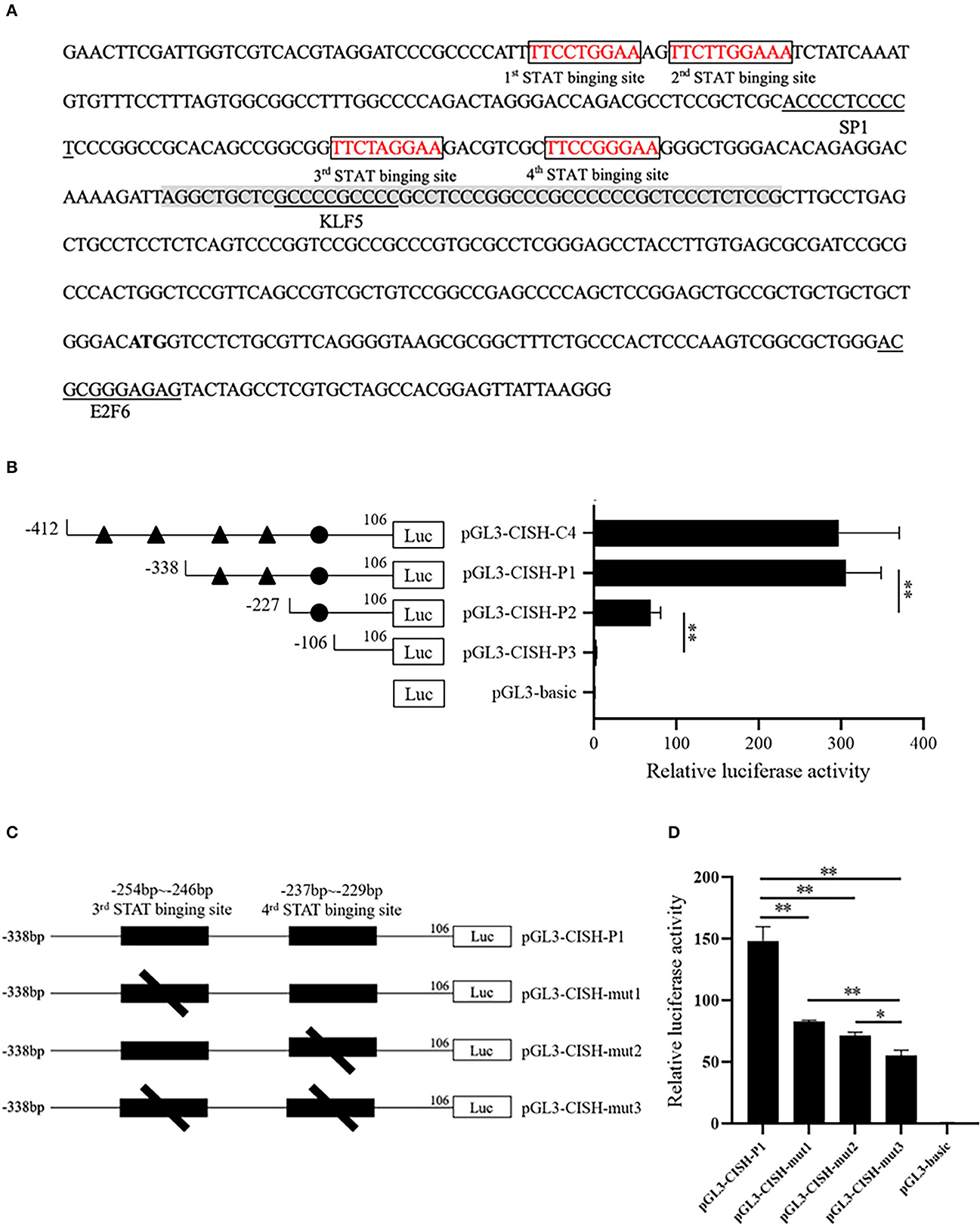 Figura 2. Sitios de unión de STATx en el promotor de CISH. (A) Los factores transcripcionales predichos y el promotor central en el fragmento CISH-C4. (B) 5′-Análisis de deleción del fragmento CISH-C4. (C) Presentación esquemática de dos sitios putativos de unión a STAT y mutantes de deleción para cada uno de los sitios. (D) Mutagénesis dirigida al sitio en los sitios de unión de STATx mediante ensayo de luciferasa. Los resultados se expresan como proporciones de actividades relativas que se representaron mediante la normalización a pRL-TK y luego se normalizaron contra la actividad de pGL3-basic en células IPEC-J2. Los valores se muestran como medias ± SE de tres repeticiones y las diferencias estadísticas se analizaron en las mismas celdas. *P < 0,05, **P < 0,01.
Figura 2. Sitios de unión de STATx en el promotor de CISH. (A) Los factores transcripcionales predichos y el promotor central en el fragmento CISH-C4. (B) 5′-Análisis de deleción del fragmento CISH-C4. (C) Presentación esquemática de dos sitios putativos de unión a STAT y mutantes de deleción para cada uno de los sitios. (D) Mutagénesis dirigida al sitio en los sitios de unión de STATx mediante ensayo de luciferasa. Los resultados se expresan como proporciones de actividades relativas que se representaron mediante la normalización a pRL-TK y luego se normalizaron contra la actividad de pGL3-basic en células IPEC-J2. Los valores se muestran como medias ± SE de tres repeticiones y las diferencias estadísticas se analizaron en las mismas celdas. *P < 0,05, **P < 0,01.
3.3. Efecto de STAT3 y STAT5A sobre la actividad transcripcional de CISH porcino y la expresión de ARNm
En este estudio, basado en la literatura, se seleccionaron STAT3 y STAT5A porcinos para verificar su efecto sobre la actividad transcripcional de CISH y la expresión de ARNm. El plásmido de expresión pCMV-HA-STAT3 o pCMV-HA-STAT5A, junto con los plásmidos reporteros de luciferasa promotores de CISH pGL3-CISH-C4 o pGL3-CISH-P1, se transfectaron a IPEC-J2. El análisis de la actividad de la luciferasa mostró que la sobreexpresión de STAT3 podría aumentar significativamente la actividad transcripcional de CISH (P < 0,01) (Figuras 3A-C). Del mismo modo, la sobreexpresión de STAT5A reguló al alza la actividad transcripcional de CISH (P < 0,01) (Figuras 3D-F). La RT-qPCR reveló que la sobreexpresión de STAT3 no afectó a la expresión del ARNm de CISH (Figuras 3G, H). Sin embargo, STAT5A promovió la expresión de CISH a las 48 h después de la transfección (P < 0,01) (Figuras 3I, J).
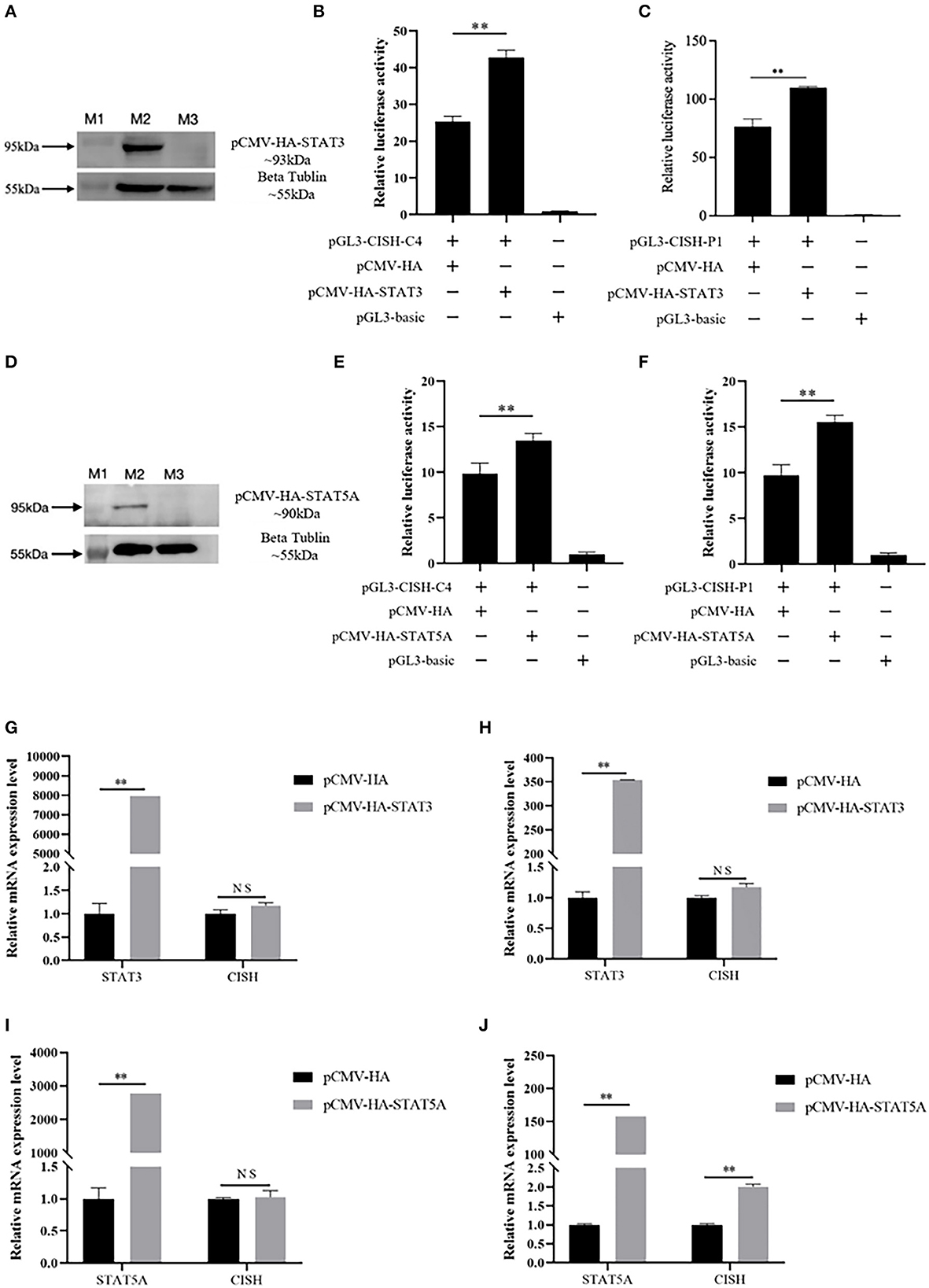 Figura 3. Efecto de STAT3 y STAT5A porcinos sobre la actividad transcripcional de CISH y la expresión de ARNm. (A) Sobreexpresión de STAT3 porcino en IPEC-J2. Lane M1, marcador proteico. Lane M2, la proteína expresada de pCMV-HA-STAT3; Carril M3, control de pCMV-HA. (B) La sobreexpresión de STAT3 regula al alza la actividad de CISH-C4 en IPEC-J2. (C) La sobreexpresión de STAT3 regula al alza la actividad de CISH-P1 en IPEC-J2. (D) Sobreexpresión de STAT5A porcino en IPEC-J2. Lane M1, marcador proteico. Lane M2, la proteína expresada de pCMV-HA-STAT5A; Carril M3, control de pCMV-HA. (E) La sobreexpresión de STAT5A regula al alza la actividad de CISH-C4 en IPEC-J2. (F) La sobreexpresión de STAT5A regula al alza la actividad de CISH-P1 en IPEC-J2. (G, H) La sobreexpresión de STAT3 no tiene ningún efecto sobre la expresión del ARNm de CISH en IPEC-J2. (I) La sobreexpresión de STAT5A no tiene ningún efecto sobre la expresión de ARNm de CISH después de 24 h en IPEC-J2. (J) La sobreexpresión de STAT5A aumentó la expresión de ARNm de CISH después de 48 h en IPEC-J2. Los valores se muestran como la media ± DE (n = 3). **P < 0,01.
Figura 3. Efecto de STAT3 y STAT5A porcinos sobre la actividad transcripcional de CISH y la expresión de ARNm. (A) Sobreexpresión de STAT3 porcino en IPEC-J2. Lane M1, marcador proteico. Lane M2, la proteína expresada de pCMV-HA-STAT3; Carril M3, control de pCMV-HA. (B) La sobreexpresión de STAT3 regula al alza la actividad de CISH-C4 en IPEC-J2. (C) La sobreexpresión de STAT3 regula al alza la actividad de CISH-P1 en IPEC-J2. (D) Sobreexpresión de STAT5A porcino en IPEC-J2. Lane M1, marcador proteico. Lane M2, la proteína expresada de pCMV-HA-STAT5A; Carril M3, control de pCMV-HA. (E) La sobreexpresión de STAT5A regula al alza la actividad de CISH-C4 en IPEC-J2. (F) La sobreexpresión de STAT5A regula al alza la actividad de CISH-P1 en IPEC-J2. (G, H) La sobreexpresión de STAT3 no tiene ningún efecto sobre la expresión del ARNm de CISH en IPEC-J2. (I) La sobreexpresión de STAT5A no tiene ningún efecto sobre la expresión de ARNm de CISH después de 24 h en IPEC-J2. (J) La sobreexpresión de STAT5A aumentó la expresión de ARNm de CISH después de 48 h en IPEC-J2. Los valores se muestran como la media ± DE (n = 3). **P < 0,01.
3.4. Efecto de GATA1 sobre la actividad transcripcional del CISH porcino y la expresión del ARNm
El análisis in silico mostró que el fragmento CISH-G contenía sitios de unión a GATA1 aguas arriba de los sitios de unión del factor de transcripción STATx del tetrámero (Figura 4A). El plásmido de reconstrucción pCMV-HA-GATA1 y pGL3-CISH-G se transfectaron a IPEC-J2. El análisis de la actividad de la luciferasa mostró que la sobreexpresión de GATA1 reprimió significativamente la actividad transcripcional de CISH (P < 0,01) (Figuras 4B, C). La RT-qPCR reveló que la sobreexpresión de GATA1 redujo la expresión de ARNm de CISH a las 24 y 48 h después de la transfección (Figuras 4D, E).
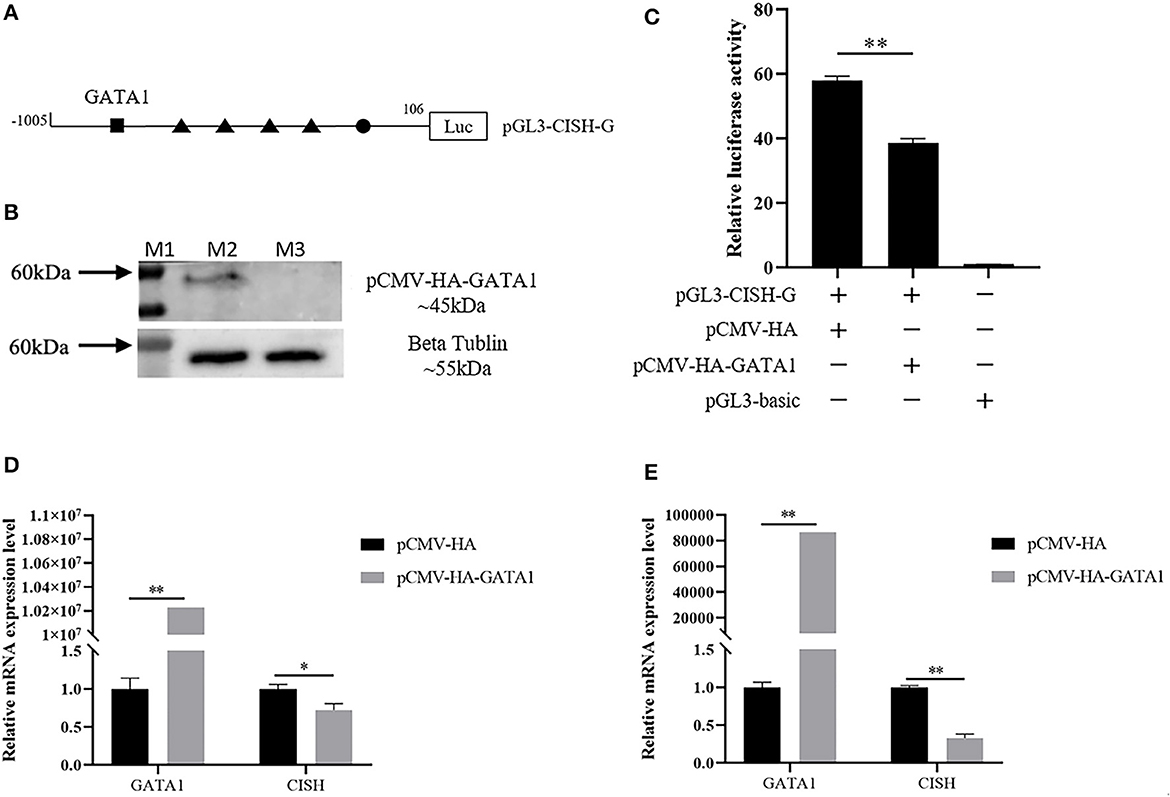 Figura 4. Efecto de GATA1 porcino sobre la actividad transcripcional de CISH y la expresión de ARNm. (A) Sitios de unión a GATA1 en el promotor CISH. (B) Sobreexpresión de GATA1 porcino en IPEC-J2. (C) La sobreexpresión de GATA1 regula a la baja la actividad de CISH-G en IPEC-J2. (D) La sobreexpresión de GATA1 regula a la baja la expresión de ARNm de CISH en IPEC-J2 después de la transfección durante 24 h. (E) La sobreexpresión de GATA1 regula a la baja la expresión de ARNm de CISH en IPEC-J2 después de la transfección durante 48 h. Los valores se muestran como la media ± DE (n = 3). *P < 0,05, **P < 0,01.
Figura 4. Efecto de GATA1 porcino sobre la actividad transcripcional de CISH y la expresión de ARNm. (A) Sitios de unión a GATA1 en el promotor CISH. (B) Sobreexpresión de GATA1 porcino en IPEC-J2. (C) La sobreexpresión de GATA1 regula a la baja la actividad de CISH-G en IPEC-J2. (D) La sobreexpresión de GATA1 regula a la baja la expresión de ARNm de CISH en IPEC-J2 después de la transfección durante 24 h. (E) La sobreexpresión de GATA1 regula a la baja la expresión de ARNm de CISH en IPEC-J2 después de la transfección durante 48 h. Los valores se muestran como la media ± DE (n = 3). *P < 0,05, **P < 0,01.
3.5. Identificación de los SNP de función putativa en STAT5A porcino
En este estudio, se recuperaron un total de 49 SNPs en la región codificante STAT5A porcina de la base de datos Ensembl. Entre los 14 nsSNPs, solo c.870G>T fue el nsSNP deletéreo predicho con Polyphen-2 (Tabla Suplementaria S2). A continuación, se amplificó un fragmento de 1618 pb que contenía un intrón17 de 210 pb, un exón18 de 1384 pb (que contenía 3’UTR de 1221 pb) y una región aguas abajo de 24 pb de STAT5A porcino. Aunque se identificaron 9 SNPs en este fragmento, no se predijo que ninguno de estos SNPs alterara los sitios de unión a los miRNAs (datos no dados). De manera similar, se amplificó un fragmento de 909 pb que contenía 5’UTR de 231 pb y 678 pb de STAT5A porcino y se identificaron tres SNP (g.373C>G, g.508A>C y g.566C>T) en el intrón 1. Según JASPAR, g.373C>G causó un polimorfismo de proteína 454 (ZNF454) de dedos de zinc; se predijo que g.508A>C alteraría el sitio de unión del factor de transcripción E2F 4 (E2F4) o del factor de transcripción Dp-1 (TFDP1), donde E2F4 o TFDP1 se unirían al alelo C pero no al alelo A; g.566C>T alteró los sitios de unión del dominio PR que contiene el protooncogén 4 (PRDM4) o RELB, y la subunidad NF-kB (RELB) (Tabla suplementaria S3). Sin embargo, entre estos SNPs, PROMO reveló que solo g.566C>T alteró la unión del factor de transcripción ETS ELK1 (Elk-1) (Tabla suplementaria S2). En conjunto, el análisis in silico indicó que g.508A>C o g.566C>T eran los SNP funcionales putativos.
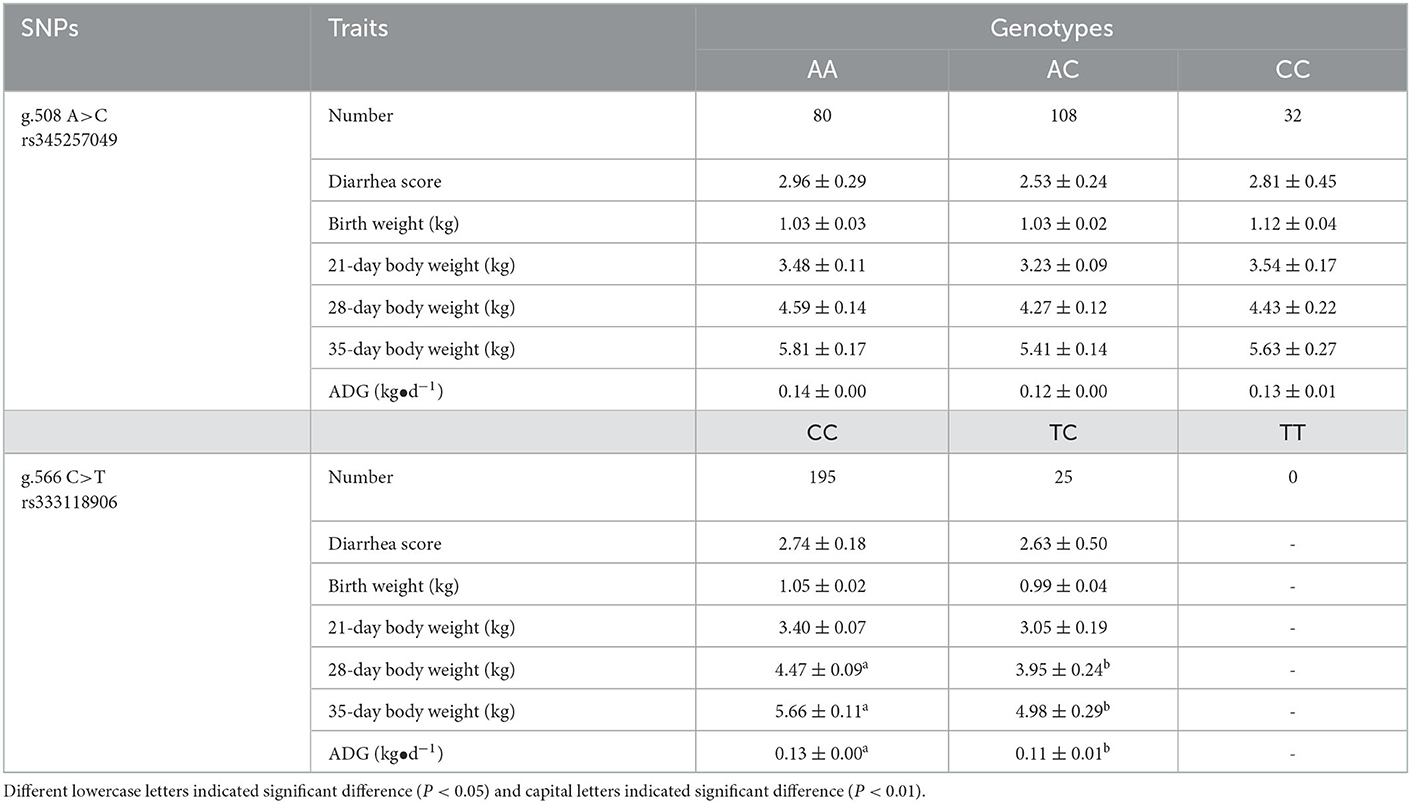
3.6. Genotipado y análisis de asociación en población Min y Landrace
g.508A>C y g.566C>T fueron genotipificados por PCR-SSCP en poblaciones Min y Landrace. Como se muestra en la Figura 4, se observaron tres genotipos (AA, AC y CC) de g.508A>C (Figuras 5A), mientras que solo se encontraron genotipos CC y CT de g.566C>T (Figuras 5A, D, E). La frecuencia del alelo A (g.508A>C) fue de 0,61 en los cerdos Min, y la del alelo C (g.566C>T) fue de 0,94 en esta población. Sin embargo, estos dos alelos se fijaron en la población Landrace. En los cerdos Min, el Ho y el He de g.508A>C fueron de 0,49 y 0,48, mientras que el Ho y el He de g.566C>T fueron de 0,11 y 0,11, respectivamente; el número alélico efectivo (Ne) de g.508A>C y g.566C>T fue de 1,91 y 1,12, respectivamente; y los valores de contenido de información de polimorfismo (PIC) fueron de 0,36 y 0,10, respectivamente (Tabla 1). El análisis estadístico mostró que g.508A>C no se asoció con los rasgos fenotípicos de los lechones Min, mientras que los animales con genotipo CC en g.566C>T tuvieron más peso corporal a los 28 días, peso corporal a los 35 días y GMD que los del genotipo CT (P < 0,05) (Tabla 2). Además, se construyeron tres haplotipos y Hap1:AC fue el haplotipo mayor con una frecuencia de 0,61, mientras que el menor Hap3:CT fue de 0,06 (Tabla 3). El análisis de asociación indicó que los lechones Min con Hap1:AC tenían un mayor peso corporal a los 28 días, un peso corporal a los 35 días y una GMD superiores en comparación con Hap3:CT (P < 0,05) (Tabla 3).
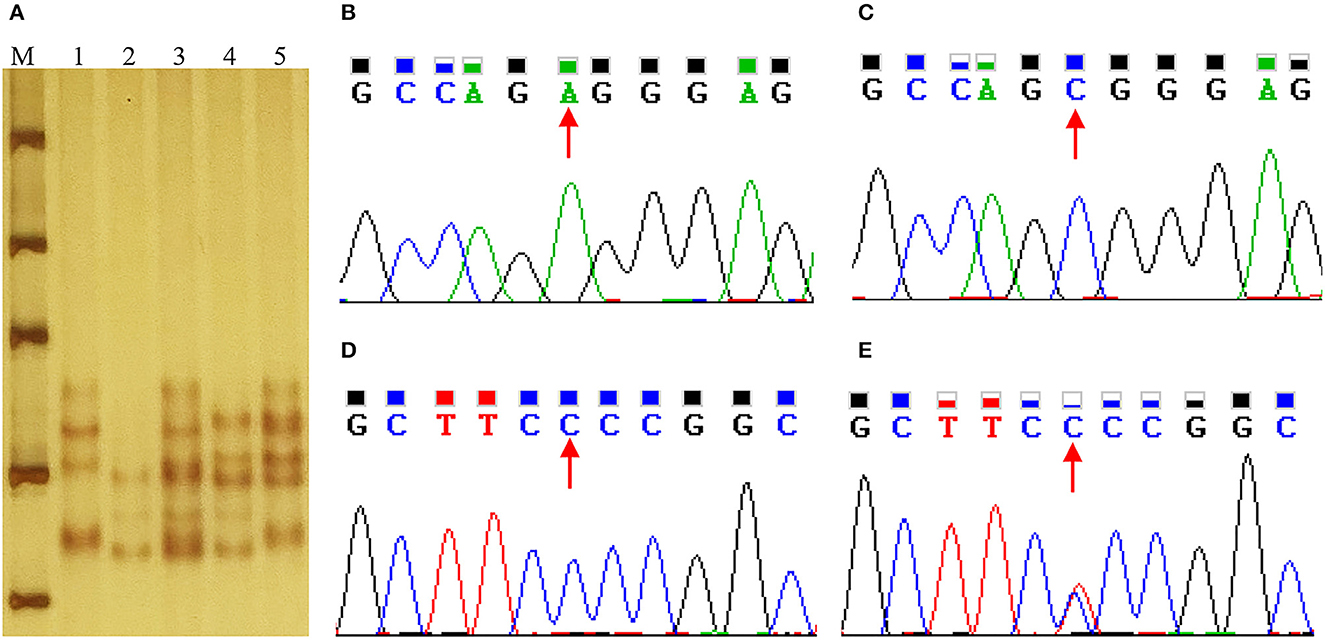 Figura 5. Secuencia y genotipado de g.508A>C y g.566C>T en la región promotora de STAT5A porcina. (A) Genotipado de g.508A>C y g.566C>T en el intrón 1 de STAT5A porcino. marcador molecular Lane M DL1000; Los carriles 1 ~ 5 muestran el genotipo de AACC, CCCC, ACCC, CCTC y ACTC. (B, C) Secuencia de g.508A>C en el intrón 1 de STAT5A porcino. (D, E) Secuencia de g.566C>T en el intrón 1 de STAT5A porcino.
Figura 5. Secuencia y genotipado de g.508A>C y g.566C>T en la región promotora de STAT5A porcina. (A) Genotipado de g.508A>C y g.566C>T en el intrón 1 de STAT5A porcino. marcador molecular Lane M DL1000; Los carriles 1 ~ 5 muestran el genotipo de AACC, CCCC, ACCC, CCTC y ACTC. (B, C) Secuencia de g.508A>C en el intrón 1 de STAT5A porcino. (D, E) Secuencia de g.566C>T en el intrón 1 de STAT5A porcino.
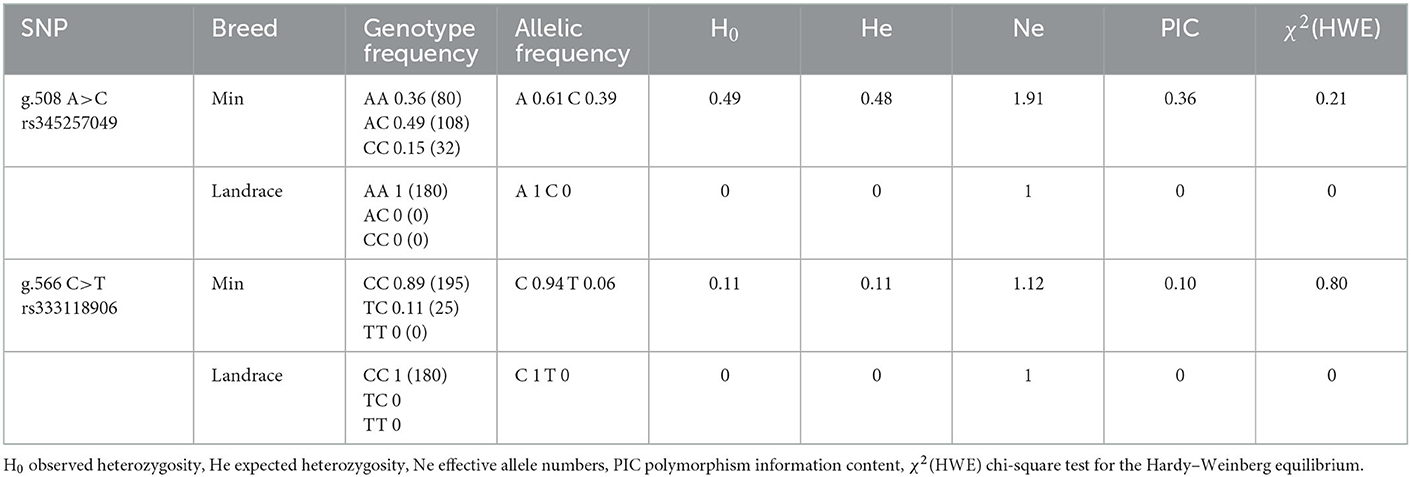 Tabla 1. Genotipo y frecuencia alélica de g.508A>C y g.566C>T en poblaciones de cerdos Min y Landrace.
Tabla 1. Genotipo y frecuencia alélica de g.508A>C y g.566C>T en poblaciones de cerdos Min y Landrace.
3.7. Efecto de los SNPs sobre la actividad transcripcional de STAT5A porcino
Como se muestra en la Figura 6A, se construyeron el indicador de luciferasa promotora de STAT5A pGL3-STAT5A-Luc (g.508A y g.566C), pGL3-STAT5A-SNP1 (g.508C y g. 566C) y pGL3-STAT5A-SNP2 (g.508A y g.566T) para verificar la función putativa de g.508A>C o g.566C>T. Después de la transfección, se calculó y comparó la actividad luciferasa de estos vectores reporteros. El análisis estadístico indicó que la actividad luciferasa del alelo g.508A>C A fue mayor que la del alelo C (P < 0.05), mientras que la diferencia entre el alelo C y T de g.566C>T no fue significativa (Figura 6B).
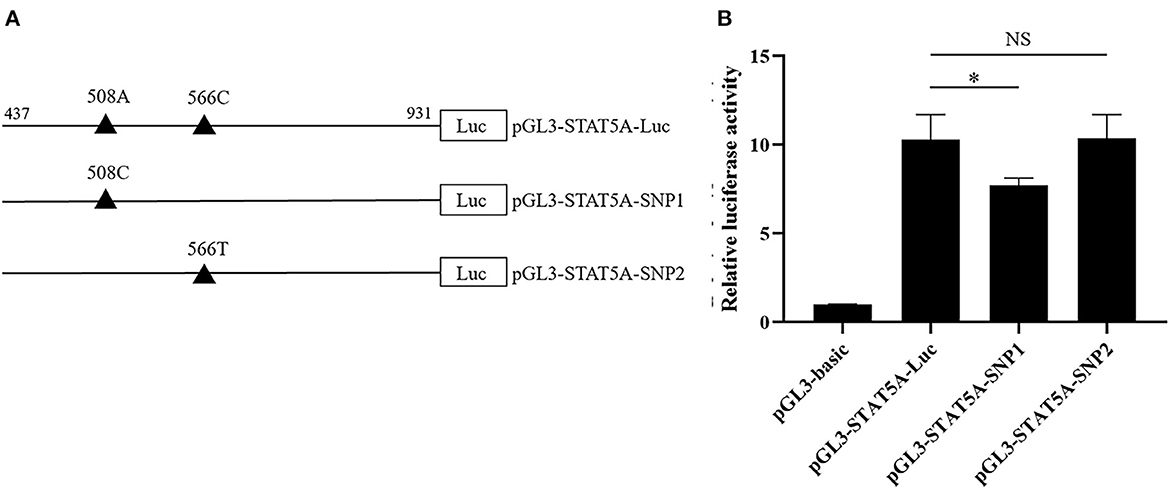 Figura 6. Los efectos de g.508A>C o g.566C>T sobre la actividad transcripcional porcina STAT5A. (A) Presentación esquemática del constructo reportero de luciferasa. Los SNPs fueron nombrados y numerados a partir del primer nucleótido del primer exón de STAT5A porcino (ENSSSCT00000064828.3), que se asumió como el sitio inicial transcripcional putativo y se asignó como +1. (B) Ensayos de luciferasa de diferentes SNPs que el reportero de luciferasa construye en células IPEC-J2. Los valores se muestran como la media ± DE (n = 3). *P < 0,05.
Figura 6. Los efectos de g.508A>C o g.566C>T sobre la actividad transcripcional porcina STAT5A. (A) Presentación esquemática del constructo reportero de luciferasa. Los SNPs fueron nombrados y numerados a partir del primer nucleótido del primer exón de STAT5A porcino (ENSSSCT00000064828.3), que se asumió como el sitio inicial transcripcional putativo y se asignó como +1. (B) Ensayos de luciferasa de diferentes SNPs que el reportero de luciferasa construye en células IPEC-J2. Los valores se muestran como la media ± DE (n = 3). *P < 0,05.
4. Discusión
El CISH porcino se expresó ampliamente en múltiples tejidos, incluido el intestino delgado de lechones y cerdos adultos (15, 39), lo que indica que el CISH funcionaba en la fisiología intestinal del cerdo. En este estudio, Hela e IPEC-J2 fueron seleccionados como modelos in vitro para identificar el promotor central de CISH. Hela es un modelo celular general ampliamente utilizado en la investigación biológica. IPEC-J2, una línea celular de epitelio yeyuno aislada de un lechón neonatal, se ha utilizado para estudiar la salud intestinal porcina (40). Curiosamente, la mayor actividad luciferasa de la región CISH −412/−258 se presentó en estas dos líneas celulares, lo que sugiere la existencia de elementos de transcripción reguladores positivos. Los siguientes análisis bioinformáticos, análisis de deleción 5′ y mutagénesis dirigida al sitio verificaron la existencia de sitios de unión a STATx en el promotor CISH porcino, lo cual fue consistente con el estudio en el promotor CISH de ratón donde se identificaron dos conjuntos de sitios de unión STAT5 en tándem conservados (41, 42).
Entre los siete números de la familia STAT, STAT3 y STAT5, relacionados con las respuestas inmunitarias de la mucosa intestinal, se incluyen en los loci candidatos relacionados con la susceptibilidad a la EII (21-23). STAT5 se compone de STAT5A y STAT5B, que se organizan a lo largo de los cromosomas de la cabeza a la cabeza. A pesar de que STAT5A y STAT5B mostraron similitudes de secuencia de más del 90%, se dirigieron a fenotipos o funciones distintivas (43, 44). Un estudio reciente sobre la respuesta del huésped a la infección por PEDV en IPEC-J2 reveló la alta expresión de la familia STAT, excepto STAT5B y STAT6 después de la infección (20). Teniendo en cuenta el papel de CISH en la inmunidad y la enfermedad, se seleccionaron STAT3 y STAT5A para estimar sus efectos sobre la transcripción y expresión de CISH porcino en este estudio. Encontramos que estos dos factores regulaban positivamente la transcripción de CISH en IPEC-J2, mientras que el aumento de la expresión de ARNm de CISH se encontró solo en células sobreexpresadas de STAT5A. Curiosamente, la sobreexpresión de GATA1 reguló a la baja la transcripción y la expresión de CISH en IPEC-J2 a las 24 h después de la transfección, mientras que STAT5A reguló al alza CISH a las 48 h en lugar de a las 24 h. Los estudios existentes demostraron que STAT5 estaba involucrado en la transducción de señales de múltiples factores, y la fosforilación de STAT5 resultó en la dimerización, la translocación al núcleo y la unión del ADN a los sitios de unión al objetivo en promotores y potenciadores (45, 46). Por lo tanto, la posible explicación es que STAT5A necesita ser fosforilado antes de regular rápidamente los genes diana posteriores.
Para mejorar la precisión del método in silico, se recomendó utilizar al menos cuatro o cinco herramientas para obtener medidas consistentes de los efectos de los nsSNPs en la estructura y función de la proteína diana (38). Sin embargo, utilizando esta estrategia, se identificó el nsSNP más deletéreo dentro de STAT5A. Los nsSNPs estudiados en esta investigación fueron recuperados de la base de datos pública con información limitada. Se sugiere secuenciar la región codificante de STAT5A utilizando ADN genómico con diferentes antecedentes genéticos para proporcionar más información sobre los SNP.
Se ha identificado un SNP funcional en el promotor STAT3 porcino asociado con la diarrea de los lechones (47), mientras que ninguno de los SNP genotipados en STAT5A afectó la diarrea de los lechones. Teniendo en cuenta la contribución de STAT3 a la replicación de PEDV (48), el presente estudio indica que STAT3 podría ser el gen candidato para la diarrea en lechones. Para el rasgo de rendimiento, los cerdos Min con genotipo CC en g.566C>T tuvieron mayor peso corporal al destete y GMD. Curiosamente, el alelo C se fijó en Landrace, y la frecuencia del alelo C fue de 0,94 en los cerdos Min. El Min es una raza local china con una tasa de crecimiento más baja en comparación con la Landrace, una raza general seleccionada por sus rasgos de rendimiento (http://afs.okstate.edu/breeds/swine/minzhu/index.html/). El hallazgo de este estudio es consistente con las características de estas dos razas, y se sugiere eliminar el alelo T para avanzar en la mejora de los rasgos de crecimiento del cerdo Min.
Las herramientas in silico revelaron que tanto g.508A>C como g.566C>T causan polimorfismo transcripcional, sin embargo, el ensayo posterior del reportero de luciferasa verificó que solo g.508A>C, que se predijo que alteraría los sitios de unión del factor transcripcional E2F4 o TFDP1, mostró una actividad transcripcional diversa. De acuerdo con JASPAR, el alelo C de g.508A>C poseía un sitio de unión E2F4 o TFDP1, y se encontró la menor actividad luciferasa del alelo C. Es necesario verificar si E2F4 o TFDP1 contribuyen a la transcripción de STAT5A a través del polimorfismo g.508A>C explorando la interacción entre estas proteínas y el promotor de STAT5A. Para el g.566C>T, aunque este SNP se asoció con el fenotipo, la actividad luciferasa consistente de diversos alelos indicó que este SNP podría ser un marcador relacionado. En el futuro, se sugieren más exploraciones en el promotor de STAT5A para comprender el mecanismo de regulación transcripcional de STAT5A y proporcionar nuevos marcadores moleculares causales para la selección de mejoramiento.
5. Conclusión
STAT5A regula positivamente la transcripción y expresión de CISH porcino en la célula IPEC-J2. STAT5A podría seleccionarse como un gen candidato para el rasgo de crecimiento de lechones Min. La mutación intrónica g.566C>T se asoció con el rasgo de crecimiento de los lechones Min.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio están incluidas en el artículo/Material complementario, las consultas posteriores pueden dirigirse al autor correspondiente.
Declaración ética
El estudio con animales fue revisado y aprobado por el Comité de Manejo de Animales de Laboratorio de la Universidad Agrícola del Noreste.
Contribuciones de los autores
BN y DG diseñaron y supervisaron el experimento. DY, ZC, YZ, XG y GX llevaron a cabo el experimento. DY, YZ y ZC analizaron los datos. XW, SD y JC proporcionaron los animales. BN, DG, DY y XY contribuyeron a la redacción del manuscrito. Todos los autores contribuyeron al artículo y aprobaron la versión enviada.
Financiación
Este estudio contó con el apoyo de la Fundación de Ciencias Naturales de la Provincia de Heilongjiang (LH2020C015) y la Fundación para Profesores Clave de la Universidad de Agricultura del Noreste (19XG11).
Conflicto de intereses
JC es empleado de Lanxi Breeding Farm.
El resto de los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un potencial conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2022.1090833/full#supplementary-material
Referencias
1. Trengove MC, Ward AC. Proteínas SOCS en desarrollo y enfermedad. Am J Exp Clin Immunol. (2013) 2:1–29.
2. Metcalf D, Greenhalgh CJ, Viney E, Willson TA, Starr R, Nicola NA, et al. Gigantismo en ratones carentes de supresor de la señalización de citoquinas-2. Naturaleza. (2000) 405:1069–73. doi: 10.1038/35016611
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Mori H, Hanada R, Hanada T, Aki D, Mashima R, Nishinakamura H, et al. La deficiencia de Socs3 en el cerebro eleva la sensibilidad a la leptina y confiere resistencia a la obesidad inducida por la dieta. Nat Med. (2004) 10:739–43. DOI: 10.1038/NM1071
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Krebs DL, Uren RT, Metcalf D, Rakar S, Zhang JG, Starr R, et al. SOCS-6 se une al sustrato 4 del receptor de insulina, y los ratones que carecen del gen SOCS-6 exhiben un retraso leve del crecimiento. Mol Cell Biol. (2002) 22:4567–78. doi: 10.1128/MCB.22.13.4567-4578.2002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Krebs DL, Metcalf D, Merson TD, Voss AK, Thomas T, Zhang JG, et al. Desarrollo de hidrocefalia en ratones carentes de SOCS7. Proc Natl Acad Sci U S A. (2004) 101:15446–51. doi: 10.1073/pnas.0406870101
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Yang XO, Zhang H, Kim BS, Niu X, Peng J, Chen Y, et al. El supresor de señalización CIS controla el desarrollo de células T proalérgicas y la inflamación alérgica de las vías respiratorias. Nat Immunol. (2013) 14:732–40. doi: 10.1038/ni.2633
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Khor CC, Vannberg FO, Chapman SJ, Guo H, Wong SH, Walley AJ, et al. CISH y susceptibilidad a enfermedades infecciosas. N Engl J Med. (2010) 362:2092–101. doi: 10.1056/NEJMoa0905606
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Sun L, Jin YQ, Shen C, Qi H, Chu P, Yin QQ, et al. Contribución genética de los polimorfismos promotores de CISH a la susceptibilidad a la tuberculosis en niños chinos. PLoS UNO. (2014) 9:e92020. doi: 10.1371/journal.pone.0092020
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Hu Z, Yang J, Wu Y, Xiong G, Wang Y, Yang J, et al. Los polimorfismos en el gen CISH se asocian con la infección persistente por el virus de la hepatitis B en la población china Han. PLoS UNO. (2014) 9:E100826. doi: 10.1371/journal.pone.0100826
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Zhang A, Gu W, Lu H, Zeng L, Zhang L, Du D, et al. Contribución genética de los polimorfismos supresores de señalización de citoquinas a la susceptibilidad a la infección después de una lesión traumática. Clin Exp Immunol. (2018) 194:93–102. doi: 10.1111/cei.13160
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Rosier F, Brisebarre A, Dupuis C, Baaklini S, Puthier D, Brun C, et al. Predisposición genética a la mortalidad en pacientes con shock séptico: desde el GWAS hasta la identificación de una variante reguladora que modula la actividad de un potenciador de CISH. Int J Mol Sci. (2021) 22:5852. doi: 10.3390/ijms22115852
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Naser W, Maymand S, Rivera LR, Connor T, Liongue C, Smith CM, et al. La proteína que contiene el dominio SH2 inducible por citocinas contribuye a la regulación de la adiposidad, la ingesta de alimentos y el metabolismo de la glucosa. FASEB J. (2022) 36:e22320. doi: 10.1096/fj.202101882R
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Chen G, Chen J, Wu J, Ren X, Li L, Lu S, et al. Los análisis integrativos del perfil de expresión del ARNm revelan que SOCS2 y CISH desempeñan un papel importante en la deposición excesiva de grasa abdominal inducida por la mutación ghr en el pollo enano ligado al sexo. Frente Genet. (2021) 11:610605. doi: 10.3389/fgene.2020.610605
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Niu B, Guo D, Liu Z, Han X, Wang X. Caracterización del gen de la proteína SH2 inducible por citoquinas porcinas y su asociación con los rasgos de diarrea de lechones. Asian-Australas J Anim Sci. (2017) 30:1689–95. doi: 10.5713/ajas.16.0169
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Niu B, Chen Z, Yao D, Kou M, Gao X, Sun Y, et al. Un indel de 12 pb en el gen 3’UTR del CISH porcino asociado con la puntuación de diarrea de lechones autóctonos. Res Vet Sci. (2022) 146:53–9. doi: 10.1016/j.rvsc.2022.03.012
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Gillinder KR, Tuckey H, Bell CC, Magor GW, Huang S, Ilsley MD, et al. Dianas directas de la señalización de pSTAT5 en la eritropoyesis. PLoS UNO. (2017) 12:e0180922. doi: 10.1371/journal.pone.0180922
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Smit LS, Vanderkuur JA, Stimage A, Han Y, Luo G, Yu-Lee LY, et al. Fosforilación de tirosilo inducida por la hormona del crecimiento y actividad de unión al ácido desoxirribonucleico de Stat5A y Stat5B. Endocrinología. (1997) 138:3426–34. doi: 10.1210/endo.138.8.5332
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. O’Shea JJ, Schwartz DM, Villarino AV, Gadina M, McInnes IB, Laurence A. La vía JAK-STAT: impacto en la enfermedad humana e intervención terapéutica. Annu Rev Med. (2015) 66:311–28. doi: 10.1146/annurev-med-051113-024537
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Villarino AV, Kanno Y, Ferdinand JR, O’Shea JJ. Mecanismos de señalización de Jak/STAT en la inmunidad y la enfermedad. J Immunol. (2015) 194:21–7. doi: 10.4049/jimmunol.1401867
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Hu Z, Li Y, Du H, Ren J, Zheng X, Wei K, et al. El análisis del transcriptoma revela la modulación de la familia STAT en células IPEC-J2 infectadas por PEDV. BMC Genómica. (2020) 21:891. doi: 10.1186/s12864-020-07306-2
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Jostins L, Ripke S, Weersma RK, Duerr RH, McGovern DP, Hui KY, et al. Las interacciones huésped-microbio han dado forma a la arquitectura genética de la enfermedad inflamatoria intestinal. Naturaleza. (2012) 491:119–24. doi: 10.1038/nature11582
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Kasembeli MM, Bharadwaj U, Robinson P, Tweardy DJ. Contribución de STAT3 a las enfermedades inflamatorias y fibróticas y perspectivas de su diana terapéutica. Int J Mol Sci. (2018) 19:2299. doi: 10.3390/ijms19082299
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Hedl M, Sun R, Huang C, Abraham C. Los umbrales de señalización STAT3 y STAT5 determinan una regulación distinta para las citocinas inflamatorias inducidas por receptores innatos, y las variantes de la enfermedad STAT3/STAT5 modulan estos resultados. J Immunol. (2019) 203:3325–38. doi: 10.4049/jimmunol.1900031
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Khatib H, Monson RL, Schutzkus V, Kohl DM, Rosa GJ, Rutledge JJ. Las mutaciones en el gen STAT5A se asocian con la supervivencia embrionaria y la composición de la leche en el ganado. J Lácteos Sci. (2008) 91:784–93. doi: 10.3168/jds.2007-0669
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Schennink A, Bovenhuis H, Léon-Kloosterziel Léon-Kloosterziel KM, van Arendonk JA, Visker MH. Efecto de los polimorfismos en los genes FASN, OLR1, PPARGC1A, PRL y STAT5A sobre la composición de la grasa láctea bovina. Anim Genet. (2009) 40:909–16. doi: 10.1111/j.1365-2052.2009.01940.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Raven LA, Cocks BG, Kemper KE, Chamberlain AJ, Vander Jagt CJ, Goddard ME, et al. La imputación dirigida de variantes de secuencia y perfiles de expresión génica identifica doce genes candidatos asociados con el volumen de lactancia, la composición y el intervalo entre partos en el ganado lechero. Genoma de Mamm. (2016) 27:81–97. doi: 10.1007/s00335-015-9613-8
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Hax LT, Schneider A, Jacometo CB, Mattei P, da Silva TC, Farina G, et al. Asociación entre polimorfismos en genes del eje somatotrópico y fertilidad de vacas lecheras Holstein. Teriogenología. (2017) 88:67–72. doi: 10.1016/j.theriogenology.2016.03.044
Resumen de PubMed | Texto completo de CrossRef | Google Académico
28. Silva Silveira PA, Butler WR, da Silva TC, Barros CC, Corrêa MN, Schneider A. Asociación de polimorfismos en los genes IGF-I, GHR y STAT5A con la concentración sérica de IGF-I y el rendimiento reproductivo de vacas lecheras Holstein. Anim Reprod Sci. (2019) 211:106206. doi: 10.1016/j.anireprosci.2019.106206
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. An XP, Hou JX, Zhao HB, Bai L, Peng JY, Zhu CM, et al. Identificación de polimorfismos en los genes DGAT1 y STAT5A de cabra y asociación con rasgos de producción de leche. Checo J Anim Sci. (2013) 58:321–7. doi: 10.17221/6862-CJAS
30. Abousoliman I, Reyer H, Oster M, Muráni E, Mourad M, Rashed MA, et al. Análisis de genes candidatos para rasgos de crecimiento y rendimiento de la leche en la oveja barki egipcia. Animales (Basilea). (2020) 10:197. doi: 10.3390/ani10020197
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Gao S, Tao R, Tong X, Xu Q, Zhao J, Guo Y, et al. Identificación de polimorfismos funcionales de un solo nucleótido en el gen de la HSD17B14 porcina asociado con la diferencia en el comportamiento del celo entre primerizas blancas grandes y primerizas. Biomoléculas. (2020) 10:1545. doi: 10.3390/biom10111545
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Jacob KK, Radhika G, Aravindakshan TV. Una evaluación in silico de polimorfismos de un solo nucleótido no sinónimos de genes de resistencia a la mastitis en bovinos. Anim Biotechnol. (2020) 31:25–31. doi: 10.1080/10495398.2018.1524770
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Li YD, Wang WJ Li ZW, Wang N, Xiao F, Gao HH, et al. La integración de métodos de asociación y computacionales revela variantes funcionales del gen LEPR para el contenido de grasa abdominal en pollos. J Integr Agr. (2021) 20:2734–48. doi: 10.1016/S2095-3119(20)63575-4
34. Jones M, Sargento C, Richardson M, Groth D, Brooks S, Munyard K, et al. El SNP no sinónimo en el exón 3 del gen KIT es responsable del fenotipo gris clásico en las alpacas (Vicugna pacos). Anim Genet. (2019) 50:493–500. doi: 10.1111/age.12814
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Liu R, Deng D, Liu X, Xiao Y, Huang J, Wang F, et al. Un polimorfismo del sitio de unión de miR-18a en CDC42 3’UTR afecta la expresión del ARNm de CDC42 en placentas y se asocia con el tamaño de la camada en cerdos. Genoma de Mamm. (2019) 30:34–41. doi: 10.1007/s00335-018-9788-x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
36. Cheng B, Zhang H, Liu C, Chen X, Chen Y, Sun Y, et al. La variante intrónica funcional en el gen del retinoblastoma 1 subyace a la adiposidad de los pollos de engorde al alterar los sitios de unión del factor nuclear kB y de la proteína de la caja HMG 2 relacionada con SRY. J Agric Food Chem. (2019) 67:9727–37. doi: 10.1021/acs.jafc.9b01719
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Kelly D, O’Brien JJ, McCracken KJ. Efecto de la alimentación progresiva sobre la incidencia, duración y gravedad de la diarrea post-destete en cerdos. Res Vet Sci. (1990) 49:223–8. doi: 10.1016/S0034-5288(18)31082-8
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Emadi E, Akhoundi F, Kalantar SM, Emadi-Baygi M. Predicción de los NSSNPs con cambio de sentido más deletéreos de las isoformas proteicas del gen HLA-G humano y evaluación in silico de sus consecuencias estructurales y funcionales. BMC Genet. (2020) 21:94. doi: 10.1186/s12863-020-00890-y
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Delgado-Ortega M, Melo S, Meurens F. Expresión de ARNm SOCS1-7 y CIS en tejidos porcinos. Veterinario Inmunol Inmunopatíaco. (2011) 144:493–8. doi: 10.1016/j.vetimm.2011.08.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Geens MM, Niewold TA. Optimización de las condiciones de cultivo de una línea celular epitelial porcina IPEC-J2 a través de una caracterización histológica y fisiológica. Citotecnología. (2011) 63:415–23. doi: 10.1007/s10616-011-9362-9
Resumen de PubMed | Texto completo de CrossRef | Google Académico
41. Matsumoto A, Masuhara M, Mitsui K, Yokouchi M, Ohtsubo M, Misawa H, et al. CIS, una proteína SH2 inducible por citoquinas, es una diana de la vía JAK-STAT5 y modula la activación de STAT5. Sangre. (1997) 89:3148–54. doi: 10.1182/sangre. V89.9.3148
Resumen de PubMed | Texto completo de CrossRef | Google Académico
42. Moriggl R, Sexl V, Kenner L, Duntsch C, Stangl K, Gingras S, et al. La formación de tetrámeros Stat5 se asocia con la leucemogénesis. Célula cancerosa. (2005) 7:87–99. doi: 10.1016/j.ccr.2004.12.010
Resumen de PubMed | Texto completo de CrossRef | Google Académico
43. Grimley PM, Dong F, Rui H. Stat5a y Stat5b: gemelos fraternos de transducción de señales y activación transcripcional. Factor de crecimiento de citoquinas Rev. (1999) 10:131–57. doi: 10.1016/S1359-6101(99)00011-8
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Hao P, Waxman DJ. Regulación STAT5 de la metilación de CpG hepática dependiente del sexo en el mapeo de elementos reguladores distales a genes sesgados por sexo. Mol Cell Biol. (2021) 41:E00166–20. doi: 10.1128/MCB.00166-20
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Ehret GB, Reichenbach P, Schindler U, Horvath CM, Fritz S, Nabholz M, et al. Especificidad de unión al ADN de diferentes proteínas STAT. Comparación de la especificidad in vitro con sitios diana naturales. J Biol Chem. (2001) 276:6675–88. doi: 10.1074/jbc. M001748200
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Sathyanarayana BK Li P, Lin JX, Leonard WJ, Lee B. Los modelos moleculares de tetrámeros STAT5A complejados con ADN predicen frecuencias relativas de todo el genoma del espaciamiento entre los dos motivos de unión de dímeros de los sitios de unión de tetrámeros. PLoS UNO. (2016) 11:e0160339. doi: 10.1371/journal.pone.0160339
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. Chen Z, Yao D, Guo D, Sun Y, Liu L, Kou M, et al. Mutación funcional asociada con la diarrea de lechones parcialmente mediante la regulación de la transcripción de STAT3 porcino. Front Vet Sci. (2022) 9:1034187. doi: 10.3389/fvets.2022.1034187
Resumen de PubMed | Texto completo de CrossRef | Google Académico
48. Yang L, Xu J, Guo L, Guo T, Zhang L, Feng L, et al. La activación del receptor del factor de crecimiento epidérmico inducida por el virus de la diarrea epidérmica porcina afecta la actividad antiviral del interferón tipo I. J Virol. (2018) 92:e02095–17. doi: 10.1128/JVI.02095-17
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: porcino, CISH, STAT5A, polimorfismo, rasgo de crecimiento de lechones
Cita: Yao D, Guo D, Zhang Y, Chen Z, Gao X, Xing G, Yang X, Wang X, Di S, Cai J y Niu B (2023) Identificación de mutaciones en STAT5A porcino que contribuyen a la transcripción de CISH. Frente. Vet. Sci. 9:1090833. doi: 10.3389/fvets.2022.1090833
Recibido: 06 de noviembre de 2022; Aceptado: 28 de diciembre de 2022;
Publicado: 17 enero 2023.
Editado por:
Yalan Yang, Instituto de Genómica Agrícola de Shenzhen (CAAS), China
Revisado por:
Lingyang Xu, Instituto de Ciencias Animales (CAAS), China Xinyun Li, Universidad Agrícola de Huazhong, China
Derechos de autor © 2023 Yao, Guo, Zhang, Chen, Gao, Xing, Yang, Wang, Di, Cai y Niu. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Buyue Niu,  niubuyue@neau.edu.cn
niubuyue@neau.edu.cn
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía