Resistencia a los antimicrobianos de la salmonella
 Resistencia a los antimicrobianos y características genómicas de la salmonella de pollos de engorde en la provincia de Shandong
Resistencia a los antimicrobianos y características genómicas de la salmonella de pollos de engorde en la provincia de Shandong
 Liyuan Zhao1,2†
Liyuan Zhao1,2†  Gang Liu1†
Gang Liu1†  Wenli Tang2
Wenli Tang2  Canción de Xiangbin2
Canción de Xiangbin2  Xiaoyu Zhao2
Xiaoyu Zhao2  Chu Wang1
Chu Wang1  Youzhi Li2*
Youzhi Li2*  Ming Zou1*
Ming Zou1*- 1Facultad de Medicina Veterinaria, Universidad Agrícola de Qingdao, Qingdao, China
- 2Laboratorio Provincial Clave de Shandong de Monitoreo de Calidad de Seguridad y Evaluación de Riesgos para Productos de Origen Animal, Centro de Shandong para el Control de Calidad de Piensos y Medicamentos Veterinarios, Jinan, China
Introducción: La aparición de cepas multirresistentes (MDR) de Salmonella, que es un género de importantes patógenos zoonóticos, ha despertado una gran preocupación por la salud pública en todo el mundo.
Métodos: En este estudio, se aislaron 167 cepas de Salmonella de 947 muestras de granjas de pollos de engorde, mataderos y mercados de la provincia de Shandong. Se realizaron pruebas de sensibilidad a los antibióticos y se seleccionaron 70 cepas de Salmonella mediante secuenciación del genoma completo (WGS) para evaluar los serotipos, los genes de resistencia a los antimicrobianos (ARG), la prevalencia de integrones de clase 1, la tasa de porte de plásmidos y las características filogenéticas y para la tipificación de secuencias multilocus (MLST).
Resultados: Los resultados mostraron que los 167 aislamientos mostraron la mayor resistencia a ampicilina (AMP, 87,4%), sulfametoxazol (SF, 87,4%), sulfametoxazol compuesto (SXT, 81,4%), ácido nalidíxico (NAL, 80,2%) y amoxicilina/ácido clavulánico (A/C, 77,8%). Todas las cepas fueron sensibles al meropenem (MEM) y el 91,0% de los aislados fueron cepas MDR. Se examinaron un total de 45 ARG, con la tasa de detección más alta observada para el gen de resistencia a tetraciclina (TET) tet (A) (81,4%). Se detectaron un total de 21 tipos de replicones plásmidos en Salmonella, de los cuales IncX1 fue el más común (74,3%), y el 62,9% de los aislados portaban un integrón de clase 1. Además, se detectaron un total de 11 serotipos diferentes, siendo S. enteritidis el serovar predominante, seguido de S. infantis y S. Newport. Se detectaron doce tipos de secuencias (ST) diferentes, entre los cuales ST11 fue el tipo principal. Hubo una fuerte correspondencia entre los serotipos y las ST. También encontramos que S. Indiana y S. Kentucky tenían tasas extremadamente altas de resistencia a la ciprofloxacina (CIP) y a las cefalosporinas de tercera generación. El análisis del genoma de todo el sistema mostró la ocurrencia de transmisión a larga distancia a través de campos.
Conclusión: En conclusión, la detección de multirresistencia y aislamientos portadores de genes multirresistentes es el principal problema, por lo que se deben implementar estrategias de emergencia para abordar este problema.
Introducción
La salmonela es un patógeno común transmitido por los alimentos en todo el mundo que está ampliamente distribuido en el medio ambiente y en la cadena alimentaria mundial, lo que representa una grave amenaza para la seguridad alimentaria y la salud pública. En 2019, la Unión Europea informó de que la salmonelosis es la segunda infección gastrointestinal humana más común después de la infección por Campylobacter y es una causa importante de brotes de transmisión alimentaria en la UE, con 87.923 casos confirmados de salmonelosis humana (1). Hasta la fecha, se han identificado más de 2.600 serovares de Salmonella enterica (2). En Europa, la gran mayoría (72,4%) de los brotes de Salmonella transmitida por los alimentos son causados por S. enteritidis (1). En China, el 70% ~ 80% de los brotes bacterianos transmitidos por los alimentos se pueden atribuir a la infección por Salmonella, mientras que S. enteritidis y S. typhimurium son los serotipos más comunes asociados con las infecciones intestinales humanas (3). En los últimos años, algunos nuevos serotipos, como S. Telkebier, S. Uzaramo y S. Changwanni, se han descubierto gradualmente en China (4-6).
La salmonelosis generalmente se asocia con la ingestión de alimentos de origen animal contaminados con Salmonella, particularmente pollo y productos avícolas, que son la fuente más común de transmisión de Salmonella a los humanos (7). Muchos antibióticos se utilizan actualmente en la producción de animales destinados a la producción de alimentos para promover el crecimiento y prevenir (profiláctica), tratar (terapéutica) y controlar (metafiláctica) la salmonelosis, pero un problema grave asociado con el uso de antibióticos es el desarrollo de resistencia a los antibióticos por parte de los patógenos. El nivel y el grado de resistencia cambian constantemente en todo el mundo y se ven afectados por las prácticas de uso de antimicrobianos en humanos y animales y las diferencias geográficas (8). El uso indiscriminado de antibióticos en la ganadería ha sido identificado como el factor impulsor para el desarrollo de cepas multirresistentes (MDR), y la resistencia puede transmitirse a los humanos a través de la cadena alimentaria (9). Por ejemplo, se han identificado bacterias resistentes a los medicamentos en varias muestras ambientales, granjas y productos cárnicos al por menor (10-14). Con el tiempo, esto reducirá la eficacia de los antibióticos y, finalmente, conducirá al fracaso del tratamiento. Por lo tanto, el uso de «medicamentos antibacterianos cruciales» como las fluoroquinolonas y las cefalosporinas de tercera generación para tratar la Salmonella se clasifica como la máxima prioridad, y estos son los medicamentos antibacterianos más importantes en la medicina humana, y la colistina también se considera la «última línea de defensa» para tratar infecciones graves causadas por patógenos gramnegativos MDR (15).
La resistencia puede ocurrir a través de mutaciones puntuales en el genoma bacteriano o transferencia horizontal a través de elementos genéticos portadores de genes de resistencia a antibióticos (ARG) (16). La forma más efectiva por la cual los ARG se transfieren entre microorganismos es la transferencia horizontal a través de elementos genéticos móviles, que se puede lograr mediante la inclusión de uno o más integrones, transposones y plásmidos que albergan un ARG (17, 18). Esta capacidad de transferencia horizontal aumenta el riesgo de fracaso del tratamiento para las infecciones clínicas causadas por S.almonella. Por lo tanto, es particularmente importante monitorear la resistencia de Salmonella a estos antibióticos. Se ha demostrado que la secuenciación del genoma completo (WGS), un método molecular para caracterizar organismos, es un método de seguimiento rápido, específico y rentable en comparación con los métodos anteriores (19) y, lo que es más importante, la WGS puede utilizarse para detectar y predecir amenazas emergentes que pueden dar lugar a infecciones graves en humanos y animales en las primeras etapas (20).
La provincia de Shandong es la provincia de producción avícola más grande de China, representando el 30,0% de la producción total de China (21). En China, especialmente en la provincia de Shandong, se han notificado varios brotes recurrentes de salmonelosis aviar durante la última década, en los que se desconoce el agente causal preciso. Los estudios previos sobre la prevalencia y la resistencia a los medicamentos de Salmonella en pollos de engorde en la provincia de Shandong se limitan a una determinada región o enlace, sin investigación horizontal a gran escala (22-24). Este estudio caracterizó los aislamientos relevantes de pollos de engorde e investigó la diversidad fenotípica y genotípica de Salmonella de diferentes fuentes y sus relaciones genéticas mediante la realización de pruebas de sensibilidad a medicamentos y análisis de WGS en pollos de engorde y sus productos en la provincia de Shandong. Nuestros hallazgos contribuyen a evaluar los riesgos para la salud pública y proporcionan información sobre la prevención de la contaminación por Salmonella y la resistencia a los medicamentos.
Materiales y métodos
Recogida de muestras
Se recolectaron aleatoriamente un total de 947 muestras de 15 granjas de pollos de engorde (n = 618), 2 mataderos de pollos de engorde (n = 293) y 3 mercados (n = 36) en la provincia de Shandong, incluidos hisopos cloacales, hisopos ambientales (herramientas de segmentación, agua, contenedores) e hisopos de canales. El conjunto de muestras incluyó 8 de las principales ciudades productoras de pollo de la provincia de Shandong. El número de muestras cloacales recolectadas en cada granja varió de 30 a 86, las muestras de canales recolectadas en cada matadero variaron de 73 a 125, las muestras ambientales recolectadas en cada matadero variaron de 35 a 60 y se recolectaron 12 muestras de productos de pollo de cada mercado. Todas las muestras se recogieron con eSwabs con 1 mL de medio Amies líquido (Copán Brescia, Italia). Se almacenaron a 4 °C en un refrigerador y se transfirieron al laboratorio para su procesamiento dentro de las 24 h. La información de distribución de las cepas se muestra en la Tabla Suplementaria S1.
Aislamiento e identificación de bacterias
El aislamiento de Salmonella se realizó de acuerdo con los métodos descritos anteriormente (25). Brevemente, las muestras de hisopos fecales o cloacales se enriquecieron en 10 mL de agua de peptona tamponada (BPW; Landbridge, Pekín, China). Tras el preenriquecimiento inicial en BPW, se añadieron 0,1 mL de la muestra preenriquecida a 10 mL de caldo de selenito cistina (Landbridge, Pekín, China) y se incubó a 37°C durante 12 a 18 h. El aislamiento final de la colonia se realizó en agar xilosa-lisina-tergitol-4 (BD Biosciences, Estados Unidos). Los aislados positivos de Salmonella se identificaron además mediante espectrometría de masas de tiempo de vuelo por ionización por desorción láser asistida por matriz (MALDI-TOF MS) (Bruker MALDI Biotyper System, Alemania).
Pruebas de susceptibilidad a los antimicrobianos
Las concentraciones mínimas inhibitorias (CMI) de los aislados de Salmonella se determinaron mediante el método de microdilución en caldo, que empleó los siguientes 20 agentes antimicrobianos: ampicilina (AMP), amoxicilina/ácido clavulánico (A/C), gentamicina (GEM), tetraciclina (TET), ceftazidima (CAZ), colistina (CL), meropenem (MEM), sulfisoxazol (SF), espectinomicina (SPE), enrofloxacina (ENR), ofloxacino (OFX), amikacina (AMK), doxiciclina (DOX), ácido nalidíxico (NAL), sulfametoxazol compuesto (SXT), ceftiofur (CEF), ciprofloxacina (CIP), kanamicina (KAN), cloranfenicol (CHL) y florfenicol (FLO). E. coli Se utilizó ATCC 25922 como cepa de control siguiendo las directrices del CLSI (26). Los puntos de ruptura resistentes utilizados fueron los siguientes: ≥32 mg/L para AMP, ≥32/16 mg/L para A/C, ≥8 mg/L para GEN, ≥16 mg/L para TET, ≥16 mg/L para CAZ, ≥4 mg/L para CL, ≥4 mg/L para MEM, ≥512 mg/L para SF, ≥2 mg/L para ENR, ≥8 mg/L para OFX, ≥16 mg/L para AMK, ≥16 mg/L para DOX, ≥32 mg/L para NAL, ≥4/76 mg/L para SXT, ≥4 mg/L para CIP, ≥64 mg/L para KAN y ≥ 32 mg/L para CHL (CLSI M100-ED33). Mientras que el punto de ruptura de resistencia utilizado para SPE (≥128 mg/L) fue seguido por el punto de corte clínico para Pasteurella multocida-bovino, CEF (≥8 mg/L) y FLO (≥16 mg/L) fueron seguidos por el punto de corte clínico para Enterobacterales-porcino (CLSI VET01S ED6). La resistencia a tres o más clases de fármacos se consideró resistencia múltiple.
Análisis de secuenciación del genoma completo
El TIANamp Bacteria DNA Kit (Tiangen Biotech, Pekín, China) se utilizó de acuerdo con las recomendaciones del fabricante para extraer el ADN genómico de 70 cepas de Salmonella seleccionadas (la selección de las 70 cepas de Salmonella se basó en la sensibilidad a los medicamentos, diferentes fuentes regionales y diferentes procesos de producción). Posteriormente, se llevaron a cabo pruebas de calidad de ADN bacteriano y la construcción de la biblioteca de ADN de Salmonella, y se realizó WGS utilizando la plataforma Illumina NovaSeq 6,000 (Sinobiocore, Beijing, China). Se utilizaron herramientas en línea para enviar secuencias completas de nucleótidos al servidor web del Centro de Epidemiología Genómica1 para la tipificación de plásmidos, la identificación de ARG y la tipificación de secuencias multilocus (MLST). Además, se obtuvo un tipo de integrón a través de la anotación de genes en el sitio web de Rast.2 El gen central se definió como un gen que estaba presente en el ≥99% de los genomas. Todos los genes centrales se utilizaron para construir árboles filogenéticos utilizando Fasttree v.2.1.11. Los mapas de calor de la agrupación de ARG y serotipos se crearon utilizando TBtools v.1.116.
Análisis de datos
El análisis de los resultados obtenidos y la generación de figuras se realizaron utilizando el GraphPad Prism (GraphPad, San Diego, CA, Estados Unidos) versión 7.03, y en este estudio se utilizó la prueba t de Student con corrección de Welch.
Resultados
Resistencia a antibióticos y perfiles MDR
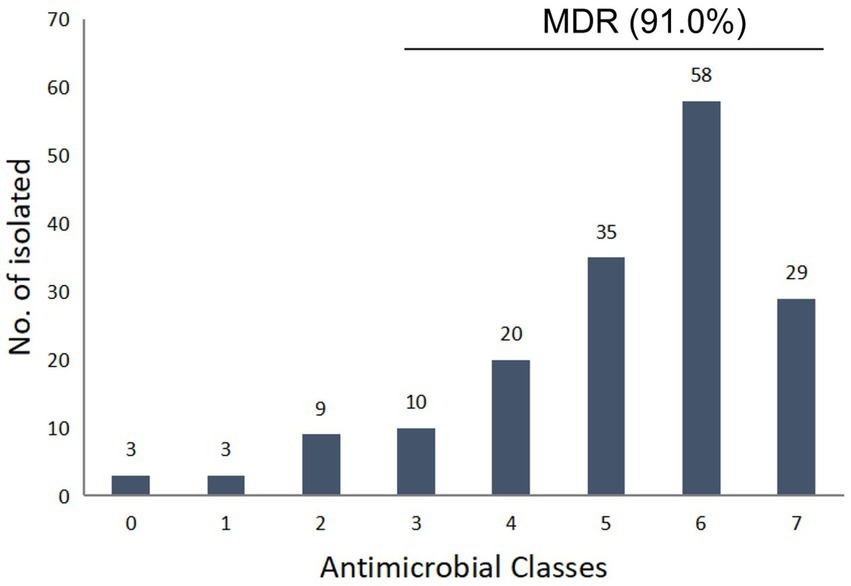
Entre las 947 muestras, se identificaron un total de 167 cepas de Salmonella, y la tasa de contaminación total de pollos de engorde y productos fue de 17.6%, con una tasa de positividad de 32.8% en mataderos, que es significativamente mayor que las granjas avícolas (11.2%) y muestras de mercado (5.6%), respectivamente (p < 0.05). Los resultados de las pruebas de susceptibilidad a los antimicrobianos mostraron que los aislados de Salmonella obtenidos tenían la mayor resistencia (˃80%) a AMP (87,4%), SF (87,4%), SXT (81,4%), NAL (80,2%), alta resistencia (50-80%) a A/C (77,8%), TET (66,5%), DOX (65,3%), CL (62,9%), FLO (53,2%) y CHL (51,5%) y resistencia moderada (30-50%) a KAN (47,9%), SPE (47,9%), CEF (44,9%), OFX (33,5%) (Tabla 1). Un total de 26,3 y 21,0% de las cepas mostraron resistencia a cefalosporinas de tercera generación y CIP, respectivamente (Tabla 1). El 94,4% de las cepas resistentes a CIP también son resistentes a NAL y OFX, y el 61,1% de las cepas resistentes a CIP también son resistentes a ENR. La mayoría de los fármacos tenían una CMI alta50 y MIC90 especialmente AMP, SF y NAL, el MIC50 y MIC90 de los cuales fueron superiores a la concentración más alta ensayada (>128 mg/L, >512 mg/L y >128 mg/L, respectivamente). Los aislados mostraron menor resistencia a AMK (5,4%), y todas las cepas mostraron sensibilidad a MEM. De los 167 aislamientos, 164 (98,2%) fueron resistentes a al menos un agente antimicrobiano, mientras que 3 (1,8%) fueron sensibles a todos los agentes antimicrobianos probados. Un total de 152 cepas aisladas (91,0%) presentaron multirresistencia, siendo 5 y 6 cepas las que concentraron la mayor proporción, representando el 21,0 y el 34,7% del total de bacterias, respectivamente (Figura 1). Los aislados de granjas avícolas mostraron la mayor resistencia a AMP, NAL, SF, SXT y FLO, con múltiples cepas resistentes que representaron el 91,3% del total de aislamientos de granjas; las cepas aisladas de los mataderos mostraron la mayor resistencia a SF, AMP, SXT, A/C, NAL y CL, con múltiples cepas resistentes que representaron el 90,6% del total de cepas aisladas de los mataderos; todas las cepas procedentes de los mercados son cepas multirresistentes (cuadro suplementario S2).
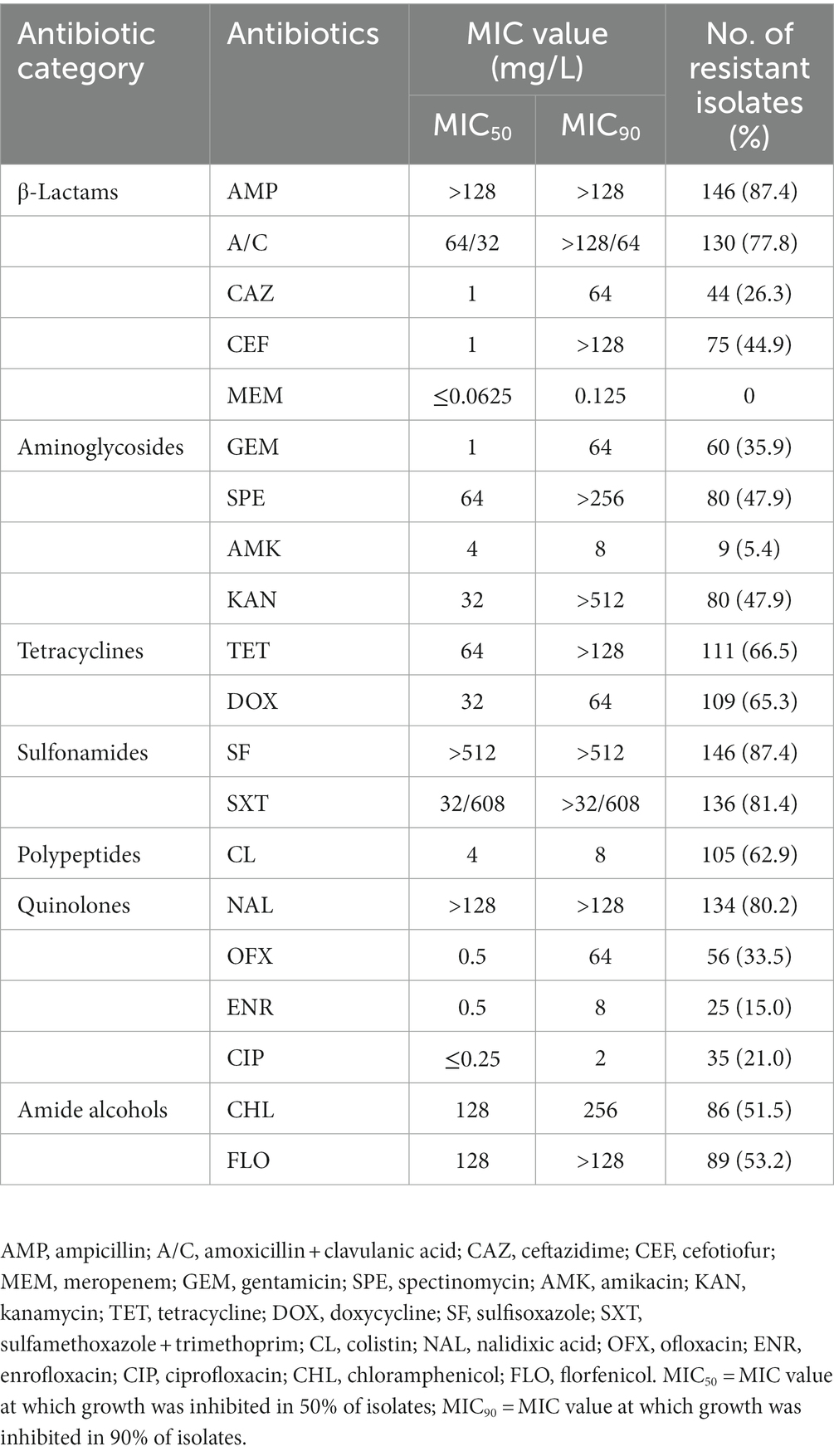 Tabla 1. Distribuciones de las CMI de 167 aislamientos de Salmonella frente a 20 agentes antimicrobianos.
Tabla 1. Distribuciones de las CMI de 167 aislamientos de Salmonella frente a 20 agentes antimicrobianos.
Genes y serotipos de resistencia a los antimicrobianos
Se examinaron un total de 45 ARG de las 70 cepas secuenciadas, y se encontró que el 81,4% de los aislados portaban el gen tet (A), lo que confirió resistencia a TET. Las tasas de portación de los genes floR y cmlE fueron del 71,4%, lo que dotó a las cepas de resistencia a los fármacos fenólicos. La tasa de detección de genes de resistencia a β-lactámicos fue la más alta para blaTEM (50.0%). La tasa de detección de genes de resistencia a aminoglucósidos fue la más alta en el grupo aph (4) – Ia (51,4%); la tasa de detección de genes de resistencia a quinolonas fue mayor para qnrS1 (31,4%). También encontramos que el 7,1% de las cepas portaban genes de resistencia a la colistina mcr-1, lo cual es un hallazgo particularmente notable (Tabla Suplementaria S2). Se identificaron un total de 11 serotipos diferentes por secuenciación, siendo S. enteritidis el serotipo principal (31,4%), seguido de S. infantis (20,0%), S. Newport (17,1%), S. Kentucky (11,4%), S. Indiana (10,0%) y S. Thompson (2,9%). Además, también se detectaron S. Kedougou, S. Mbandaka, S. Ohio y S. Lexington. La portación de ARGs varió entre los diferentes aislados de serotipo, y en comparación con otros serotipos, S. Indiana portó la mayor cantidad de ARGs (Figura 2).
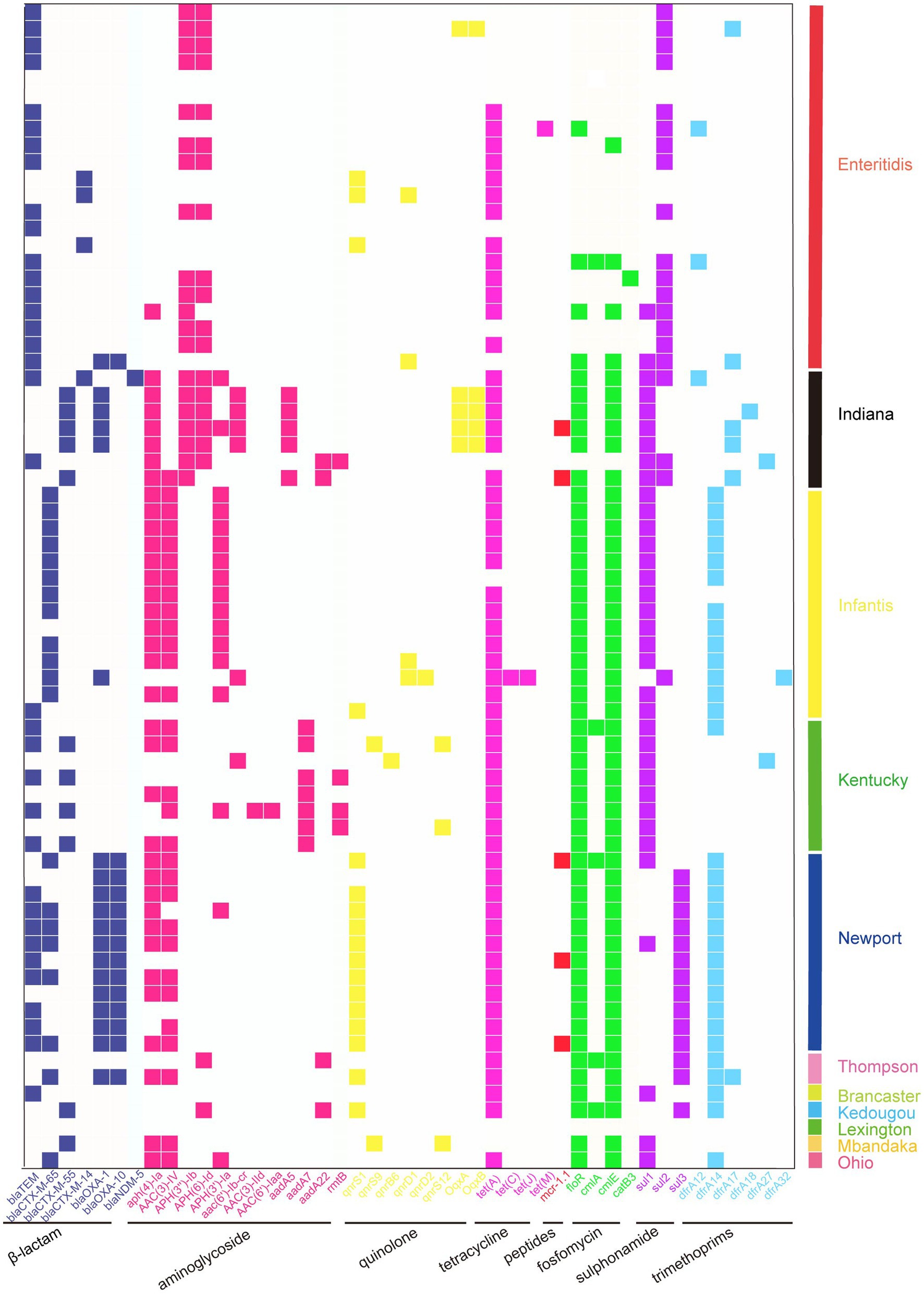 Figura 2. Mapa de calor que muestra los perfiles ARG en este trabajo. Los diferentes grupos de ARG están codificados por colores. Distribución de los ARGs entre diferentes serotipos de Salmonella.
Figura 2. Mapa de calor que muestra los perfiles ARG en este trabajo. Los diferentes grupos de ARG están codificados por colores. Distribución de los ARGs entre diferentes serotipos de Salmonella.
Tipificación de plásmidos e integrones de clase 1
Se detectaron un total de 21 tipos de replicones de todas las cepas secuenciadas. Entre los tipos de replicones plásmidos, IncX1, IncFII (S), IncFIB (S), IncHI2A, IncHI2 e IncQ1 fueron los más comunes, y el resto incluyó IncX4, IncX8, IncC, IncN, IncI1-I (Alpha), IncFIB (K), Col8282, IncI2, IncFIB (AP001918), Col3M, IncR, IncI1, ColpVC, Col156 y Col440I. La tasa total de detección de integrones de clase I fue del 62,9%. Todas las cepas de Salmonella portadoras de integrones fueron cepas MDR (Tabla Suplementaria S2). La mayoría de las cepas portaban integrones de clase I y replicones plásmidos (Figura 3).
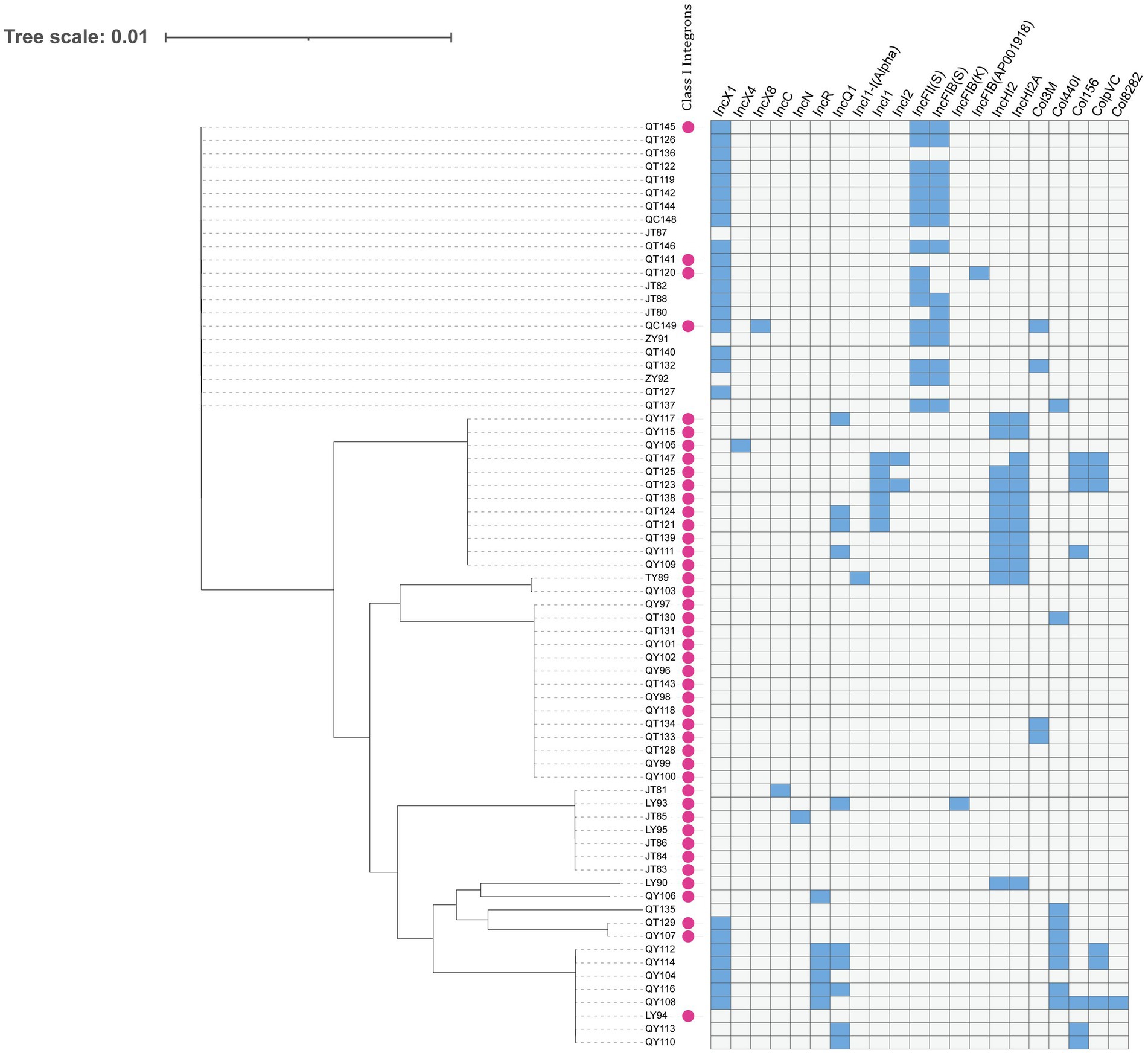 Figura 3. Dendrograma de un mapa de calor de agrupamiento jerárquico de los 70 aislados, 21 replicones de plásmidos e integrones de clase 1. Las células azules representan la presencia de plásmido; Las células grises representan la ausencia de plásmido. Los integrones de clase 1 están representados por círculos morados.
Figura 3. Dendrograma de un mapa de calor de agrupamiento jerárquico de los 70 aislados, 21 replicones de plásmidos e integrones de clase 1. Las células azules representan la presencia de plásmido; Las células grises representan la ausencia de plásmido. Los integrones de clase 1 están representados por círculos morados.
MLST y relaciones filogenéticas
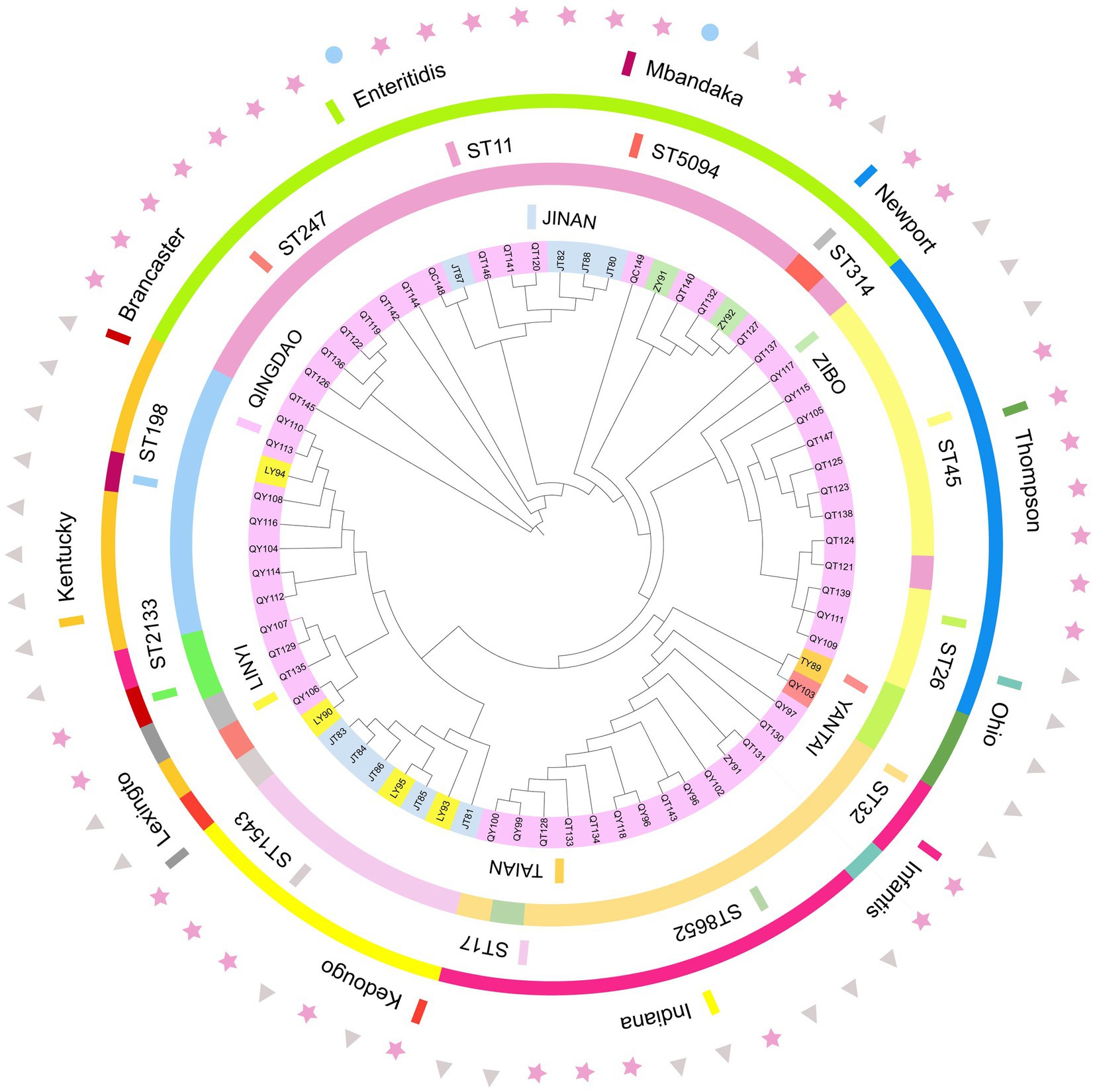
Se identificaron un total de 12 tipos de secuencia (ST) de las 70 cepas secuenciadas, siendo ST11 el tipo principal, seguido de ST32 y ST45. Otros tipos de secuencias fueron ST198, ST17, ST26, ST2133, ST247, ST314, ST1543, ST5094 y ST8652. En este estudio, el 95,5% de las cepas de S. enteritidis fueron ST11, el 91,7% de las cepas de S. Newport fueron ST45, el 92,3% de las cepas de S. infantis fueron ST32, el 100% de las cepas de S. Indiana fueron ST17 y las cepas de S. Kentucky fueron principalmente ST198, lo que indica que el serotipo y el ST tienen una tasa de coincidencia muy alta. Hubo 10 ST en las granjas de pollos de engorde, siendo ST198 el tipo principal. Había 7 ST en los mataderos, siendo ST11 el tipo principal. El ST de las muestras de mercado fue ST11. El árbol filogenético de Salmonella muestra que se formaron un total de 10 ramas principales. Las cepas de la misma región estaban más cerca en las ramas evolutivas y compartían estrechas relaciones genéticas; también hubo estrechas relaciones genéticas entre el mismo serotipo y el ST. Entre las muestras, las muestras de mercado se localizaron en dos ramas evolutivas separadas, lo que indica que había otras fuentes de contaminación además de las granjas y los mataderos (Figura 4).
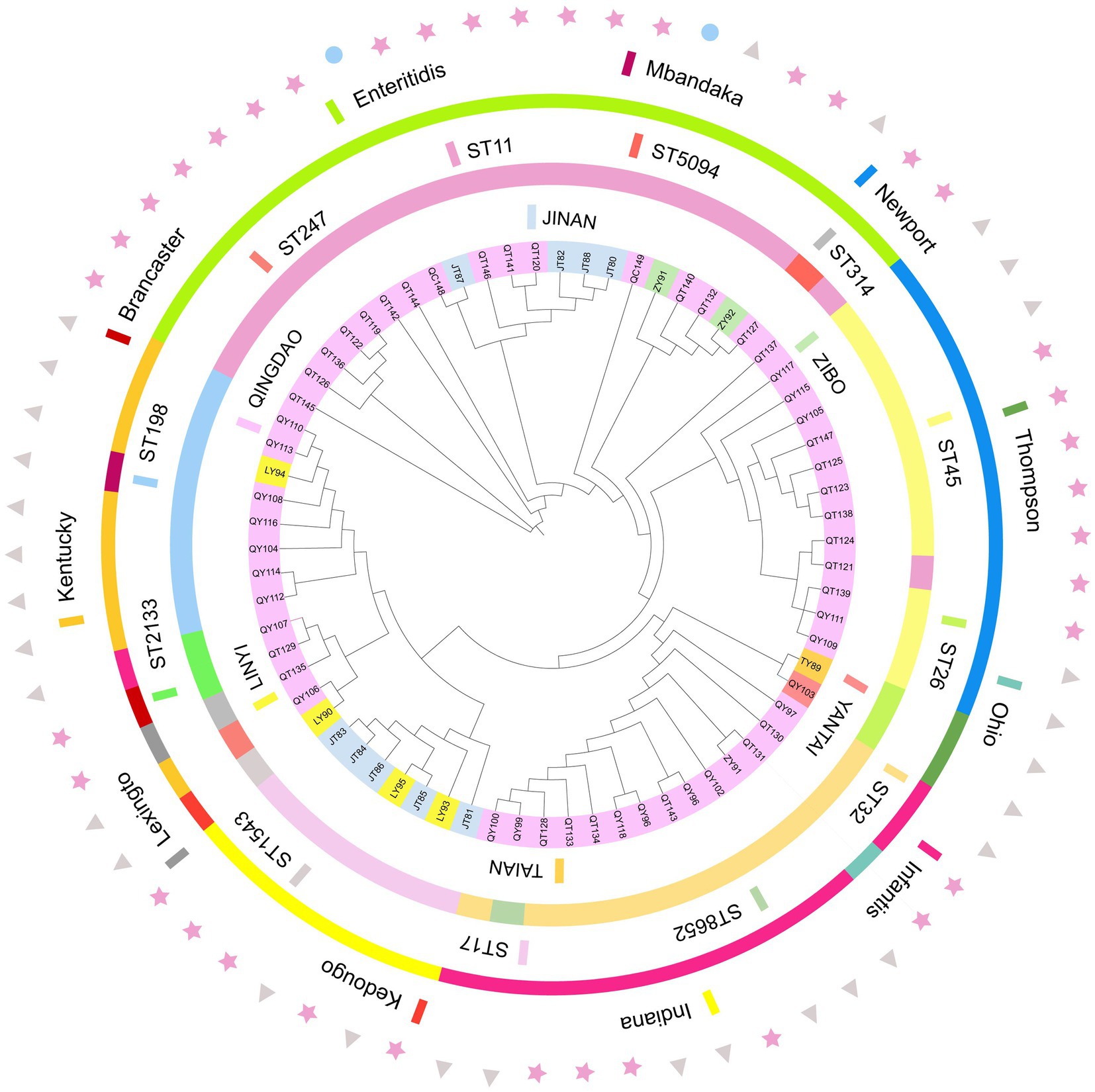 Figura 4. Relaciones filogenéticas de 70 cepas de bacterias. El árbol se creó utilizando la interfaz de usuario interactiva anotada de iTOL (https://itol.embl.de). Los círculos, de adentro hacia afuera, indican las fuentes regionales de cepas (círculo 1), el ST de la cepa (círculo 2) y el tipo de serotipo de la cepa (círculo 3); Los triángulos representan las granjas, las estrellas representan los mataderos y los círculos representan los mercados (círculo 4).
Figura 4. Relaciones filogenéticas de 70 cepas de bacterias. El árbol se creó utilizando la interfaz de usuario interactiva anotada de iTOL (https://itol.embl.de). Los círculos, de adentro hacia afuera, indican las fuentes regionales de cepas (círculo 1), el ST de la cepa (círculo 2) y el tipo de serotipo de la cepa (círculo 3); Los triángulos representan las granjas, las estrellas representan los mataderos y los círculos representan los mercados (círculo 4).
Discusión
En este estudio, la tasa de positividad en las granjas avícolas de la provincia de Shandong fue del 11,2%, ligeramente superior a la tasa de positividad en las granjas de pollos de engorde del sudeste asiático y las regiones de Zhejiang y Fujian (27, 28). La salmonela en los mataderos se ha detectado en varias etapas del sacrificio, lo que indica que puede propagarse a lo largo de la línea de sacrificio. La tasa de detección de Salmonella en las muestras de mercado fue consistente con los resultados de investigaciones previas (29).
Debido al uso excesivo de antibióticos y a la transmisión horizontal de los ARG en la cría de animales en las últimas décadas, la resistencia a los antibióticos de Salmonella se ha convertido en una amenaza mundial (30). Los antibióticos seleccionados en este estudio son las clases más utilizadas en la cadena de producción avícola en la provincia de Shandong, el uso a largo plazo de estos antibióticos puede provocar problemas de resistencia a los antibióticos (31). Entre las 167 cepas de Salmonella en este estudio, 152 (91,0%) fueron resistentes a más de tres fármacos y ampliamente resistentes a los antibióticos convencionales. El MIC50 y MIC90 puede reflejar la resistencia a los medicamentos de Salmonella en su conjunto. Los resultados mostraron que la tasa de resistencia a sulfonamidas, AMP y NAL fue la más alta, y la resistencia a A/C y TET también fue generalmente alta. El MIC50 y MIC90 de estos antibióticos también superó con creces el punto de ruptura de la resistencia a los medicamentos, lo que puede deberse a que estos son los antibióticos más comúnmente utilizados para tratar varias enfermedades infecciosas en las aves de corral. La resistencia a cefalosporinas de tercera generación fue moderada. La tasa de resistencia para el fármaco de quinolonas NAL alcanzó el 80,2% (CMI50 > 128 mg/L, CMI90 > 128 mg/L), y esta alta tasa de resistencia también se ha encontrado en otras partes de China y otras regiones (21, 32-34), especialmente para las combinaciones de trimetoprima y sulfonamidas. Esta situación puede deberse al uso generalizado de medicamentos con quinolonas en los piensos para la cría de ganado y aves de corral en China, lo que conduce a una presión selectiva sobre las bacterias (30). En las 70 cepas secuenciadas, se detectaron 45 ARG, y las cepas aisladas portaban diferentes genes de resistencia, lo que probablemente esté relacionado con la complejidad de los fenotipos de resistencia a la cepa. Los genes de resistencia de la mayoría de las cepas fueron consistentes con sus fenotipos, mientras que algunas cepas tenían ciertos fenotipos de resistencia pero no portaban los ARG correspondientes, lo que puede estar relacionado con mecanismos de resistencia desconocidos o el funcionamiento inespecífico de múltiples genes redundantes similares a bombas de eflujo. Del mismo modo, la presencia de genes de resistencia no conduce necesariamente a la resistencia fenotípica y, a veces, los aislados muestran resistencia a los antibióticos sin albergar genes de resistencia conocidos (35). En nuestro estudio, blaTEM fue el gen de resistencia más común. La tasa de detección del gen de resistencia a aminoglucósidos aph (4) – Ia fue la más alta, y un pequeño número de cepas albergaba el gen aac (6′) – Ib-cr. El gen aac (6′) – Ib-cr es un mediador clave de la resistencia bacteriana a la CIP (36). Además, se detectaron múltiples genes de resistencia a quinolonas, incluidos qnrS1, OqxA, OqxB, qnrS12, qnrD1, qnrS9, qnrB6 y qnrD2. La alta prevalencia de genes de resistencia a quinolonas mediada por plásmidos (PMQR) destaca la importancia del uso cauteloso de los fármacos fluoroquinolonas para minimizar la resistencia a las fluoroquinolonas.
El análisis del serotipo mostró una diversidad significativa, con S. enteritidis presente en la mayoría de las muestras, lo que indica que tiene una ventaja absoluta, lo cual es consistente con los resultados de otros estudios en China (37, 38). S. Indiana puede tener un mayor potencial de resistencia, ya que transportó más ARG. Más importante aún, S. Indiana era 100% resistente a CIP y cefalosporinas de tercera generación, y una de las cepas de S. Indiana también portaba el bla NDM-5 gen de resistencia. Además, S. Kentucky también fue 100% resistente a CIP y tuvo una alta resistencia a las cefalosporinas de tercera generación, lo que fue similar a los resultados de investigaciones anteriores (39). El gen de la betalactamasa de espectro extendido (BLEE) es el determinante más importante de la resistencia a las cefalosporinas de tercera generación en Salmonella y blaCTX-M-55 es el gen dominante de la BLEE. En nuestro estudio, este ARG se detectó principalmente en los serotipos de Indiana y Kentucky, y en el blaCTX-M-55 El gen también se detectó principalmente en S. Kentucky (29). La CIP y las cefalosporinas son los fármacos más utilizados para tratar la salmonelosis en humanos. La doble resistencia a la CIP y a las cefalosporinas de tercera generación supone una enorme amenaza para la salud humana, ya que el fracaso del tratamiento puede tener graves consecuencias (40). Por lo tanto, el sur de Indiana y el sur de Kentucky pueden tener un mayor riesgo de resistencia a los medicamentos. También descubrimos un cierto número de serotipos de S. Newport, que han causado varios brotes en los Estados Unidos, infectando a millones de personas anualmente (41), y a menudo se detectan en cadenas avícolas en Brasil (42). Ha habido informes de este serotipo en otras provincias de China, pero no ha causado brotes a gran escala en China. Se cree que la resistencia a CL es causada principalmente por el gen mcr-1 ubicado en plásmidos transferibles, que se descubrió por primera vez en China a partir de animales, alimentos y humanos (43). En nuestro estudio, los aislados de S. Newport fueron 100% resistentes a CL, con tres cepas portadoras del gen mcr-1 (44). S. Newport con positividad para mcr-1 también se encontró entre los aislados de pollo. Una de las tres cepas mcr-1 positivas portaba el plásmido IncX4, mientras que las otras dos cepas mcr-1 positivas portaban el plásmido IncI2, y las tres cepas portaban integrones de clase 1. Algunos estudios han demostrado que los principales tipos de replicones portadores del plásmido mcr-1 son IncI2, IncX4, IncHI2 e IncP, lo que es consistente con nuestra investigación. En otros estudios, el gen mcr-1 también se encontró en los plásmidos IncX4 e IncI2 (45). Además, los estudios han demostrado que los plásmidos IncX4 positivos para mcr pueden propagarse entre diferentes especies bacterianas, de animales a humanos o de humanos a animales (46). Los plásmidos se consideran los principales elementos móviles que determinan la transferencia horizontal de genes (47). En nuestro estudio se detectaron un total de 21 plásmidos diferentes, entre los cuales IncX1 e IncFII (S) fueron plásmidos dominantes. Estos plásmidos se distribuyeron en diferentes regiones, etapas de reproducción y serotipos, lo que indica que estos plásmidos pueden estar ampliamente diseminados entre diferentes serotipos y regiones. Los integrones son elementos genéticos móviles que codifican genes bacterianos relacionados con la resistencia a los antibióticos que pueden transmitirse entre microorganismos. El integrón más común en Salmonella MDR es el integrón de clase 1 (48). Todas las cepas de S. Indiana, S. infantis, S. Newport y S. Thompson que aislamos portaban integrones de clase 1, mientras que solo tres cepas de S. enteritidis portaban integrones de clase 1. También encontramos que algunos ARG, replicones plásmidos e integrones de clase 1 existían simultáneamente en la misma cepa, lo que puede ser muy propicio para la transmisión horizontal de la resistencia a los medicamentos entre cepas.
Encontramos una fuerte correspondencia entre serotipos y STs, y este fenómeno también se ha observado en otros estudios (31). Debido a estas correlaciones, es posible predecir los serotipos de Salmonella hasta cierto punto. Los resultados del árbol filogenético mostraron que las cepas con el mismo tipo de secuencia se agruparon fácilmente, lo que confirma la homología genética de los aislados del mismo serotipo/ST. Las cepas aisladas de la misma granja se agruparon, lo que indica que las cepas de la misma granja compartían un ancestro común, originado en el mismo clon, y que la transmisión clonal se produjo en la granja, lo que probablemente fue causado por la falta de medidas de desinfección en la granja, lo que permitió que los patógenos se propagaran en el campo y persistieran en la granja. dando lugar a una transmisión vertical (49). Además, había relaciones genéticas muy estrechas entre los aislados de cuatro granjas en la misma región, y estas granjas pueden haber albergado las mismas cepas clonadas. Las cepas ZY91, QT140, QT132, ZY92 y QT127 se agruparon, pero se derivaron de diferentes regiones, etapas de aislamiento y fuentes de muestra. Esto indica que los clones de la misma cepa pueden propagarse a largas distancias a través del comercio de pollos de engorde o las cadenas de producción de alimentos, como las del pollo. Como último eslabón de toda la cadena de producción de pollos de engorde, los productos de pollos de engorde están estrechamente relacionados con la vida humana. Dos muestras de pollos de los mercados se localizaron en dos sucursales separadas, lo que indica que hubo otras fuentes de transmisión además de las granjas y los mataderos, durante el transporte del producto. Las cepas aisladas de los mercados fueron del serotipo S. enteritidis. Que es una de las principales causas de los brotes de enfermedades transmitidas por los alimentos (50). Los datos de vigilancia de la salmonela muestran que, desde la década de 1970, la tasa de incidencia de S. enteritidis en los alimentos ha ido en aumento en todo el mundo (51), y este crecimiento mundial está relacionado con los productos de pollo, como la carne de aves de corral y los huevos (52). Existe una necesidad urgente de llamar la atención sobre este problema y adoptar medidas adecuadas en la cría de animales y la cocción de los alimentos para reducir la aparición de Salmonella en las aves de corral y los productos avícolas. Deben aplicarse planes de vigilancia en todas las fases de la producción avícola, incluso durante la cría de las manadas de pollos, en los criaderos, en las manadas de pollos de engorde, en los mataderos y en los lugares de venta, lo que podría reducir significativamente la contaminación por Salmonella de los pollos de engorde y los productos de los pollos de engorde. Y lo que es más importante, los dos aislados del mercado eran cepas MDR y portaban una variedad de ARG y plásmidos, lo que suponía un alto riesgo de transferencia de resistencia a los antibióticos. La aparición y propagación de Salmonella resistente a los medicamentos a partir de animales destinados a la producción de alimentos o carne al por menor se han convertido en graves peligros para la salud en todo el mundo, lo que supone una grave amenaza para la salud pública.
Conclusión
Este estudio encontró que había una cantidad considerable de contaminación por Salmonella en granjas de pollos de engorde, mataderos y sitios de procesamiento y venta al por menor en Shandong, con una variedad de serotipos detectados. Además, algunas cepas de Salmonella aisladas en este estudio eran cepas MDR que ponen en peligro la salud pública y tienen el potencial de propagarse horizontalmente, lo que representa un grave riesgo para la seguridad alimentaria. Por lo tanto, es necesario implementar sistemas de gestión de medicamentos más estrictos para minimizar el riesgo de promover aún más la propagación de cepas resistentes a los medicamentos de estas bacterias peligrosas.
Declaración de disponibilidad de datos
Los datos presentados en el estudio están depositados en el repositorio del NCBI, número de acceso PRJNA1013903.
Declaración ética
Los estudios con animales fueron aprobados por la Universidad Agrícola de Qingdao. Los estudios se llevaron a cabo de acuerdo con la legislación local y los requisitos institucionales. Se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio.
Contribuciones de los autores
LZ: Curación de datos, Análisis formal, Metodología, Redacción – borrador original. GL: Conceptualización, Supervisión, Redacción – Revisión y Edición. WT: Conceptualización, Redacción – revisión y edición, Administración de proyectos. XS: Conceptualización, Administración de proyectos, Redacción, revisión y edición. XZ: Conceptualización, Administración de proyectos, Redacción, revisión y edición. CW: Redacción – revisión y edición, curación de datos. YL: Redacción, revisión y edición, adquisición de fondos. MZ: Adquisición de fondos, redacción, revisión y edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este trabajo fue apoyado por el Plan Clave de Investigación y Desarrollo de la Provincia de Shandong (2022CXGC010606-01-05); el Programa Clave de Investigación y Desarrollo de la Provincia de Shandong, China (2022CXPT022); el Proyecto Especial Clave para la Innovación Tecnológica y el Desarrollo de Aplicaciones de la Ciudad de Chongqing (CSTB2022TIAD-LUX0001); el programa clave de investigación y desarrollo de la provincia de Shandong, China (2022CXPT010); la Fundación Nacional de Ciencias Naturales de China (subvención Nº 32200151).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1292401/full#supplementary-material
Notas
Referencias
1. EFS, A, y Control, ECFD. Informe One Health 2020 Zoonosis de la Unión Europea. EFSA J. (2021) 19:E6971:E06971. doi: 10.2903/j.efsa.2021.6971
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Támesis, HT, y Sukumaran, AT. Una revisión de Salmonella y Campylobacter en carne de pollo de engorde: desafíos emergentes y medidas de seguridad alimentaria. Alimentos. (2020) 9:9. doi: 10.3390/foods9060776
3. Wang, W, Zhao, L, Hu, Y, Dottorini, T, Fanning, S, Xu, J, et al. Estudio epidemiológico sobre la prevalencia, la diversidad de serovar, la multirresistencia y las β-lactamasas de espectro extendido tipo CTX-M de Salmonella spp. de pacientes con diarrea, alimentos de origen animal y mascotas en varias provincias de China. Agentes antimicrobianos Ch. (2020) 64:e20–92. doi: 10.1128/AAC.00092-20
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Zhang, JQ, Zhu, AJ, Yuan, SJ, Huang, SJ, Luo, XH y Xu, XB. La primera detección de bacterias sedimentarias en Changwan, ciudad de Yuyao. Vigilancia de la enfermedad de la provincia de Zhejiang. (2017) 32:399–401. doi: 10.3784/j.issn.1003-9961.2017.05.011
5. Xu, X, Chen, Y, Pan, H, Pang, Z, Li, F, Peng, X, et al. Caracterización genómica de Salmonella Uzaramo para infección invasiva humana. Genoma Microb. (2020) 6:mgen000401. doi: 10.1099/mgen.0.000401
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Qiu, YF, Nambiar, RB, Xu, XB, Weng, ST, Pan, H, Zheng, KC, et al. Caracterización genómica global de Salmonella enterica Serovar Telelkebir. Microbiol frontal. (2021) 12:704152. doi: 10.3389/fmicb.2021.704152
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Dong, QL, Barker, GC, Gorris, LG, Tian, MS, Song, XY y Malakar, PK. Estado y futuro de la evaluación cuantitativa de riesgos microbiológicos en China. Tendencias Food Sci Technol. (2015) 42:70–80. doi: 10.1016/j.tifs.2014.12.003
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Zhao, S, McDermott, PF, Friedman, S, Qaiyumi, S, Abbott, J, Kiessling, C, et al. Caracterización de Salmonella resistente a antimicrobianos aislada de alimentos importados. J Food Prot. (2006) 69:500–7. doi: 10.4315/0362-028x-69.3.500
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Eiamsam-Ang, T, Tadee, P, Pascoe, B y Patchanee, P. Análisis basado en el genoma de serotipos poco frecuentes de Salmonella a través de la cadena de producción de carne de cerdo tailandesa. Microbiol frontal. (2022) 13:968695. doi: 10.3389/fmicb.2022.968695
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Sneeringer, S, MacDonald, J, Key, N, McBride, W y Mathews, K. Economía del uso de antibióticos en la producción porcina y avícola de EE. UU. Opciones. (2015) 30:1–11. Disponible en: https://www.jstor.org/stable/choices.30.1.06
11. Liljebjelke, KA, Hofacre, CL, White, DG, Ayers, S, Lee, MD y Maurer, JJ. Diversidad de fenotipos de resistencia a los antimicrobianos en Salmonella aislada de granjas avícolas comerciales. Front Vet Sci. (2017) 4:96. doi: 10.3389/fvets.2017.00096
12. Medeiros, MA, Oliveira, DC, Rodrigues, DP y Freitas, DR. Prevalencia y resistencia a los antimicrobianos de Salmonella en canales de pollo al por menor en 15 ciudades brasileñas. Rev Panam Salud Pública. (2011) 30:555–60. doi: 10.1590/s1020-49892011001200010
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Landers, TF, Cohen, B, Wittum, TE, y Larson, EL. Una revisión del uso de antibióticos en animales destinados a la alimentación: perspectiva, política y potencial. Representante de Salud Pública (2012) 127:4–22. doi: 10.1177/003335491212700103
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Parveen, S, Taabodi, M, Schwarz, JG, Oscar, TP, Harter-Dennis, J y White, DG. Prevalencia y resistencia antimicrobiana de Salmonella recuperada de aves de corral procesadas. J Food Prot. (2007) 70:2466–72. doi: 10.4315/0362-028x-70.11.2466
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Sun, J, Zhang, H, Liu, YH y Feng, Y. Hacia la comprensión de la resistencia a la colistina similar a MCR. Tendencias Microbiol. (2018) 26:794–808. doi: 10.1016/j.tim.2018.02.006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Rakitin, AL, Yushina, YK, Zaiko, EV, Bataeva, DS, Kuznetsova, OA, Semenova, AA, et al. Evaluación de la resistencia a antibióticos de los serotipos de Salmonella y secuenciación del genoma completo de cepas multirresistentes aisladas de productos alimenticios en Rusia. Antibióticos (Basilea). (2021) 11:1. doi: 10.3390/antibióticos11010001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Carattoli, A. Importancia de los integrones en la difusión de la resistencia. Res. Veterinaria. (2001) 32:243–59. doi: 10.1051/vetres:2001122
18. Carattoli, A, Villa, L, Pezzella, C, Bordi, E y Visca, P. Expansión de la resistencia a los medicamentos a través de la adquisición de integrones por plásmidos IncFI de Salmonella enterica typhimurium. Emerg Infect Dis. (2001) 7:444–7. doi: 10.3201/eid0703.010314
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Liu, CC y Hsiao, W. Genómica comparativa a gran escala para refinar la organización de la estructura de la población global de Salmonella enterica. Genoma Microb. (2022) 8:8. doi: 10.1099/mgen.0.000906
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Elbediwi, M, Pan, H, Zhou, X, Rankin, SC, Schifferli, DM y Yue, M. Detección de Salmonella Newport productora de BLEE que alberga mcr-9 aislada de un brote en un hospital universitario de animales grandes en los EE. UU. J Quimiotera antimicrobiana. (2021) 76:1107–9. doi: 10.1093/jac/dkaa544
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Li, Y, Kang, X, Ed-Dra, A, Zhou, X, Jia, C, Muller, A, et al. Evaluación basada en el genoma de la resistencia a los antimicrobianos y el potencial de virulencia de aislados de serovares de Salmonella no Pullorum/Gallinarum recuperados de aves de corral muertas en China. Microbiol Spectr. (2022) 10:e96522:e0096522. doi: 10.1128/spectrum.00965-22
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Yang, J, Gao, S, Chang, Y, Su, M, Xie, Y y Sun, S. Ocurrencia y caracterización de Salmonella aislada de granjas reproductoras a gran escala en la provincia de Shandong. China Biomed Res Int. (2019) 2019:1–08. doi: 10.1155/2019/8159567
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Zhao, X, Gao, Y, Ye, C, Yang, L, Wang, T y Chang, W. Prevalencia y características de Salmonella aislada de pollos de corral en la provincia de Shandong, China. Biomed Res Int. (2016) 2016:1–06. doi: 10.1155/2016/8183931
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Zhu, Y, Lai, H, Zou, L, Yin, S, Wang, C, Han, X, et al. Resistencia a los antimicrobianos y genes de resistencia en cepas de Salmonella aisladas de pollos de engorde a lo largo del proceso de sacrificio en China. Int J Microbiol Alimentario. (2017) 259:43–51. doi: 10.1016/j.ijfoodmicro.2017.07.023
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Yu, X, Zhu, H, Bo, Y, Li, Y, Zhang, Y, Liu, Y, et al. Prevalencia y resistencia a los antimicrobianos de la subespecie enterica de Salmonella enterica serovar Enteritidis aislada de pollos de engorde en la provincia de Shandong, China, 2013-2018. Poult Sci. (2021) 100:1016–23. doi: 10.1016/j.psj.2020.09.079
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Instituto de Estándares Clínicos y de Laboratorio. Normas de rendimiento para las pruebas de susceptibilidad a los antimicrobianos. 23º suplemento informativo, M100-S31. Pensilvania: CLSI (2022).
27. Foley, SL, Lynne, AM y Nayak, R. Desafíos de la salmonela: prevalencia en cerdos y aves de corral y patogenicidad potencial de dichos aislados. J Anim Sci. (2008) 86:E149-62. doi: 10.2527/jas.2007-0464
28. Tang, B, Elbediwi, M, Nambiar, RB, Yang, H, Lin, J y Yue, M. Caracterización genómica de Salmonella enterica resistente a los antimicrobianos en granjas de patos, pollos y cerdos y mercados minoristas en el este de China. Microbiol Spectr. (2022) 10:e125722:e0125722. doi: 10.1128/spectrum.01257-22
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Weng, R, Gu, Y, Zhang, W, Hou, X, Wang, H, Tao, J, et al. La secuenciación del genoma completo proporciona información sobre la resistencia a los antimicrobianos y las características moleculares de la Salmonella de la carne de ganado y de pacientes con diarrea en Hanzhong. China Front Microbiol. (2022) 13:899024. doi: 10.3389/fmicb.2022.899024
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Yang, X, Huang, J, Zhang, Y, Liu, S, Chen, L, Xiao, C, et al. Prevalencia, abundancia, serovares y resistencia antimicrobiana de Salmonella aislada de carne cruda de aves de corral al por menor en China. Sci Total Environ. (2020) 713:136385. doi: 10.1016/j.scitotenv.2019.136385
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Zhao, X, Yang, J, Zhang, B, Sun, S y Chang, W. Caracterización de integrones y genes de resistencia en aislados de Salmonella de animales de granja en la provincia de Shandong, China. Microbiol frontal. (2017) 8:1300. doi: 10.3389/fmicb.2017.01300
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Chen, H, Song, J, Chen, D, Zeng, X, Qiu, C y Zhou, K. Prevalencia nacional del serotipo Kentucky ST198 de Salmonella enterica con alto nivel de resistencia a la ciprofloxacina y las cefalosporinas de espectro extendido en China, 2013 a 2017. Msistemas. (2020) 6:95. doi: 10.1128/mSystems.00935-20
33. Wang, J, Li, J, Liu, F, Cheng, Y y Su, J. Caracterización de aislados de Salmonella enterica de aves de corral enfermas en el norte de China entre 2014 y 2018. Patógenos. (2020) 9:9. doi: 10.3390/pathogens9020095
34. Walker, GK, Suyemoto, MM, Hull, DM, Gall, S, Jiménez, F, Chen, LR, et al. Caracterización genómica de una cepa de Salmonella Enteritidis resistente al ácido nalidíxico causante de infecciones persistentes en pollos de engorde. Front Vet Sci. (2021) 8:725737. doi: 10.3389/fvets.2021.725737
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Piddock, LJ. Evalúe los fenotipos de resistencia a los medicamentos, no solo los genotipos. Nat Microbiol. (2016) 1:16120. doi: 10.1038/nmicrobiol.2016.120
36. Pribul, BR, Festivo, ML, de Souza, MM, y Rodrigues, DP. Caracterización de la resistencia a quinolonas en aislados de Salmonella spp. de productos alimenticios y muestras humanas en Brasil. Braz J Microbiol. (2016) 47:196–201. doi: 10.1016/j.bjm.2015.04.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Wang, Y, Yang, B, Wu, Y, Zhang, Z, Meng, X, Xi, M, et al. Caracterización molecular de Salmonella enterica serovar Enteritidis en aves de corral crudas al por menor en seis provincias y dos ciudades nacionales de China. Microbiol alimentario. (2015) 46:74–80. doi: 10.1016/j.fm.2014.07.012
38. Wang, Y, Zhang, A, Yang, Y, Lei, C, Jiang, W, Liu, B, et al. Aparición de aislados de Salmonella enterica serovar Indiana y California con resistencia concurrente a cefotaxima, amikacina y ciprofloxacina de pollos en China. Int J Microbiol Alimentario. (2017) 262:23–30. doi: 10.1016/j.ijfoodmicro.2017.09.012
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Fang, L, Lin, G, Li, Y, Lin, Q, Lou, H, Lin, M, et al. Caracterización genómica de Salmonella enterica serovar Kentucky y Londres recuperada de salmonelosis alimentaria y humana en la provincia de Zhejiang, China (2016-2021). Microbiol frontal. (2022) 13:961739. doi: 10.3389/fmicb.2022.961739
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Le Hello, S, Harrois, D, Bouchrif, B, Sontag, L, Elhani, D, Guibert, V, et al. Serotipo Kentucky ST198-X1 de Salmonella enterica altamente resistente a los medicamentos: un estudio microbiológico. Lancet infecta Dis. (2013) 13:672–9. doi: 10.1016/S1473-3099(13)70124-5
Resumen de PubMed | Texto completo de CrossRef | Google Académico
41. Medalla, F, Gu, W, Friedman, CR, Judd, M, Folster, J, Griffin, PM, et al. Aumento de la incidencia de infecciones por Salmonella no tifoidea resistente a los antimicrobianos, Estados Unidos, 2004-2016. Emerg Infect Dis. (2021) 27:1662–72. doi: 10.3201/eid2706.204486
Resumen de PubMed | Texto completo de CrossRef | Google Académico
42. Penha, FR, Ferreira, JC, Kanashiro, A, Berchieri, JA, y Darini, A. Serovares emergentes de Salmonella no tifoidea multirresistentes aislados de aves de corral en Brasil que albergan Bla (CTX-M-2) y qnrB o Bla (CMY-2) en plásmidos grandes. Diagn Microbiol Infect Dis. (2019) 95:93–8. doi: 10.1016/j.diagmicrobio.2019.04.003
43. Liu, YY, Wang, Y, Walsh, TR, Yi, LX, Zhang, R, Spencer, J, et al. Aparición del mecanismo de resistencia a la colistina mediado por plásmidos MCR-1 en animales y seres humanos en China: un estudio microbiológico y biológico molecular. Lancet infecta Dis. (2016) 16:161-68. doi: 10.1016/S1473-3099(15)00424-7
44. Ma, S, Lei, C, Kong, L, Jiang, W, Liu, B, Men, S, et al. Prevalencia, resistencia a los antimicrobianos y relación de Salmonella aislada de pollos y cerdos en granjas, mataderos y mercados en la provincia de Sichuan, China. Pathog Dis. (2017) 14:667–77. doi: 10.1089/fpd.2016.2264
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Stefaniuk, EM, y Tyski, S. Resistencia a la colistina en cepas de Enterobacterales: una visión actual. Pol J Microbiol. (2019) 68:417–27. doi: 10.33073/pjm-2019-055
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Viñas, J, Cusco, A, Napp, S, Álvarez, J, Sáez-Llorente, J.L., Rosas-Rodoreda, M, et al. Transmisión de plásmidos portadores de Mcr-1 similares entre diferentes linajes de Escherichia coli aislados del ganado y del agricultor. Antibióticos (Basilea). (2021) 10:313. doi: 10.3390/antibióticos10030313
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. Perdiz, SR, Kwong, SM, Firth, N, y Jensen, SO. 42-Elementos genéticos móviles asociados a la resistencia a los antimicrobianos. Clin Microbiol Rev. (2018) 31:31. doi: 10.1128/CMR.00088-17
Resumen de PubMed | Texto completo de CrossRef | Google Académico
48. Fei, X, Yin, K, Yin, C, Hu, Y, Li, J, Zhou, Z, et al. 43: los análisis de prevalencia y tipificación molecular revelan la propagación de la infección por Salmonella resistente a los antimicrobianos en dos granjas de pollos reproductores. Poult Sci. (2018) 97:4374–83. doi: 10.3382/ps/pey305
49. Davies, R, y Wales, A. Resistencia a los antimicrobianos en las granjas: una revisión que incluye la bioseguridad y el papel potencial de los desinfectantes en la selección de resistencias. Compr Rev Food Sci Food Saf. (2019) 18:753–74. doi: 10.1111/1541-4337.12438
Resumen de PubMed | Texto completo de CrossRef | Google Académico
50. Chousalkar, K, y Gole, VC. Salmonelosis adquirida de aves de corral. Curr Opin Infect Dis. (2016) 29:514–9. doi: 10.1097/QCO.0000000000000296
51. Galanis, E, Lo, FWD, Patrick, ME, Binsztein, N, Cieslik, A, Chalermchikit, T, et al. Vigilancia basada en la web y distribución mundial de Salmonella, 2000-2002. Emerg Infect Dis. (2006) 12:381–8. doi: 10.3201/eid1205.050854
Resumen de PubMed | Texto completo de CrossRef | Google Académico
52. Foley, SL, Nayak, R, Hanning, IB, Johnson, TJ, Han, J y Ricke, SC. Dinámica poblacional de los serotipos de Salmonella enterica en la producción comercial de huevos y aves de corral. Appl Environ Microbiol. (2011) 77:4273–9. doi: 10.1128/AEM.00598-11
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: Salmonella, WGS, resistencia a antimicrobianos, pollos de engorde, genes de resistencia
Cita: Zhao L, Liu G, Tang W, Song X, Zhao X, Wang C, Li Y y Zou M (2023) Resistencia a los antimicrobianos y características genómicas de la salmonela de pollos de engorde en la provincia de Shandong. Frente. Vet. Sci. 10:1292401. doi: 10.3389/fvets.2023.1292401
Recibido: 11 de septiembre de 2023; Aceptado: 06 de noviembre de 2023;
Publicado: 23 noviembre 2023.
Editado por:
Nora Mestorino, Universidad Nacional de La Plata, Argentina
Revisado por:
Fabiana Alicia Moredo, Universidad Nacional de La Plata, Argentina Maria Laura Marchetti, Universidad Nacional de La Plata, Argentina
Derechos de autor © 2023 Zhao, Liu, Tang, Song, Zhao, Wang, Li y Zou. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY). S
*Correspondencia: Youzhi Li, liyouzhi2009@126.com; Ming Zou, zoudnet@163.com
†Estos autores han contribuido igualmente a este trabajo y comparten la primera autoría
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía